1. INTRODUCIÓN
Las vacunas se consideran el método más eficaz para controlar la pandemia de COVID-19 y han contribuido a la recuperación de la economía global. Sin embargo, la eficacia de la respuesta inmune protectora inducida por estas vacunas frente a las variantes de preocupación (VOC, por sus siglas en inglés) presenta limitaciones. Las nuevas VOC muestran una elevada tasa de mutación en el dominio de unión al receptor (RBD) y en las secuencias del dominio N-terminal de la glicoproteína espicular (S) del SARS-CoV-2. La inmunidad basada en anticuerpos frente a estas VOC es menos eficaz en comparación con la cepa original Wuhan-Hu-1. No obstante, la respuesta de los linfocitos T frente a estas variantes permanece robusta.
Los linfocitos T reconocen más epítopos de la proteína S que los anticuerpos. Además, las respuestas de células T no decrecen tan rápidamente como las de los anticuerpos, por lo que proporcionan una protección más eficaz frente a variantes emergentes. Las vacunas que expresan la proteína S derivada de variantes como Ómicron no mejoran necesariamente la eficacia en comparación con la proteína S original. Esto sugiere que adaptar el antígeno vacunal a la forma más reciente de S no garantiza una mejora en la eficacia. El desarrollo de vacunas altamente eficaces para prevenir la COVID-19 sigue siendo una necesidad a nivel global. En el futuro, serán necesarias vacunas capaces de inducir una respuesta celular adecuada para minimizar el impacto de nuevas variantes sobre los sistemas sanitarios.
Las vacunas de ADN, utilizadas en medicina veterinaria, inducen respuestas inmunes protectoras específicas frente a patógenos. Estas vacunas son fácilmente modificables, lo que permite ensayar rápidamente múltiples candidatos vacunales frente a nuevas cepas víricas. Una de las ventajas de las vacunas de ADN es que su producción puede escalarse fácilmente. Además, estas vacunas son termoestables, lo cual elimina la necesidad de cadena de frío para su almacenamiento a largo plazo y distribución global. Esta característica resulta particularmente ventajosa para países en desarrollo. Actualmente se están ensayando varios candidatos de vacunas de ADN frente a la COVID-19 en ensayos clínicos, aunque la mayoría incluye genes de resistencia a antibióticos como marcador de selección en su proceso de fabricación.
Hemos desarrollado un nuevo candidato vacunal de ADN para prevenir la infección por SARS-CoV-2, que incluye las secuencias completas de los genes de la proteína S y de la nucleocápside (N), clonadas en el plásmido pPAL, un vector de expresión en mamíferos no replicativo y libre de genes de resistencia a antibióticos. La secuencia del gen S ha sido modificada para estabilizar la proteína resultante frente al corte por furina (Sfs) e incluye el dominio RBD, responsable de la entrada del virus en la célula hospedadora. El candidato vacunal pPAL-Sfs + pPAL-N se compone de una mezcla en proporción 1:1 en masa de ambos plásmidos disueltos en agua estéril. Se optó por mantener los genes en plásmidos separados para evitar un plásmido de mayor tamaño, lo que podría reducir el rendimiento en la producción.
El plásmido pPAL contiene una isla CpG larga y no requiere adyuvantes adicionales. La proteína N fue seleccionada debido a su alto grado de conservación entre los betacoronavirus. Recientemente se ha propuesto un papel protector para la proteína N frente a la infección por SARS-CoV-2. Además, esta proteína es la más abundante del virus, altamente inmunogénica en infecciones por coronavirus, y puede contribuir a ampliar la respuesta de células T y mejorar la reactividad cruzada.
Por tanto, evaluamos la eficacia del candidato vacunal pPAL-Sfs + pPAL-N frente a SARS-CoV-2 mediante un régimen de inmunización de primovacunación-refuerzo por vía intramuscular seguido de electroporación in vivo. Se evaluaron las respuestas inmunes humoral y celular en ratones C57BL/6J de tipo salvaje, y el nivel de protección frente a un desafío letal con SARS-CoV-2 se valoró en ratones transgénicos B6Cg-Tg(K18-hACE2)2Prlmn/J. Este estudio demuestra que el candidato vacunal pPAL-Sfs + pPAL-N es una propuesta prometedora frente a la infección por SARS-CoV-2.
2. MATERIALES Y MÉTODOS
2.1 Construccion pPAL
Las vacunas de ADN requieren un vector que permita la replicación de los genes y la expresión de los antígenos codificados. Habitualmente, se emplean genes de resistencia a antibióticos como marcadores de selección, los cuales no deben ser liberados al medio ambiente tras la comercialización del producto final. Por tanto, el uso de vectores libres de genes de resistencia a antibióticos es imprescindible.
El plásmido de expresión en mamíferos pPAL está basado en las secuencias potenciadora y promotora del citomegalovirus (CMV). Este plásmido no se replica en células de mamífero y no contiene marcadores de selección basados en resistencia a antibióticos. En su lugar, utiliza como marcador de selección el gen fabI de E. coli, que codifica para la enzima enoil-ACP reductasa. Esta enzima es inhibida por el compuesto bacteriostático triclosán, empleado como agente selectivo a una concentración óptima de 3 μM. El constructo pPAL-Sfs (Figura 1A) contiene una versión modificada del gen que codifica la proteína S del SARS-CoV-2, correspondiente al número de acceso NCBI NC045512, ID de gen 43740568. En primer lugar, la secuencia fue optimizada mediante un enfoque Monte Carlo basado en las frecuencias relativas de uso de codones. En segundo lugar, se modificó el sitio de corte por furina (PRRA → PGGS; residuos 681-684). Para ello, la secuencia nucleotídica CCTCGGCGGGCA fue sustituida por CCAGGCGGCAGC (posiciones 2041–2052).
El constructo pPAL-N contiene el gen de la proteína N del SARS-CoV-2 (también con número de acceso NC045512, ID de gen 43740575). Los constructos pPAL-Sfs y pPAL-N, flanqueados por sitios de restricción KpnI, fueron obtenidos por síntesis génica en el vector pGH (ATG Biosynthetics, Merzhausen, Alemania) y transferidos a la cepa E. coli SURE2 (Agilent, Santa Clara, CA) mediante electroporación (1.800 V, 200 W, 25 μF) (FIG 1B).
La selección se realizó en medio LB-agar suplementado con triclosán a 3 μM (Sigma-Aldrich, St. Louis, MO, EE. UU.). El plásmido pGH fue eliminado mediante digestión con la enzima KpnI (NEB, Ipswich, MA), y los constructos vacunales se circularizaron utilizando ligasa T4 (NEB). Las preparaciones libres de endotoxinas de los plásmidos pPAL, pPAL-Sfs y pPAL-N se obtuvieron con el kit PureLink™ Expi Endotoxin-Free Giga Plasmid Purification Kit (Invitrogen, Waltham, MA), siguiendo las instrucciones del fabricante.
2.2 Expresión génica de antígenos en células HEK293 transfectadas
Las células HEK293 (CRL-1573™, ATCC®, Manassas, VA) se cultivaron a 37 °C en una atmósfera con 5% de CO₂ en medio completo (CM), compuesto por DMEM suplementado con un 10% de suero fetal bovino inactivado por calor (HIFBS, Sigma-Aldrich, Burlington, MA), 100 UI de penicilina y 100 µg/mL de estreptomicina. Las células en estado de semiconfluencia fueron desprendidas por pipeteo suave, lavadas una vez con 1 mL de PBS estéril y una vez con 0,3 mL de GTporator®-M (Protean, República Checa) por cada 3 × 10⁶ células. Posteriormente, se transfectaron por electroporación con los plásmidos pPAL, pPAL-S, pPAL-Sfs o pPAL-N. Se incluyó un control de transfección sin ADN. Se transfectaron 1,2 × 10⁶ células con 5 µg de ADN en 80 µL de solución GTporator®-M, utilizando una cubeta estéril con un espacio de 2 mm (BTX, Cambridge, Reino Unido) y un equipo ECM 630 Electro Cell Manipulator Precision Plus® (BTX). Inmediatamente se añadieron 1,2 mL de medio precalentado. Se sembraron 0,2 mL de la suspensión celular en una porta de 8 pocillos (Nunc® Lab-Tek®, Sigma-Aldrich) y 1 mL en una placa de 24 pocillos. Las células se incubaron 24 h a 37 °C y 5% CO₂.
Las células transfectadas en placas de 24 pocillos se lavaron dos veces con 1 mL de CM y se lisaron con 50 μL de tampón (25 mM Tris-HCl pH 7,8, 2 mM EDTA, 2 mM DTT, 1% glicerol, 1% Triton-X100). Las proteínas se cuantificaron por el método de Bradford. Cada extracto (20 μg) fue tratado con 8,3 U/μL de TurboNuclease (Serratia marcescens, Sigma-Aldrich) durante 10 min a temperatura ambiente. Luego se prepararon para electroforesis SDS-PAGE en tampón Laemmli, calentados a 95 °C durante 5 min, y corridos a 30 mA durante 90 min en geles TGX preensamblados al 8-20% (BioRad). Las proteínas fueron transferidas por método semiseco a membranas de nitrocelulosa de 0,45 μm (BioRad) a 1,3 A y 25 V durante 10 min, utilizando el sistema TransBlot Turbo. Las membranas se bloquearon con leche desnatada al 5% en PBS-Tween 0,1% a temperatura ambiente durante 1 h, bajo agitación suave, y se lavaron tres veces. Luego, se incubaron 90 min con anticuerpos primarios en tampón de bloqueo: anti-SARS-CoV-2 S (subunidad S2, Abcam ab272504) a 1:750 y anti-N (cortesía de Mercedes Domínguez e Inmaculada Moreno, CNM-ISCIII) a 1:500. Tras los lavados, se incubó 1 h con anticuerpo secundario HRP-conjugado con anti-IgG de cabra anti-ratón (DAKO Agilent) a 1:2.000. La detección por quimioluminiscencia se realizó con ECL Western Blotting Reagent (Thermo Fisher) durante 1 min. Las imágenes se adquirieron con el sistema ChemiDoc MP (BioRad) y se analizaron en ImageLab 6.1 (BioRad).
Las células en portas de 8 pocillos se lavaron con 200 μL de solución hipotónica (agua:DMEM, 11:9) y dos veces con acetona:metanol (1:1). Se fijaron y permeabilizaron con acetona:metanol (1:1) a –20 °C durante 10 min, se secaron al aire y se retiraron los pocillos. Se realizaron tres lavados de 5 min con PBS filtrado (0,22 μm), se secaron al aire y se bloquearon con 20 μL de leche desnatada al 5% en PBS-Tween, durante 1 h a 37 °C en cámara húmeda. Después del bloqueo, se añadieron 20 μL de los anticuerpos anti-S2 y anti-N diluidos 1:50, y se incubaron 1 h. Tras un lavado de 5 min, se incubaron con 20 μL de anticuerpo secundario Alexa Fluor® 488 anti-IgG de cabra (1:200) durante 1 h en oscuridad. A los 50 min de esta incubación, se añadieron 20 μL de DAPI (10 μg/mL). Finalmente, se realizaron cuatro lavados de 5 min con PBS filtrado, se montaron con 50 μL de Mowiol 4-88 y se dejaron secar a 4 °C durante 16 h en oscuridad. Las imágenes de fluorescencia se adquirieron con un microscopio confocal SP8 STED 3X (Leica, Alemania).
2.3 Inmunización con pPAL-Sfs + pPAL-N
Se utilizaron 87 ratones hembra C57BL/6J de tipo salvaje, de ocho semanas de edad (Charles River, Wilmington, MA), para el análisis de la respuesta inmune, y 140 ratones hembra transgénicos B6Cg-Tg(K18-hACE2)2Prlmn/J (Jackson ImmunoResearch, West Grove, PA) para los experimentos de protección.
Los animales fueron alojados, por norma general, en grupos de cinco, excepto en el caso de cuatro grupos de ocho ratones C57BL/6J en el experimento de dosis-respuesta. En todo momento se cumplieron los requisitos de espacio establecidos por la legislación vigente (Directiva 2010/63/UE y el Real Decreto 53/2013, modificado por el RD 1386/2018). Los permisos necesarios fueron aprobados por el Comité de Ética del Consejo Superior de Investigaciones Científicas (CSIC) y la Comunidad Autónoma de Madrid.
Los experimentos con ratones infectados se realizaron en laboratorios de bioseguridad nivel 3+ (BSL3+, CISA-INIA-CSIC). Todos los animales recibieron alimentación y agua ad libitum. Se aplicaron medidas de bienestar animal, siguiendo los principios de reemplazo, reducción y refinamiento. También se implementaron medidas de enriquecimiento ambiental.
Durante la administración de la vacuna y la electroporación in vivo, se aplicó anestesia con isofluorano (3% para inducción, 2,5% para mantenimiento). Además, se añadió ibuprofeno al agua de bebida a una dosis de 0,2 mg/kg tras el procedimiento. Para la eutanasia, también se utilizó isofluorano seguido de punción intracardíaca. Se aplicaron todas las medidas adicionales exigidas por la normativa. En los ratones K18-hACE2 sometidos al desafío viral, se aplicó eutanasia inmediata si se detectaba una pérdida de peso igual o superior al 20%, o cualquier signo incipiente de sufrimiento. Los procedimientos empleados en los experimentos de desafío fueron posteriormente evaluados de forma retrospectiva.
Los ratones C57BL/6J y K18-hACE2 fueron inmunizados por vía intramuscular (IM) con la vacuna pPAL-Sfs + pPAL-N, compuesta por ADN libre de endotoxinas (20 μg de pPAL-Sfs y 20 μg de pPAL-N) disuelto en agua estéril CHROMASOLV™ (Honeywell Riedel-de Haën, Charlotte, NC). Los ratones control recibieron 40 μg de pPAL vacío.
La electroporación se realizó utilizando dos electrodos tipo aguja de 30G (0,3 × 13 mm), conectados a un sistema de electroporación ECM 830 Square Wave (BTX, Harvard Bioscience Inc.). Las agujas se colocaron equidistantes al punto de inyección, siguiendo la dirección de las fibras musculares, con una separación aproximada de 5 mm. El polo negativo se situó en posición posterior. Se aplicaron seis pulsos de 100 V, de 50 ms cada uno, a intervalos de 1 segundo. Los animales fueron inmovilizados sobre una superficie eléctricamente aislada y se mantuvieron bajo anestesia con isofluorano (3% inducción, 2,5% mantenimiento) durante todo el procedimiento. Posteriormente, se administró ibuprofeno en el agua a 0,2 mg/kg. Los ratones C57BL/6J fueron sacrificados una semana después de la dosis de refuerzo para evaluar la respuesta inmune celular en el bazo. Los ratones K18-hACE2 fueron desafiados con el virus dos semanas después de la dosis de refuerzo, tras evaluar la respuesta inmune humoral.
2.4 ELISA
Se recogieron 100 μL de sangre periférica a los ratones una semana antes de la vacunación y 15 días después de la misma. La sangre se dejó coagular a 4 °C durante 5 horas. El suero se obtuvo por centrifugación a 8.000 ×g durante 10 minutos y se almacenó a –20 °C hasta su utilización. Se prepararon diluciones seriadas 1:3 de los sueros en PBS con 0,05% de Tween 20 y 1% de albúmina sérica bovina (BSA). Se recubrieron placas de microtitulación de 96 pocillos de fondo plano con 50 μL de proteína recombinante S1+S2 ECD-His (Sino Biological, Beijing, China) y RBD (RayBiotech Life, Peachtree Corners, GA), a una concentración de 50 μg/mL en tampón carbonato-bicarbonato (3,36 mM/10 mM), incubándose a 4 °C durante 16 horas. Tras tres lavados con PBS-Tween 0,05% + 1% BSA, se bloqueó la placa con PBS-Tween 0,05% conteniendo 3% de BSA durante 1 hora a temperatura ambiente. Luego, se repitieron los lavados y se añadieron 100 μL de las muestras de suero diluidas, incubando durante 1 hora.
Después de los lavados, se incubó con uno de los siguientes conjugados con HRP durante 1 hora:
- Proteína A conjugada a HRP diluida 1:8.000 (Invitrogen, Waltham, MA)
- Anticuerpo IgG1 de cabra anti-ratón diluido 1:20.000
- Anticuerpo IgG2c de cabra anti-ratón diluido 1:20.000 (Bethyl Laboratories, Montgomery, TX)
Posteriormente se realizaron tres lavados finales. El desarrollo de color se llevó a cabo utilizando el kit TMB Substrate (Thermo Scientific, Waltham, MA), incubando 100 μL durante 10 minutos. La reacción se detuvo añadiendo 100 μL de H₂SO₄ 2 N. La absorbancia a 450 nm (A450) se midió con un lector Microplate Reader 680 (BioRad, Hercules, CA) y el software Microplate Manager 5.2.1 (BioRad).
2.4.1 Ensayos de neutralización de SARS-CoV-2
La cepa viral MAD6 de SARS-CoV-2 y la variante B.1.617.2 (Delta) fueron gentilmente proporcionadas por el Dr. Luis Enjuanes y el Dr. Juan Francisco García-Arriaza (CNB-CSIC, Madrid, España), respectivamente. La secuencia genómica de la cepa MAD6 es idéntica a la del aislado original Wuhan-Hu-1 (GenBank MN908947). Se prepararon diluciones seriadas de sueros de ratón previamente inactivados por calor (dilución inicial 1:10) y se incubaron con 100 unidades formadoras de placas (PFU) del virus SARS-CoV-2 MAD6 o de la variante Delta (B.1.617.2) durante 1 hora a 37 °C, en placas de 96 pocillos de fondo plano. Posteriormente, se sembraron 2 × 10⁴ células Vero E6 por pocillo sobre la mezcla de suero/virus, y se incubaron durante 3 días a 37 °C con 5% de CO₂. Tras la incubación, se retiró el medio de cultivo y las células se fijaron con paraformaldehído al 2%. A continuación, se tiñeron con cristal violeta al 2%. Los títulos de anticuerpos neutralizantes (NAb) se calcularon como la dilución del suero en la que se observó menos del 50% de efecto citopático en los pocillos replicados.
2.4.2 Ensayos ELISpot y tinción intracelular de citoquinas
Se obtuvieron suspensiones de esplenocitos en 15 mL de PBS conteniendo 0,3 mM de EDTA y 2% de suero fetal bovino inactivado por calor (HIFBS), utilizando filtros celulares de 0,45 μm (BD Falcon Cell Strainers, BD Biosciences, San José, CA). Las células se recolectaron por centrifugación a 500 ×g durante 7 minutos, y los eritrocitos se lisaron a temperatura ambiente durante 2 minutos con 5 mL de tampón de lisis de eritrocitos (1X RBC Lysis Buffer, pluriSelect Life Science, Leipzig, Alemania). A continuación, se añadieron inmediatamente 25 mL de solución de lavado y se centrifugó nuevamente a 500 ×g durante 7 minutos. Tras un lavado adicional, las células se resuspendieron en 5 mL de medio de proliferación (RPMI suplementado con 10% HIFBS, 100 UI de penicilina – 100 μg/mL de estreptomicina y 4,5 μM de β-mercaptoetanol). Las suspensiones se filtraron nuevamente mediante filtros de 0,45 μm, y se diluyó una alícuota 1:1 con azul tripano al 0,4% (Sigma-Aldrich) para el recuento de células viables, usando portaobjetos TC10 y un contador automático TC10 (BioRad).
2.4.3 ELISpot
Los ensayos ELISpot se realizaron utilizando el kit Murine IFN-γ ELISpot (Abcam, Reino Unido), siguiendo las instrucciones del fabricante. En la fase de estimulación, se sembraron 2 × 10⁵ esplenocitos en 200 μL de medio de proliferación con el estímulo correspondiente. La estimulación se realizó por triplicado, incubando 20 horas a 37 °C y 5% de CO₂. Los estímulos empleados fueron:
- Concanavalina A (1 μg/mL) como control positivo (Sigma-Aldrich),
- Una mezcla equimolar de péptidos representando la secuencia completa de la proteína S (25 μg/mL), preparada a partir de PepTivator® SARS-CoV-2 Prot_S1 y Prot_S (Miltenyi Biotec, Alemania),
- Una mezcla equimolar de péptidos representando la proteína N completa (25 μg/mL), a partir de PepTivator® SARS-CoV-2 Prot_N (Miltenyi Biotec).
2.4.4 Tinción intracelular de citoquinas (citometría de flujo)
Los esplenocitos se sembraron en placas de cultivo de 96 pocillos (1 × 10⁶ células/pocillo) y se estimularon con el pool de péptidos de la proteína S (25 μg/mL, Miltenyi Biotec), o con PMA (50 ng/mL) e ionomicina (1 μg/mL) como control positivo (Sigma-Aldrich). El grupo negativo no recibió estímulo.
Durante la estimulación se añadieron:
- Brefeldina A (5 μg/mL),
- Monensina (2 mM),
- Anticuerpo anti-CD107a (1 μg/mL; PE anti-mouse CD107a, clon 1D4B, Biolegend).
Las células se incubaron durante 4 horas adicionales a 37 °C, en atmósfera humidificada con 5% CO₂. Posteriormente, se lavaron con PBS y se tiñeron con el marcador de viabilidad LIVE/DEAD™ Fixable Near-IR (Thermo Fisher) durante 20 minutos sobre hielo. Luego se lavaron con tampón de tinción (PBS + 2% HIFBS + 0,02% azida sódica) y se tiñeron los marcadores de superficie con los siguientes anticuerpos anti ratón, conjugados con fluoróforos:
- CD8a (Alexa Fluor® 488, clon 53-6.7),
- CD4 (Brilliant Violet 510™, clon GK1.5),
- CD3 (PerCP/Cy5.5, clon 17A2), todos de Biolegend.
Tras dos lavados adicionales, las células se fijaron y permeabilizaron usando el kit Cytofix/Cytoperm (BD Biosciences), según las instrucciones del fabricante. Luego se tiñeron durante 30 minutos sobre hielo para las citoquinas intracelulares:
- TNF-α (Brilliant Violet 421™, clon MP6-XT22),
- IFN-γ (Alexa Fluor® 647, clon XMG1.2),
- IL-2 (Brilliant Violet 785™, Biolegend).
Finalmente, las células se lavaron según el protocolo del kit, se resuspendieron en tampón de tinción y se adquirieron en un citómetro FACSCelestaSORP (BD Biosciences). El análisis de datos se realizó con el software FlowJo (BD Biosciences).
2.4.5 Mantenimiento del SARS-CoV-2 y desafío infeccioso
La línea celular Calu-3 se cultivó en medio DMEM suplementado con un 20% de suero fetal bovino inactivado por calor (HIFBS) y 10 mM de HEPES. Esta línea celular fue proporcionada amablemente por el Dr. Luis Enjuanes (CNB-CSIC, Madrid, España) y se utilizó para la propagación viral a una multiplicidad de infección (MOI) de 0,001 unidades formadoras de placa (PFU) por célula. El sobrenadante se recogió a las 72 horas postinfección (hpi), se sometió a tres ciclos de congelación/descongelación y se clarificó por centrifugación a 1.970 ×g durante 10 minutos. El virus fue titulado y almacenado a –80 °C hasta su uso. La titulación se realizó utilizando la línea celular Vero E6 (CRL-1586, ATCC®, Manassas, VA), mantenida en medio DMEM suplementado con 5% de HIFBS. La adsorción viral se permitió durante 1 hora sobre monocapas de células Vero E6 en estado de confluencia del 70–80%. Luego se añadieron 2 mL de agar semisólido al 0,5% en medio DMEM suplementado con 2% de HIFBS. El recuento de unidades formadoras de placa (PFU) se realizó tras 6 días de incubación. Los ratones fueron desafiados con 10⁵ PFU de las cepas MAD6 o Delta de SARS-CoV-2 por vía intranasal, 15 días después de la dosis de refuerzo. A partir de entonces, se monitorizó diariamente el peso corporal y el estado clínico de los animales.
El seguimiento del peso y del estado clínico se realizó en grupos de 10 ratones K18-hACE2 por condición experimental (grupo control pPAL y grupo vacunado pPAL-Sfs + pPAL-N). El índice clínico se calculó según los criterios especificados en la Tabla Suplementaria 1. Para la evaluación de la carga viral, se emplearon 20 ratones por grupo en cada experimento, distribuidos en subgrupos de cinco individuos, sacrificados en los días 2, 4, 7 y 14 tras el desafío.
2.4.6 Evaluación del estado clínico
Los ratones fueron observados y pesados diariamente tras el desafío vírico. Los signos clínicos se puntuaron según los criterios recogidos en la Tabla S1. La puntuación total (sumatoria) de los signos clínicos —que incluye peso corporal, aspecto, movilidad y respiración— se utilizó como indicador de la gravedad de la enfermedad. Se estableció un criterio de finalización humanitaria (eutanasia) cuando esta puntuación superaba el valor de 50, con el objetivo de minimizar el sufrimiento animal.
2.4.7 Evaluación de la carga viral en órganos diana
Se obtuvieron muestras de los órganos diana (pulmón, corazón y cerebro) en los días 2, 4, 7 y 14 tras el desafío. Estas muestras fueron sometidas a tres ciclos de congelación/descongelación, seguidos de 20 ciclos de sonicación por minuto a 5 W durante 2 minutos. Los lisados se centrifugaron a 220 ×g durante 5 minutos para obtener sobrenadantes virales clarificados. La extracción de ARN total se realizó utilizando el reactivo Trizol (Invitrogen, Waltham, MA), siguiendo las instrucciones del fabricante. Para la detección del ARN viral, se llevó a cabo una PCR cuantitativa en dos pasos (qRT-PCR) utilizando transcriptasa reversa SuperScript III (Invitrogen) y ADN polimerasa EHF (Roche, Basilea, Suiza), siguiendo el protocolo de Toussaint et al. (18).
Se emplearon los siguientes cebadores y sonda específicos para el gen N del SARS-CoV-2:
N1-F: GACCCCAAAATCAGCGAAAT
N1-R: TCTGGTTACTGCCAGTTGAATCTG
N1-P (sonda): FAM-ACCCCGCATTACGTTTGGTGGACC-BHQ1
Como gen de referencia (β-actina), se utilizaron:
ACT_F_1005-1029: CAGCACAATGAAGATCAAGATCATC
ACT_R_1135-1114:CGGACTCATCGTACTCCTGCTT
ACT_P_1081-1105: JOE-TCGCTGTCCACCTTCCAGCAGATGT-BHQ1
Los mismos sobrenadantes clarificados de los órganos se emplearon para determinar la replicación viral mediante ensayos de placas en células Vero, expresándose los resultados como PFU por gramo de tejido.
2.4.8 Análisis estadístico
Para los experimentos de tinción intracelular de citoquinas, se utilizó ANOVA de dos factores con prueba post-hoc de Fisher (Least Difference). En todos los demás experimentos, se aplicó la prueba t de Student, ajustando los valores de p mediante el método de Holm-Sidak.
3. RESULTADOS
3.1 Las células HEK293 transfectadas con la vacuna candidata pPAL-Sfs + pPAL-N expresan los genes Sfs y N de SARS-CoV-2
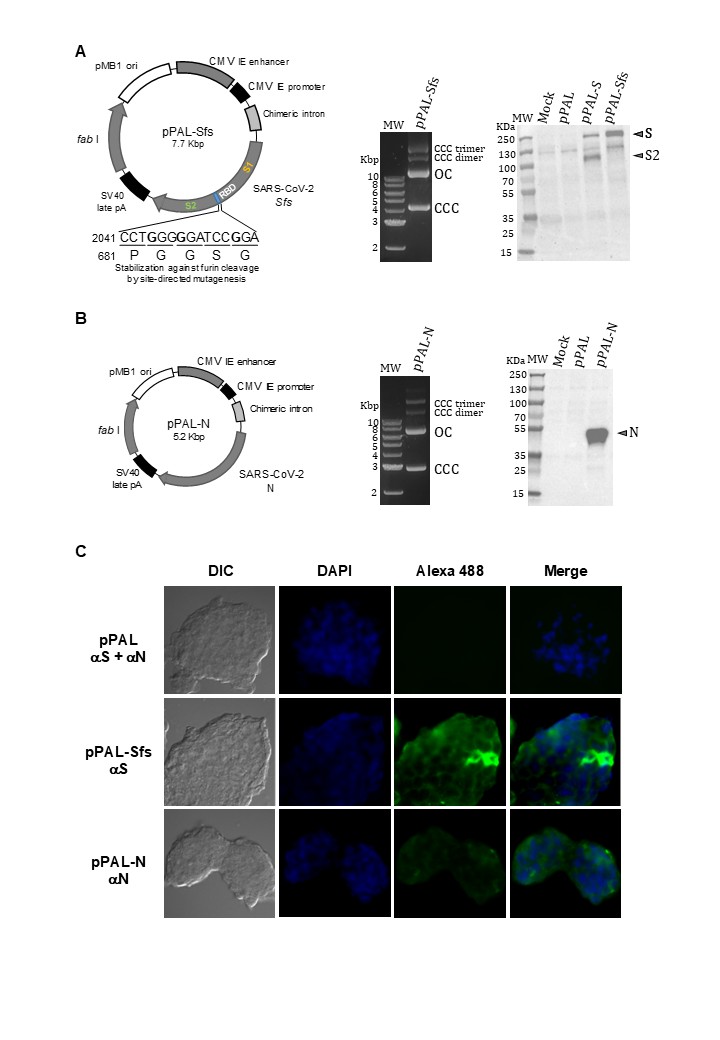
Las secuencias génicas de las proteínas S y N se obtuvieron del genoma del aislado Wuhan-1 de SARS-CoV-2 (GenBank Acc. No. MN908947). La variante Sfs es una versión modificada del gen S, diseñada para evitar el corte por furina (cambio en la secuencia: 2041 CCTCGGCGGGCA → CCAGGCGGCAGC; aminoácidos 681 PRRA → PGGS). La furina endógena de las células HEK293 cliva la glicoproteína S nativa, pero no Sfs, lo que se demuestra por la presencia del fragmento S2 solo en células transfectadas con pPAL-S (Figura 1A).
Los genes Sfs y N fueron clonados por separado en el vector de expresión en mamíferos pPAL, bajo el control del potenciador/promotor del CMV, generando la vacuna candidata pPAL-Sfs + pPAL-N (Figura 1). La transfección de células HEK293 con cada constructo produjo expresión de los antígenos correspondientes, según se confirmó mediante Western blot (Figuras 1A, B). La inmunofluorescencia indirecta utilizando anticuerpos policlonales anti-S2 y anti-N también confirmó la expresión de Sfs y N (Figuras 1C, D).
Figura 1. La vacuna candidata pPAL-Sfs + pPAL-N. (A) Mapa pPAL-Sfs. Sfs es una versión modificada optimizada para codones de la secuencia de referencia del SARS-CoV-2 de Wuhan que contiene una modificación para evitar la escisión del producto proteico por la furina. Preparación en laboratorio a gran escala del plásmido pPAL-Sfs que contiene conformadores superenrollados (CCC), formas circulares abiertas (OC), dímeros CCC y trímeros CCC. Western blot de extractos de proteínas enteras de células HEK293 transfectadas con pPAL, pPAL-S y pPAL-Sfs. El anticuerpo primario fue el anticuerpo policlonal anti-S de cabra, diluido 1:800 (Abcam #ab272504, Cambridge, Reino Unido), que solo identifica la subunidad S2. El anticuerpo secundario fue Ig policlonal anti-conejo conjugado con HRP diluido 1:2.000. La furina escinde la proteína S obtenida de pPAL-S, pero no la Sfs de pPAL-Sfs. (B) Preparación de laboratorio a gran escala del plásmido pPAL-N que contiene conformadores CCC, OC, CCC-dímero y CCC-trímero. Western blot de extractos de proteínas enteras de células HEK293 transfectadas con pPAL y pPAL-N. El anticuerpo primario fue un anticuerpo policlonal anti-N de conejo diluido 1:500. El anticuerpo secundario fue Ig policlonal anti-conejo conjugado con policlonal anti-conejo conjugado con HRP diluido 1:2.000. (C) y (D) inmunofluorescencia de células HEK293 cultivadas transfectadas con plásmidos pPAL, pPAL-Sfs y pPAL-N. Las diluciones de anticuerpos anti-S y anti-N fueron 1:50. El control de pPAL se incubó con ambos anticuerpos. El anticuerpo secundario fue Alexa Fluor® 488 IgG anti-conejo conjugado en cabra diluido a 1:200. Se observa la expresión de Sfs y N.
3.2 La vacunación con pPAL-Sfs + pPAL-N induce respuestas inmunes humorales y celulares específicas frente a SARS-CoV-2 en ratones
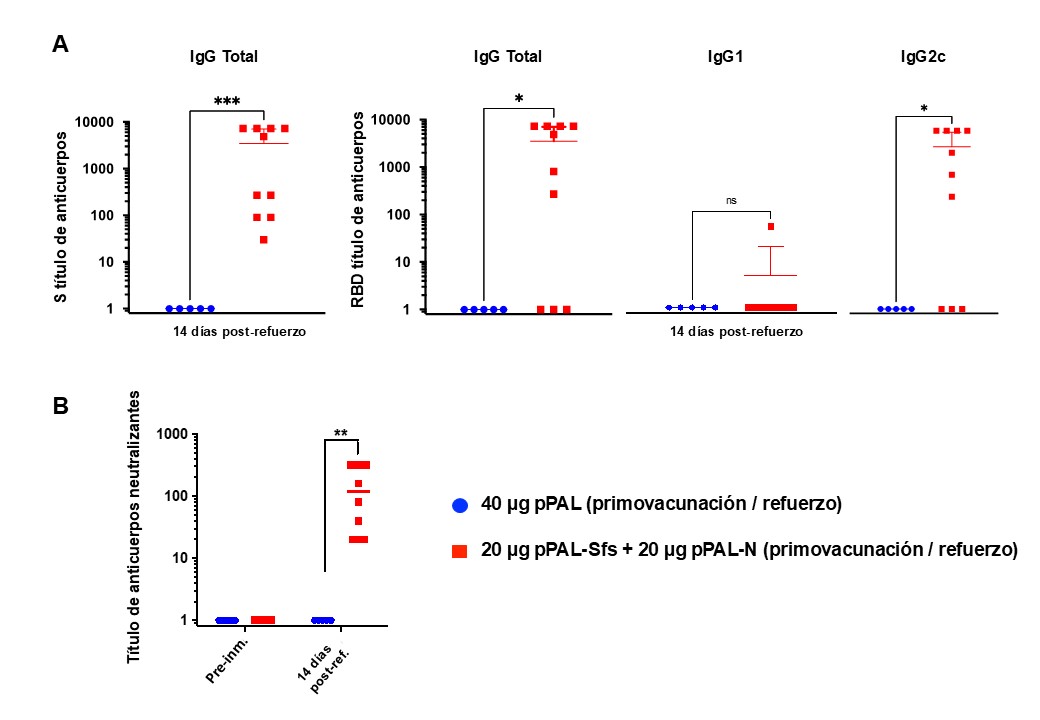
Ratones C57BL/6J (B6) fueron inmunizados por vía intramuscular con 20 μg de pPAL-Sfs y 20 μg de pPAL-N, siguiendo un esquema homólogo primovacunación/refuerzo con un intervalo de 15 días e incluyendo electroporación in vivo. Los títulos promedio de IgG anti-S, IgG anti-RBD e IgG2c anti-RBD superaron los 2.000. La razón IgG2c/IgG1 fue aproximadamente 750 en los ratones vacunados (Figura 2A). Todos los animales vacunados presentaron anticuerpos neutralizantes (NAb), con títulos promedio en torno a 100 tras la dosis de refuerzo (Figura 2B).
Figura 2. Las vacunas candidatas pPAL-Sfs + pPAL-N activan la inmunidad humoral específica del SARS-CoV-2 S y RBD en ratones C57BL/6J. Las muestras de suero se recogieron 15 días después de la dosis de refuerzo. (A) Determinaciones de ELISA de títulos individuales y promedio de IgG anti-S y anti-RBD circulante total determinados por ELISA utilizando proteína A conjugada con HRP. Determinaciones de ELISA de títulos individuales y promedio de IgG, IgG1 e IgG2c circulantes anti-RBD utilizando IgG1, anti-IgG2 y anti-IgG2c de cabra conjugada con HRP. Los títulos medios de IgG anti-S y anti-RBD son 1:2000. La relación IgG2c/IgG1 es de 750 en los animales vacunados con respecto al grupo control de pPAL. (B) El promedio del título de anticuerpos neutralizantes es x100. La inferencia estadística se realizó mediante la prueba t de Student aplicando el ajuste del valor p por el método de Holm-Sidak ( = 0,05; *p<0,05; **p<0,01; ***p<0,001).
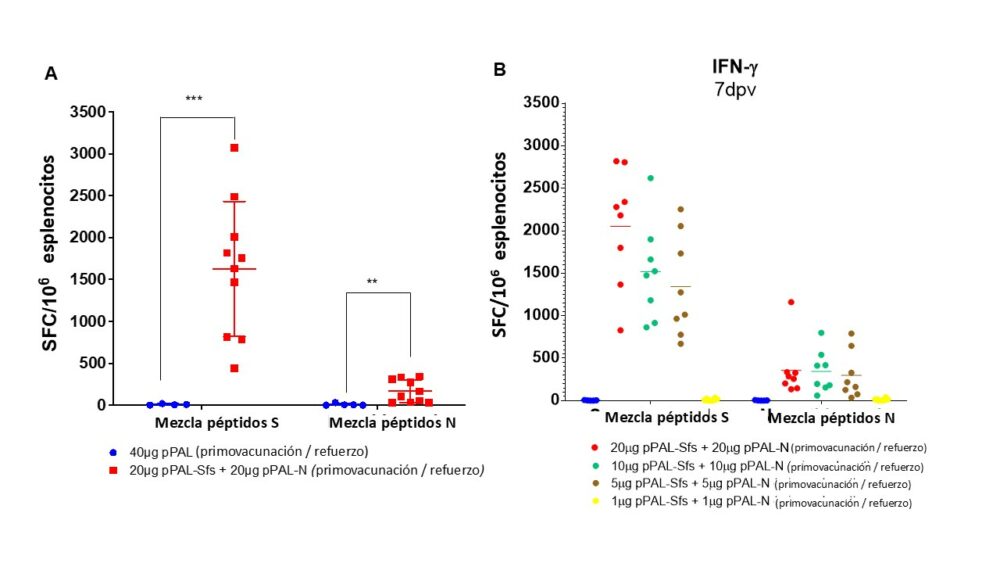
En el ensayo ELISpot, todos los ratones vacunados presentaron más de 500 colonias formadoras de Spot Forming Colonies (SFC) productoras de IFN-γ por millón de esplenocitos al día 7 tras el refuerzo, específicamente frente a un pool de péptidos de 11 residuos solapados que cubre toda la proteína S del virus. El promedio fue de ≈1.600, con un máximo de ≈3.100 (Figura 3A). Frente a la proteína N, la media fue mucho menor (≈200 SFC/millón) (Figura 3A). Un segundo ensayo ELISpot independiente confirmó los altos niveles de producción de IFN-g y mostró una respuesta dosis-dependiente con >2.000, ≈1.500, ≈1.300 y 0 SFC/millón tras inmunización con 20, 10, 5 y 1 μg de cada plásmido, respectivamente (Figura 3B).
Figura 3. La activación específica de S y N de los esplenocitos productores de IFN-γ inducida por el candidato vacunal pPAL-Sfs + pPAL-N en ratones C57BL/6J depende de la dosis. Los esplenocitos se obtuvieron 7 días después del refuerzo después de la inoculación de pPAL y pPAL-Sfs + pPAL-N. Los esplenocitos (2×105 células/pocillo) se estimularon a 37 ºC en placas ELISpot de IFN-γ durante 16 h con grupos de péptidos superpuestos de 8 aminoácidos de 11-mer (25 μg/pocillo) que representan todas las secuencias de aminoácidos S y N en cantidades equimolares. Los controles de estimulación negativos se restaron de los valores de SFC por millón de células. La inferencia estadística se realizó mediante la prueba t de Student, aplicando el ajuste del valor p por el método de Holm-Sidak (= 0,05; *p<0,05; **p<0,01; ***p<0,001). (A) Se observa una fuerte activación específica de S y N de los esplenocitos productores de IFN- con un régimen de inmunización de cebado/refuerzo con pPAL-Sfs + pPAL-N. (B) La activación específica de S de los esplenocitos productores de IFN- con pPAL-Sfs + pPAL-N depende de la dosis.
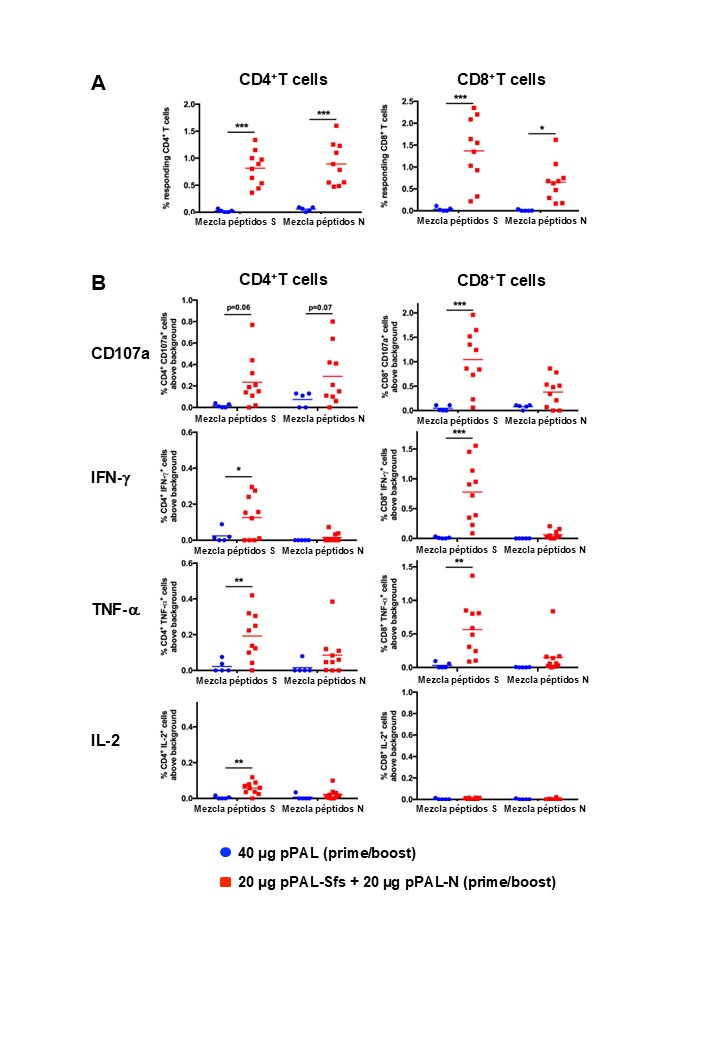
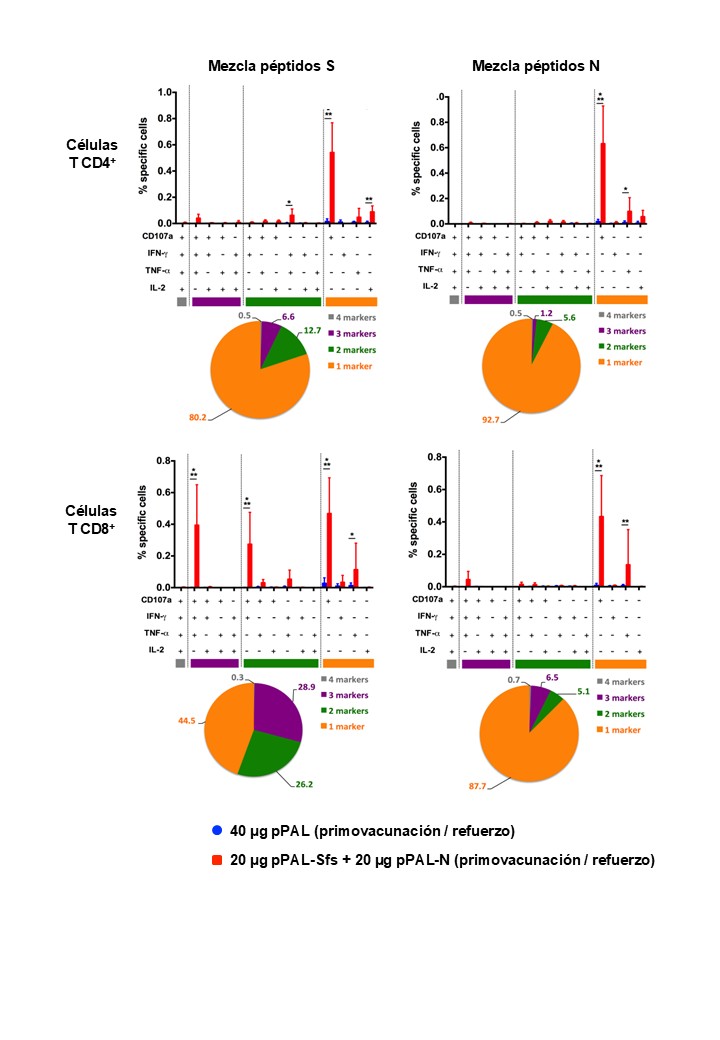
Mediante citometría de flujo con tinción intracelular de citoquinas (ICS), se evaluó la activación de linfocitos CD4⁺ y CD8⁺ frente a los pools de péptidos de S y N. Todos los ratones vacunados produjeron linfocitos CD4⁺ y CD8⁺ específicos para S y N (Figura 4A). Se observaron células activadas positivas para CD107a, IFN-γ, IL-2 y TNF-α, excepto IL-2 en CD8⁺ frente a N (Figura 4B). La activación frente a N fue inferior, salvo CD107a en CD4⁺. No se detectó activación para IFN-g ni IL-2 frente a N.
Figura 4. Respuesta específica de linfocitos T CD3+CD4+ y CD3+CD8+ inducida por pPAL-Sfs + pPAL-N en ratones C57BL/6J. Los esplenocitos se obtuvieron de ratones después de la inoculación de primoinfección/refuerzo con pPAL y pPAL-Sfs + pPAL-N. Los esplenocitos se estimularon durante 4 h con grupos de péptidos superpuestos de 8 aminoácidos de 11 dodecámeros que representaban todas las secuencias de aminoácidos S y N en cantidades equimolares. (A) Porcentaje de linfocitos T CD3+CD4+ y CD3+CD8+ que producen CD107a, IFN-γ, TNF-α e IL-2 en respuesta a la estimulación con grupos de péptidos S o N. (B) Porcentaje de linfocitos T CD4+ y CD8+ que responden a los grupos de péptidos S y N por encima del fondo, calculado como la suma de los linfocitos T positivos para CD107a y/o IFN-γ y/o TNF-α y/o IL-2. Inferencia estadística utilizando ANOVA de dos vías con la prueba post-hoc de LD de Fisher (*p<0.05; **p<0.01; ***p<0.001).
Las células CD8⁺ mostraron mayor polifuncionalidad que las CD4⁺. Aproximadamente el 30% de los linfocitos CD8⁺ fueron positivos para tres marcadores (Figura 5). Las células CD8⁺ específicas de S que expresaban CD107a, IFN-γ y TNF-α fueron las más abundantes.
En conjunto, la vacuna pPAL-Sfs + pPAL-N indujo una activación robusta de linfocitos T tipo Th1, CD4⁺ citotóxicos y CD8⁺ citotóxicos polifuncionales. La presencia de CD4⁺ citotóxicos es coherente con estudios previos.
Figura 5. Análisis de polifuncionalidad de poblaciones específicas de linfocitos T CD3+CD4+ y CD3+CD8+ estimuladas por la inmunización con pPAL-Sfs + pPAL-N en ratones C57BL/6J. Análisis de la expresión concomitante de CD107a, IFN-γ, TNF-α e IL-2 en linfocitos T CD3+CD4+ y linfocitos T CD3+CD8+ en respuesta a la estimulación del grupo de péptidos N o del grupo de péptidos S. Los gráficos circulares dividen el porcentaje de células T que expresan marcadores 1, 2, 3 o 4 tras la estimulación. Inferencia estadística utilizando ANOVA de dos vías con la prueba post-hoc de LD de Fisher (*p<0.05; **p<0.01; ***p<0.001).
3.3 La vacunación con pPAL-Sfs + pPAL-N confiere protección completa en ratones K18-hACE2
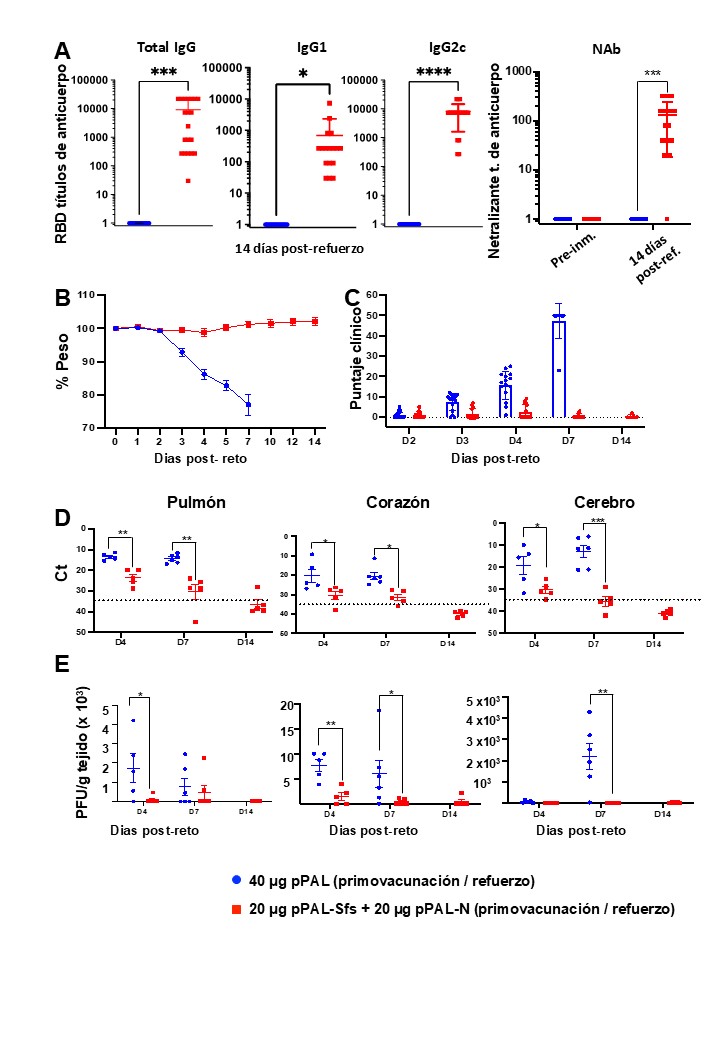
Para evaluar la eficacia protectora, se inmunizaron ratones transgénicos K18-hACE2 con el mismo protocolo. A los 15 días del refuerzo, todos los ratones vacunados presentaban títulos elevados de IgG total anti-RBD, IgG1 e IgG2c (≈10.000, ≈600 y ≈7.000, respectivamente), con una razón IgG2c/IgG1 de ≈12 (Figura 6A). Los títulos de neutralización también fueron elevados, salvo en un ratón. Tras el desafío intranasal con una dosis letal (10⁵ PFU) de la cepa MAD6, los ratones vacunados no perdieron peso, a diferencia de los controles, que comenzaron a perder peso a partir del día 3 (Figura 6B). Los signos clínicos también se redujeron significativamente (Figura 6C), y todos los controles debieron ser sacrificados antes del día 7. Subgrupos de cinco ratones se sacrificaron en los días 4, 7 y 14 post-desafío para evaluar carga viral por qRT-PCR y ensayo de placas en pulmón, corazón y cerebro. Los ratones vacunados presentaron una reducción significativa de ARN viral en todos los órganos (Figura 6D). En la mayoría, no se detectó ARN en día 14.
Los niveles de virus infeccioso fueron indetectables en la mayoría de los tejidos de los ratones vacunados (Figura 6E). Solo 2 de 5 ratones presentaron virus en pulmón (día 7), y 3 de 5 en corazón (día 4), con cargas significativamente menores (2,2 × 10³ vs 9 × 10³ PFU/g).
En el cerebro, el virus solo fue detectable en los controles. Estos datos indican que la vacunación controló completamente la replicación viral y previno la diseminación sistémica.
Figura 6. La vacuna pPAL-Sfs + pPAL-N confiere una protección total contra el desafío con 105 PFU del aislado MAD6 del SARS-CoV-2 en el modelo murino K18-hACE2. (A) Titulación de anticuerpos circulantes anti-RBD IgG, IgG1, IgG2c y neutralizantes 15 días después de la inmunización de cebado/refuerzo con pPAL-Sfs + pPAL-N. (B) Evolución del peso corporal después de 105 PFU SARS-CoV-2 desafío. Los animales vacunados mantuvieron su peso durante todo el experimento. El peso disminuyó en el grupo control hasta que se tuvo que aplicar el criterio de valoración (20% de pérdida de peso). (C) Seguimiento de signos clínicos después de la provocación. (D) Reducción de la carga viral en los órganos diana. (Ct que explica los niveles de ARNm del SARS-CoV-2. β-actina fue el gen de referencia. (E) Reducción de la replicación viral (PFU/g de tejido) en células VERO. La inferencia estadística se realizó mediante la prueba t de Student, aplicando el ajuste del valor p por el método de Holm-Sidak ((= 0,05; *p<0,05; **p<0,01; ***p<0,001).
3.4 La vacuna pPAL-Sfs + pPAL-N confiere inmunidad a largo plazo
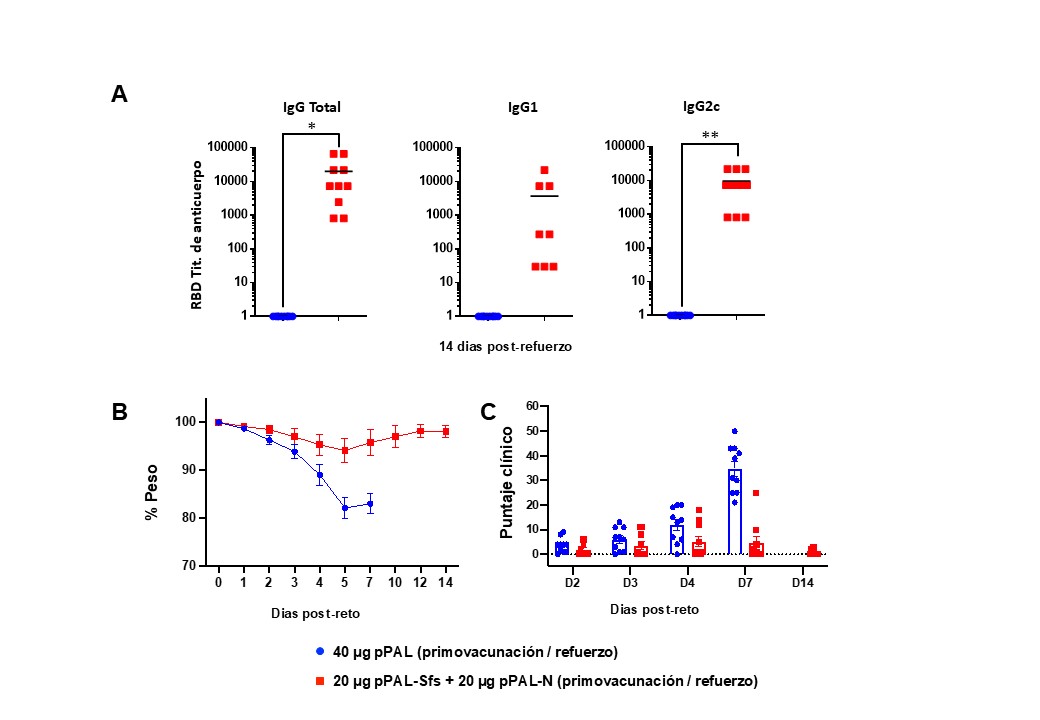
Para evaluar la memoria inmunológica, se vacunaron ratones K18-hACE2 con dos dosis separadas por 15 días y se desafiaron con SARS-CoV-2 tres meses después. Los títulos de anticuerpos seguían siendo significativamente elevados (Figura 7A), con predominio de IgG2c, indicando una respuesta Th1. Los ratones control perdieron peso progresivamente hasta ser sacrificados a los 7 días post-desafío, mientras que los vacunados mantuvieron su peso (Figura 7B). La puntuación clínica fue también significativamente menor en el grupo vacunado (Figura 7C), lo que confirma que la vacuna confiere inmunidad duradera.
Figura 7. Protección completa de ratones K18-hACE2 después de tres meses de vacunación con pPAL-Sfs+ pPAL-N contra el desafío con 105 PFU del aislado MAD6 SARS-CoV-2 en el modelo murino K18-hACE2. (A) Titulación de las IgG, IgG1 e IgG2c anti-RBD circulantes tres meses después de la administración de la dosis de refuerzo pPAL-Sfs + pPAL-N. (B) Evolución del peso corporal después de 10⁵ PFU SARS-CoV-2 desafío. Los animales vacunados mantuvieron su peso durante todo el experimento, mientras que el peso disminuyó en el grupo de control. (C) Seguimiento de signos clínicos tras provocación con 10⁵ PFU SARS-CoV-2. La inferencia estadística se realizó mediante la prueba t de Student, aplicando el ajuste del valor p por el método de Holm-Sidak (( = 0,05; *p<0,05; **p<0,01; ***p<0,001).
3.5 La vacunación con pPAL-Sfs + pPAL-N es eficaz frente a la variante dominante B.1.617.2 (Delta)
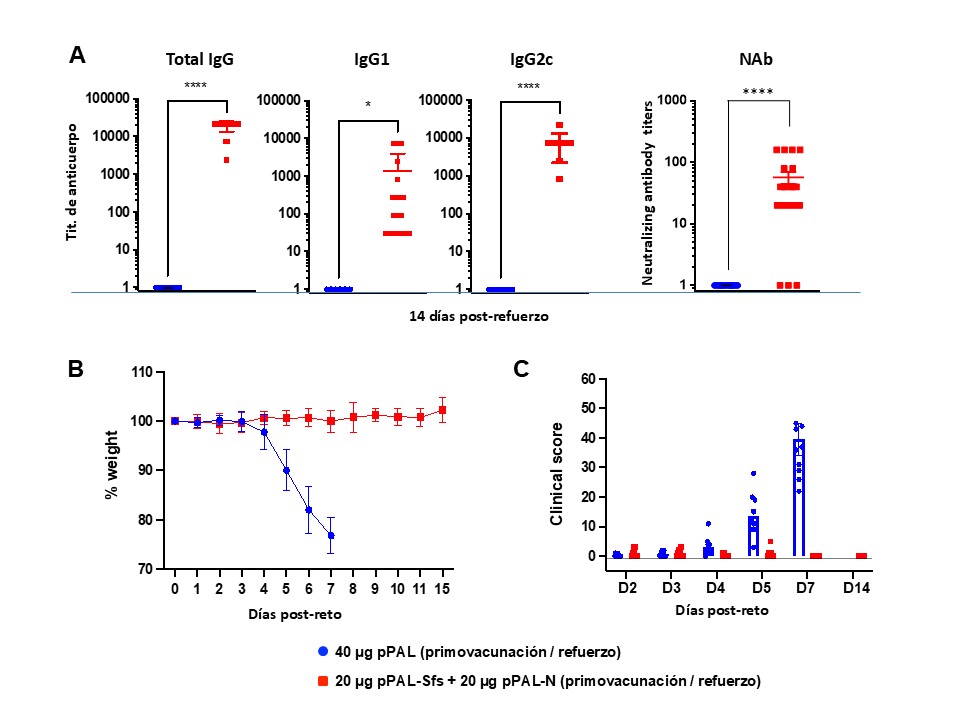
Se probó la eficacia del mismo esquema de vacunación frente a un desafío letal con 10⁵ PFU de la variante Delta. Los títulos de IgG anti-RBD, IgG1 e IgG2c fueron elevados (>10.000, ≈1.000 y ≈8.000, respectivamente), con una razón IgG2c/IgG1 de ~8, reflejando una respuesta Th1 (Figura 8A). Tras el desafío, los ratones vacunados no perdieron peso, mientras que los controles mostraron una pérdida progresiva (Figura 8B). Los signos clínicos fueron leves o inexistentes en los vacunados (Figura 8C), lo que indica que la vacuna pPAL-Sfs + pPAL-N protegió eficazmente frente a la variante Delta.
Figura 8. La vacuna pPAL-Sfs + pPAL-N confiere una protección completa contra el desafío con 10⁵ PFU de la variante Delta del SARS-CoV-2 en el modelo murino K18-hACE2. (A) Titulación de anticuerpos circulantes anti-RBD IgG, IgG2c y neutralizantes 15 días después de la inmunización de cebado/refuerzo con pPAL-Sfs + pPAL-N. (B) Evolución del peso corporal después de 10⁵ PFU SARS-CoV-2 desafío. Los animales vacunados mantuvieron su peso durante todo el experimento, mientras que el peso disminuyó en el grupo de control. (C) Seguimiento de signos clínicos tras provocación con 10⁵ PFU SARS-CoV-2. La inferencia estadística se realizó mediante la prueba t de Student, aplicando el ajuste del valor p por el método de Holm-Sidak (( = 0,05; *p<0,05; **p<0,01; ***p<0,001).
4. DISCUSIÓN
La pandemia de COVID-19 continúa siendo una preocupación global, a pesar de que una alta proporción de individuos en países desarrollados ha sido vacunada con vacunas que protegen frente a síntomas graves. Las vacunas actuales están basadas en el antígeno S, administrado mediante vectores adenovirales, ARNm o formulaciones de proteínas recombinantes. La mayoría son eficaces para reducir la gravedad de la enfermedad (22).
Las vacunas de ARNm, usualmente vehiculadas en nanopartículas lipídicas, inducen altos niveles de protección (23–27), pero requieren cadena de frío estricta (–80 o –20 ºC), lo cual dificulta su distribución en países en desarrollo. Estas vacunas pueden causar efectos adversos raros como anafilaxia y miocarditis (22). Las vacunas basadas en vectores virales no replicativos también requieren refrigeración (2–8 ºC) y pueden provocar efectos adversos poco frecuentes como trombosis con trombocitopenia, síndrome de Guillain-Barré y respuestas proinflamatorias (22,28). Las vacunas de segunda generación, basadas en proteínas recombinantes con adyuvantes, inducen respuestas humorales específicas elevadas pero baja activación de células T (29,30). Aunque las vacunas existentes protegen frente a la enfermedad grave, variantes dominantes como Ómicron (B.1.1.529) presentan menor sensibilidad a estas vacunas (5). Dosis adicionales difícilmente resolverán el problema del escape vacunal (31).
Se requieren nuevas vacunas que protejan frente a un espectro más amplio de variantes preocupantes (VOC). Las vacunas de ADN son una opción prometedora por su facilidad de modificación ante nuevas variantes, bajo coste, manufactura sencilla y termoestabilidad, lo que facilita su distribución global (32). Son especialmente adecuadas para países con recursos limitados, donde no se dispone de cadena de frío (12).
En este estudio presentamos una vacuna de ADN contra SARS-CoV-2 administrada por vía intramuscular seguida de electroporación in vivo. Esta vacuna induce una potente respuesta inmune humoral y celular específica frente al virus, que protege a ratones frente a desafíos letales tanto con la cepa Wuhan como con la variante Delta.
La proteína N es una proteína estructural crítica en coronavirus (33,34). Su estabilidad genética y conservación la convierten en una candidata vacunal adecuada (34). El gen S fue modificado (Sfs) para evitar el corte por furina. Células HEK293 transfectadas con pPAL-Sfs + pPAL-N expresaron adecuadamente las versiones optimizadas de los genes Sfs y N.
Ratones C57BL/6J vacunados con dos dosis de pPAL-Sfs + pPAL-N mediante electroporación produjeron una robusta respuesta humoral con IgGs específicas frente a la proteína S y su dominio RBD. El análisis de subclases mostró predominancia de IgG2c, lo que indica una respuesta Th1. Este perfil Th1 favorece el aclaramiento viral (35), mientras que un sesgo Th2 se ha asociado con enfermedad severa (36, 7). También se generaron anticuerpos neutralizantes, lo que sugiere una protección efectiva (38,39).
Las respuestas de células T también son esenciales frente a la infección por SARS-CoV-2. Se ha observado correlación entre recuperación y fuerte respuesta T, especialmente CD8⁺ (40–42). La vacunación con pPAL-Sfs + pPAL-N activó linfocitos T específicos frente a los antígenos S y N. Se evaluaron cuatro marcadores funcionales: CD107a, IFN-g, IL-2 y TNF-a. Se detectaron células CD4⁺ y CD8⁺ citotóxicas, siendo estas últimas más polifuncionales. Más del 50% de las CD8⁺ específicas de S expresaban dos o más citoquinas, lo cual es indicativo de efectores polifuncionales (44).
En cambio, las CD8⁺ específicas de N mostraron menor polifuncionalidad y fenotipo puramente citotóxico. Las CD4⁺ frente a S y N también expresaron CD107a, indicando capacidad citotóxica. La presencia de CD4⁺ citotóxicas específicas para SARS-CoV-2 ha sido reportada y se asocia con protección en infecciones como la fiebre del Nilo Occidental (43).
Una cuestión interesante es el papel de los anticuerpos anti-N no neutralizantes, que pueden facilitar el aclaramiento viral en virus envueltos (45–47). Se ha descrito recientemente que la ligasa E3 TRIM21 puede dirigir la proteína N hacia la degradación proteasomal mediante estos anticuerpos, estimulando respuestas T citotóxicas efectivas (15). Esto apoya la inclusión del gen N en la formulación vacunal.
La vacuna pPAL-Sfs + pPAL-N protegió frente a la cepa Wuhan y la variante Delta. La qRT-PCR y los ensayos de PFU demostraron que la vacuna previno la diseminación viral hacia órganos como el corazón y el cerebro, implicados en efectos secundarios graves de la COVID-19 (19). La protección fue completa incluso en animales con títulos bajos de anticuerpos específicos.
Además, esta protección se mantuvo tres meses después de la vacunación.
Por tanto, la vacuna induce inmunidad duradera y protección completa frente a SARS-CoV-2. Está lista para avanzar a ensayos clínicos en humanos.
5. CONCLUSIONES
En resumen, la administración homóloga de pPAL-Sfs + pPAL-N (20 μg de cada plásmido por dosis), disuelto en agua estéril y administrado por vía intramuscular con electroporación in vivo, confiere protección completa frente a un desafío con 10⁵ PFU del aislado Wuhan-Hu-1 (MAD6) y de la variante Delta. La vacuna induce una respuesta Th1, linfocitos T CD8⁺ polifuncionales citotóxicos y producción de anticuerpos neutralizantes. Esta vacuna de ADN es segura, fácil de producir a escala industrial y apta para distribución y almacenamiento a temperatura ambiente.
6. REFERENCIAS
- Richner JM, Himansu S, Dowd KA, Butler SL, Salazar V, Fox JM, et al. Modified mRNA Vaccines Protect against Zika Virus Infection. Cell. 2017;169(1):176.
- Pardi N, Hogan MJ, Pelc RS, Muramatsu H, Andersen H, DeMaso CR, et al. Zika virus protection by a single low-dose nucleoside-modified mRNA vaccination. Nature. 2017;543(7644):248-51.
- Reynolds CJ, Gibbons JM, Pade C, Lin KM, Sandoval DM, Pieper F, et al. Heterologous infection and vaccination shapes immunity against SARS-CoV-2 variants. Science. 2022;375(6577):183-92.
- Muik A, Lui BG, Wallisch AK, Bacher M, Muhl J, Reinholz J, et al. Neutralization of SARS-CoV-2 Omicron by BNT162b2 mRNA vaccine-elicited human sera. Science. 2022;375(6581):678-80.
- Mannar D, Saville JW, Zhu X, Srivastava SS, Berezuk AM, Tuttle KS, et al. SARS-CoV-2 Omicron variant: Antibody evasion and cryo-EM structure of spike protein-ACE2 complex. Science. 2022;375(6582):760-4.
- Ledford H. ‘Killer’ immune cells still recognize Omicron variant. Nature. 2022;601(7893):307.
- Toh ZQ, Anderson J, Mazarakis N, Neeland M, Higgins RA, Rautenbacher K, et al. Comparison of Seroconversion in Children and Adults With Mild COVID-19. JAMA Netw Open. 2022;5(3):e221313.
- Choi SJ, Kim DU, Noh JY, Kim S, Park SH, Jeong HW, et al. T cell epitopes in SARS-CoV-2 proteins are substantially conserved in the Omicron variant. Cell Mol Immunol. 2022;19(3):447-8.
- He X, Aid M, Chandrashekar A, Yu J, McMahan K, Wegmann F, et al. A homologous or variant booster vaccine after Ad26.COV2.S immunization enhances SARS-CoV-2-specific immune responses in rhesus macaques. Sci Transl Med. 2022;14(638):eabm4996.
- Altmann DM, Boyton RJ. COVID-19 vaccination: The road ahead. Science. 2022;375(6585):1127-32.
- Tebas P, Kraynyak KA, Patel A, Maslow JN, Morrow MP, Sylvester AJ, et al. Intradermal SynCon(R) Ebola GP DNA Vaccine Is Temperature Stable and Safely Demonstrates Cellular and Humoral Immunogenicity Advantages in Healthy Volunteers. J Infect Dis. 2019;220(3):400-10.
- Momin T, Kansagra K, Patel H, Sharma S, Sharma B, Patel J, et al. Safety and Immunogenicity of a DNA SARS-CoV-2 vaccine (ZyCoV-D): Results of an open-label, non-randomized phase I part of phase I/II clinical study by intradermal route in healthy subjects in India. EClinicalMedicine. 2021;38:101020.
- Alcolea PJ, Alonso A, Larraga V. The antibiotic resistance-free mammalian expression plasmid vector pPAL for development of third generation vaccines. Plasmid. 2019;101:35-42.
- Scialo F, Daniele A, Amato F, Pastore L, Matera MG, Cazzola M, et al. ACE2: The Major Cell Entry Receptor for SARS-CoV-2. Lung. 2020;198(6):867-77.
- Gussow AB, Auslander N, Faure G, Wolf YI, Zhang F, Koonin EV. Genomic determinants of pathogenicity in SARS-CoV-2 and other human coronaviruses. Proc Natl Acad Sci U S A. 2020;117(26):15193-9.
- Caddy SL, Vaysburd M, Papa G, Wing M, O’Connell K, Stoycheva D, et al. Viral nucleoprotein antibodies activate TRIM21 and induce T cell immunity. EMBO J. 2021;40(5):e106228.
- Winkler ES, Bailey AL, Kafai NM, Nair S, McCune BT, Yu J, et al. SARS-CoV-2 infection of human ACE2-transgenic mice causes severe lung inflammation and impaired function. Nat Immunol. 2020;21(11):1327-35.
- Toussaint JF, Sailleau C, Breard E, Zientara S, De Clercq K. Bluetongue virus detection by two real-time RT-qPCRs targeting two different genomic segments. J Virol Methods. 2007;140(1-2):115-23.
- Collier AY, Brown CM, McMahan KA, Yu J, Liu J, Jacob-Dolan C, et al. Characterization of immune responses in fully vaccinated individuals after breakthrough infection with the SARS-CoV-2 delta variant. Sci Transl Med. 2022;14(641):eabn6150.
- Zhuang Z, Lai X, Sun J, Chen Z, Zhang Z, Dai J, et al. Correction: Mapping and role of T cell response in SARS-CoV-2-infected mice. J Exp Med. 2021;218(11).
- Goel RR, Painter MM, Apostolidis SA, Mathew D, Meng W, Rosenfeld AM, et al. mRNA vaccines induce durable immune memory to SARS-CoV-2 and variants of concern. Science. 2021;374(6572):abm0829.
- Fiolet T, Kherabi Y, MacDonald CJ, Ghosn J, Peiffer-Smadja N. Comparing COVID-19 vaccines for their characteristics, efficacy and effectiveness against SARS-CoV-2 and variants of concern: a narrative review. Clin Microbiol Infect. 2022;28(2):202-21.
- Baden LR, El Sahly HM, Essink B, Kotloff K, Frey S, Novak R, et al. Efficacy and Safety of the mRNA-1273 SARS-CoV-2 Vaccine. N Engl J Med. 2021;384(5):403-16.
- El Sahly HM, Baden LR, Essink B, Doblecki-Lewis S, Martin JM, Anderson EJ, et al. Efficacy of the mRNA-1273 SARS-CoV-2 Vaccine at Completion of Blinded Phase. N Engl J Med. 2021;385(19):1774-85.
- Frenck RW, Jr., Klein NP, Kitchin N, Gurtman A, Absalon J, Lockhart S, et al. Safety, Immunogenicity, and Efficacy of the BNT162b2 Covid-19 Vaccine in Adolescents. N Engl J Med. 2021;385(3):239-50.
- Polack FP, Thomas SJ, Kitchin N, Absalon J, Gurtman A, Lockhart S, et al. Safety and Efficacy of the BNT162b2 mRNA Covid-19 Vaccine. N Engl J Med. 2020;383(27):2603-15.
- Thomas SJ, Perez JL, Lockhart SP, Hariharan S, Kitchin N, Bailey R, et al. Efficacy and safety of the BNT162b2 mRNA COVID-19 vaccine in participants with a history of cancer: subgroup analysis of a global phase 3 randomized clinical trial. Vaccine. 2022;40(10):1483-92.
- Ahmed A, Nezami M, Alkattan A. Pitfalls at Chemistry of Adenoviral Vector Vaccine against COVID-19 and How to Circumvent it. Adv Pharm Bull. 2022;12(2):217-8.
- He Q, Mao Q, Peng X, He Z, Lu S, Zhang J, et al. Immunogenicity and protective efficacy of a recombinant protein subunit vaccine and an inactivated vaccine against SARS-CoV-2 variants in non-human primates. Signal Transduct Target Ther. 2022;7(1):69.
- Gurunathan S, Klinman DM, Seder RA. DNA vaccines: immunology, application, and optimization*. Annu Rev Immunol. 2000;18:927-74.
- Waltz E. Omicron-targeted vaccines do no better than original jabs in early tests. Nature. 2022.
- Mallapaty S. India’s DNA COVID vaccine is a world first – more are coming. Nature. 2021;597(7875):161-2.
- Malone B, Urakova N, Snijder EJ, Campbell EA. Structures and functions of coronavirus replication-transcription complexes and their relevance for SARS-CoV-2 drug design. Nat Rev Mol Cell Biol. 2022;23(1):21-39.
- Grifoni A, Sidney J, Zhang Y, Scheuermann RH, Peters B, Sette A. A Sequence Homology and Bioinformatic Approach Can Predict Candidate Targets for Immune Responses to SARS-CoV-2. Cell Host Microbe. 2020;27(4):671-80 e2.
- Gil-Etayo FJ, Garcinuno S, Utrero-Rico A, Cabrera-Marante O, Arroyo-Sanchez D, Mancebo E, et al. An Early Th1 Response Is a Key Factor for a Favorable COVID-19 Evolution. Biomedicines. 2022;10(2).
- Gutiérrez-Bautista JF, Rodriguez-Nicolas A, Rosales-Castillo A, Jimenez P, Garrido F, Anderson P, et al. Negative Clinical Evolution in COVID-19 Patients Is Frequently Accompanied With an Increased Proportion of Undifferentiated Th Cells and a Strong Underrepresentation of the Th1 Subset. Front Immunol. 2020;11:596553.
- Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395(10223):497-506.
- Khoury DS, Cromer D, Reynaldi A, Schlub TE, Wheatley AK, Juno JA, et al. Neutralizing antibody levels are highly predictive of immune protection from symptomatic SARS-CoV-2 infection. Nat Med. 2021;27(7):1205-11.
- Pollard AJ, Bijker EM. A guide to vaccinology: from basic principles to new developments. Nat Rev Immunol. 2021;21(2):83-100.
- Luo M, Liu J, Jiang W, Yue S, Liu H, Wei S. IL-6 and CD8+ T cell counts combined are an early predictor of in-hospital mortality of patients with COVID-19. JCI Insight. 2020;5(13).
- Diao B, Wang C, Tan Y, Chen X, Liu Y, Ning L, et al. Reduction and Functional Exhaustion of T Cells in Patients With Coronavirus Disease 2019 (COVID-19). Front Immunol. 2020;11:827.
- Hellerstein M. What are the roles of antibodies versus a durable, high quality T-cell response in protective immunity against SARS-CoV-2? Vaccine X. 2020;6:100076.
- Brien JD, Uhrlaub JL, Nikolich-Zugich J. West Nile virus-specific CD4 T cells exhibit direct antiviral cytokine secretion and cytotoxicity and are sufficient for antiviral protection. J Immunol. 2008;181(12):8568-75.
- Boyd A, Almeida JR, Darrah PA, Sauce D, Seder RA, Appay V, et al. Correction: Pathogen-Specific T Cell Polyfunctionality Is a Correlate of T Cell Efficacy and Immune Protection. PLoS One. 2015;10(9):e0138395.
- Mayr LM, Su B, Moog C. Non-Neutralizing Antibodies Directed against HIV and Their Functions. Front Immunol. 2017;8:1590.
- Bootz A, Karbach A, Spindler J, Kropff B, Reuter N, Sticht H, et al. Protective capacity of neutralizing and non-neutralizing antibodies against glycoprotein B of cytomegalovirus. PLoS Pathog. 2017;13(8):e1006601.
- Carragher DM, Kaminski DA, Moquin A, Hartson L, Randall TD. A novel role for non-neutralizing antibodies against nucleoprotein in facilitating resistance to influenza virus. J Immunol. 2008;181(6):4168-76.