Objetivo: Exponer los principales polimorfismos genéticos que han sido asociados a la respuesta del carcinoma de cabeza y cuello al cetuximab.
Método: Se realizó una revisión no exhaustiva de artículos publicados en el período de enero de 2000 a diciembre de 2022, para ello se emplearon las bases de datos Medline (vía Pubmed) y Science Direct. En la evaluación de la calidad metodológica de los artículos incluidos se utilizó la guía para los estudios de asociación genética (Q-Genie).
Resultados: Se identificaron un total de 206 artículos, de los cuales 12 cumplieron con los criterios para el análisis final.
Se reportaron varios polimorfismos, tales como: EGFR-R521K (AA/GA), FcγRIIIa (158VV) y FcγRIIa (131HH), KRASLCS6 (TG/GG), AKT2:rs8100018, PTEN: rs12569998 en sus variantes mutadas, HIF-1α (CT/TT) y XRCC5 (GG/AA) que se asociaron con las variables de supervivencia, riesgo de progresión, tiempos hasta la progresión de la enfermedad, así como toxicidad cutánea.
Conclusiones: Varios polimorfismos pueden asociarse con la respuesta del carcinoma de cabeza y cuello al tratamiento con cetuximab, siendo EGFR-R521K y FcγR IIIa-V158F los más estudiados. La enorme incertidumbre de los resultados alcanzados no permite alcanzar conclusiones firmes sobre la influencia de los polimorfismos genéticos en la respuesta al cetuximab; sin embargo, pueden convertirse en biomarcadores farmacogenéticos en la práctica clínica como una valiosa herramienta en la medicina personalizada, para predecir la respuesta medicamentosa. Para ello se requiere la realización de ensayos controlados con estratos por genotipo, con asignación aleatoria del tratamiento y el análisis de otras variables con valor pronóstico conocido.Palabras Clave: polimorfismos genéticos; cetuximab; carcinoma de cabeza y cuello
ABSTRACT:
Objective: To present the main genetic polymorphisms that have been associated with the response of head and neck carcinoma to cetuximab.
Method: A non-exhaustive review of articles published in the period from January 2000 to December 2022 was carried out, for this purpose the Medline (via Pubmed) and Science Direct databases were used. The guide for genetic association studies (Q-Genie) was used to evaluate the methodological quality of the included articles.
Results: A total of 206 articles were identified, of which 12 met the criteria for the final analysis. Several polymorphisms were reported, such as: EGFR-R521K (AA/GA), FcγRIIIa (158VV) and FcγRIIa (131HH), KRAS-LCS6 (TG/GG), AKT2:rs8100018, PTEN: rs12569998 in its mutated variants, HIF- 1α (CT/TT) and XRCC5 (GG/AA) that were associated with survival variables, risk of progression, times to disease progression, as well as skin toxicity.
Conclusions: Several polymorphisms can be associated with the response of head and neck carcinoma to treatment with cetuximab, being EGFR-R521K and FcγR IIIa-V158F the most studied. The enormous uncertainty of the results obtained does not allow firm conclusions to be reached about the influence of genetic polymorphisms on the response to cetuximab; however, they can become pharmacogenetic biomarkers in clinical practice as a valuable tool in personalized medicine, to predict drug response. This requires carrying out controlled trials with strata by genotype, with random assignment of treatment and the analysis of other variables with known prognostic value.
Keywords: genetic polymorphism; cetuximab; head and neck neoplasms
1. INTRODUCCIÓN
Los carcinomas de cabeza y cuello (CCC) constituyen un grupo heterogéneo de tumores que se localizan en labio, cavidad oral, orofaringe, nasofaringe, hipofaringe, laringe, cavidad nasal y
senos paranasales, así como las glándulas salivales. La mayor frecuencia de estos tumores es de etiología epitelial (>90%) y el carcinoma escamoso es el tipo histológico más frecuente (1,2).
Entre todos los tipos de cáncer, los CCC representan la sexta neoplasia más frecuente a nivel mundial y séptima en cuanto a mortalidad. Cada año se diagnostican unos 600 000 casos nuevos y
ocurren aproximadamente unos 300 000 fallecimientos anuales por esta causa (3,4).
Los CCC se caracterizan por ser agresivos, genéticamente complejos y el tratamiento de los pacientes que lo padecen es complicado (5). Generalmente, los pacientes en estadios iniciales (I
y II) al diagnóstico se tratan con una sola modalidad terapéutica [cirugía o radioterapia (RT)]. En los pacientes en estadios avanzados (III-IV) se realiza una terapia multimodal secuencial que engloba cirugía, RT y quimioterapia (QT) (6).
Más del 80% de los casos de CCC se diagnostican con enfermedad avanzada. A pesar de los avances en el tratamiento multimodal, más de la mitad de los pacientes no se curan y recaen localmente o en sitios distantes en algún momento de su evolución.
De estos, sólo un número muy limitado de pacientes son candidatos para cirugía o re-irradiación. Los pacientes no aptos para dicho tratamiento, suelen recibir QT con carácter paliativo. El pronóstico es desfavorable, pues la supervivencia global (SG) esperada en pacientes con cáncer epidermoide recurrente y/o metastásico se encuentra en torno a los 6-12 meses (7,8).
La incorporación de los anticuerpos monoclonales (AcM) específicamente dirigidos al receptor del factor de crecimiento epidérmico (EGFR) en el tratamiento del carcinoma de células escamosas de cabeza y cuello (CCECC) en estadios avanzados, en combinación con los citotóxicos clásicos y/o RT demostró una mejoría significativa en el control locorregional (CLR) y en la supervivencia de los pacientes (9,10). El EGFR se sobreexpresa en el 90% o más de los casos de CCECC y está asociado con un mal pronóstico y resistencia a los tratamientos oncológicos convencionales (11,12).
El cetuximab es un AcM quimérico de inmunoglobulina G1 (IgG1), antagonista del EGFR (13,14). Fue aprobado en el 2006 por la Agencia Reguladora de Drogas y Alimentos de los Estados Unidos (FDA, siglas en inglés) para el tratamiento del CCECC localmente avanzado, de forma concomitante con RT (15,16). Posteriormente en el ensayo clínico (EC) fase III EXTREME, se comprobó que la adición de cetuximab a la combinación de cisplatino y 5-fluorouracilo (5-FU) seguido de cetuximab como tratamiento de mantenimiento, aumentaba la mediana de supervivencia libre de progresión (SLP) de 3,3 a 5,6 meses y la mediana de SG de 7,4 a 10,1 meses en comparación con la QT sola, con tasas de respuesta general de 20 a 36% y tasas de control de la enfermedad (TCE) superiores al 80%, convirtiéndose en el “estándar de oro” en el tratamiento de primera línea en el CCECC recurrente y/o metastásico (R/M), por ello la FDA aprobó este esquema en el 2011 (17).
A pesar de los prometedores resultados obtenidos en los EC en la práctica clínica se ha constatado que sólo una minoría de los pacientes tratados se beneficia de la terapia con cetuximab y aquellos que inicialmente logran una respuesta adecuada eventualmente manifiestan progresión de la enfermedad (18). La literatura refiere que la eficacia del tratamiento con cetuximab es baja, con una tasa de respuesta objetiva de un 13% en monoterapia y un 36% en combinación con QT (19). Los datos sugieren que los mecanismos de resistencia intrínseca y adquirida han limitado considerablemente el beneficio de la terapia con cetuximab en el CCECC, por tal razón, la comprensión de los mecanismos moleculares de resistencia reviste un enorme interés clínico en la actualidad y representa uno de los mayores desafíos en el tratamiento de esta enfermedad (19,20).
Se han investigado las posibles transformaciones y vías moleculares implicadas en la resistencia al tratamiento con cetuximab en el CCECC. Entre los mecanismos estudiados se reportan: las alteraciones de la unión EGFR-ligando y de los efectores de señalización aguas abajo del EGFR, así como las vías RAS/MAPK y PI3K/Akt/mTOR, activación de vías paralelas para contrarrestar el bloqueo de la señalización del EGFR, cambios en las proteínas implicadas en los procesos de proliferación, apoptosis, invasión y metástasis como la proteína supresora de tumores p53. La transición epitelial a mesenquimal (EMT), alteraciones genéticas, epigenéticas y el establecimiento de un microambiente tumoral inmunosupresor (21). Hasta la fecha ninguna de estas teorías han sido validadas y no se han realizado suficientes EC que permitan confirmar su utilidad clínica.
Dada la naturaleza inmunológica del cetuximab, su eficacia puede verse afectada por variaciones en los genes responsables de su reconocimiento, presentación y metabolismo, lo cual puede generar variabilidad entre los pacientes (22), es por ello que los polimorfismos genéticos se proponen como uno de los mecanismos de resistencia al tratamiento con el AcM (21), los que se definen como variaciones en la secuencia del ácido desoxirribonucleico (ADN) que aparecen con una frecuencia alélica del 1% o más en la población. Los más frecuentes son los polimorfismos de nucleótido simple o único (SNP) que consisten en la sustitución de un nucleótido por otro (23). Los SNP en la región codificante de un gen a menudo provocan cambios en los aminoácidos, que pueden alterar el funcionamiento de las proteínas afectadas. En la literatura se describe que algunos SNP se correlacionan con la respuesta de los AcM anti-EGFR como el cetuximab (24).
El conocimiento de los mecanismos de resistencia permitirá la identificación de biomarcadores que pueden utilizarse en la práctica clínica para predecir la respuesta a este AcM y de este modo seleccionar adecuadamente a los pacientes con mayor probabilidad de beneficiarse con el cetuximab (18).
Teniendo en cuenta que hasta la fecha no se ha validado un biomarcador molecular predictivo de eficacia para el cetuximab en el CCECC y que se han publicado varios estudios que evalúan la posible aplicación clínica de los SNP en la predicción de la respuesta al tratamiento con este AcM, se propone realizar una revisión aunque no exhaustiva de la literatura para exponer la evidencia científica sobre los principales polimorfismos genéticos que han sido asociados a la respuesta del CCECC al cetuximab.
2. MÉTODOS
Se realizó una revisión en las bases de datos Medline (vía Pubmed) y Science Direct de artículos publicados en el período de enero de 2000 a diciembre de 2022. Para definir los términos de búsqueda se empleó el Medical Subject Headings (MeSH). La sintaxis de búsqueda en las bases de datos fue la siguiente: ((squamous cell carcinoma of head and neck[MeSH Terms]) OR (head and neck neoplasms[MeSH Terms])) AND (cetuximab[MeSH Terms]) AND ((polymorphism, single nucleotide[MeSH Terms]) OR (polymorphism genetics[MeSH Terms])).
Se aplicaron diversos filtros para la realización de la búsqueda. En Pubmed se restringieron los resultados a “estudios clínicos”, “ensayos clínicos”, “ensayos clínicos randomizados” y “estudios observacionales”. En el caso de Science Direct la búsqueda se restringió a artículos originales. Para la identificación de los artículos se verificó que los términos de búsqueda se encontraran en el título o en el resumen.
2.1. Criterios de elegibilidad e identificación del estudio
Se incluyeron los estudios originales con acceso a texto completo, realizados exclusivamente en pacientes con CCECC que recibieran tratamiento con cetuximab en monoterapia o en combinación con RT y/o QT y que evaluaran la asociación entre los diferentes polimorfismos y la respuesta clínica obtenida. Se excluyeron los estudios realizados in vitro, los trabajos duplicados, los publicados en un idioma diferente al inglés, así como aquellos que en el título/ resumen no presentaran los términos de búsqueda. En el caso de los estudios que no estuvieran disponibles a texto
completo se contactó con los autores para acceder a toda la información. Seleccionados los estudios que cumplían con los criterios de inclusión, se revisaron sus citas y también se examinaron los artículos similares sugeridos por el buscador para encontrar trabajos potencialmente relevantes no identificados por la búsqueda sistemática anterior.
2.2. Extracción de datos y evaluación de la calidad metodológica
Los datos de los estudios incluidos fueron extraídos de forma independiente según los criterios anteriormente mencionados. De cada estudio se registró la siguiente información: apellido del primer autor y año de publicación, número de pacientes incluidos, diseño del estudio, esquema de tratamiento aplicado, gen estudiado, polimorfismo identificado, método de discriminación alélica, estadio del tumor, variables clínicas evaluadas, el país y la etnicidad de los pacientes.
Para la evaluación de la calidad metodológica de los estudios incluidos se utilizó la guía de los estudios de asociación genética (Q-Genie), en la cual cada ítem se califica en una escala de 7 puntos: “1 punto – pobre”; “2 y 3 puntos – bien”; “4, 5 y 6 puntos – muy bien” y “7 – excelente”. Para los estudios con grupo control, la calidad general se clasifica de la siguiente manera: las puntuaciones ≤35 indican estudios de mala calidad, >35 y ≤45 indican estudios de calidad moderada y >45 indican estudios de buena calidad. Para estudios sin grupos control, las puntuaciones
≤32 indican estudios de mala calidad, >32 y ≤40 indican estudios de calidad moderada, y >40 indican estudios de buena calidad (25,26).
Dos autores realizaron la extracción de datos y la evaluación de la calidad de cada estudio de forma independiente. Las discrepancias se resolvieron mediante discusión con la participación de un tercer autor hasta llegar a un consenso. Se calculó el coeficiente kappa de Cohen () para valorar el grado de concordancia en la selección de los artículos entre los autores.
3. RESULTADOS
3.1. Selección de los estudios
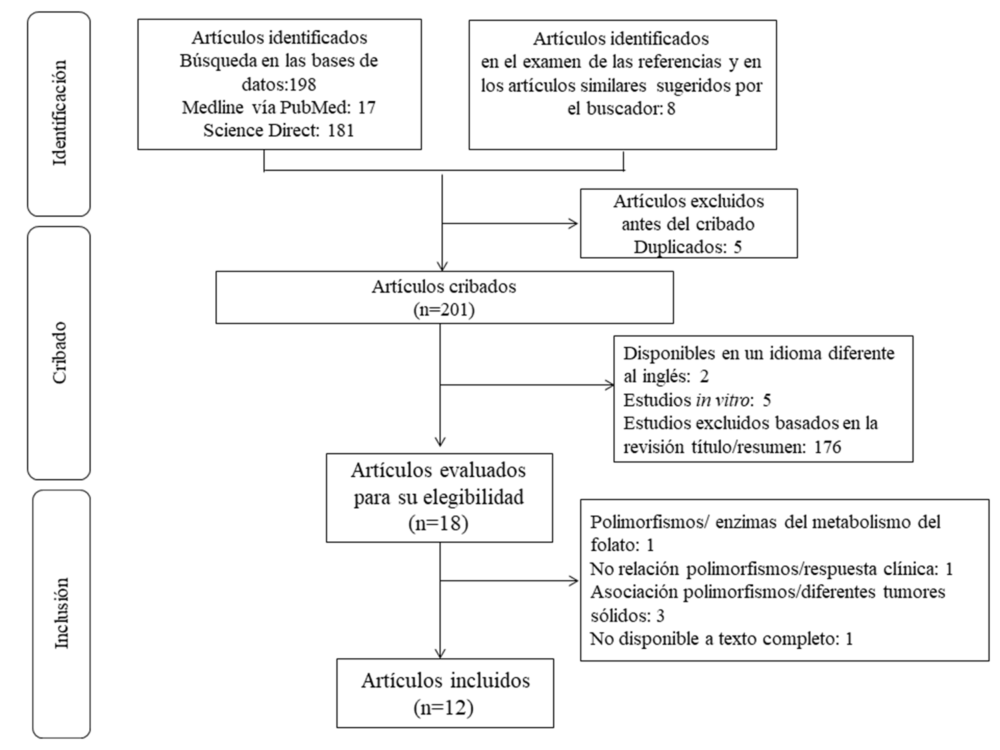
La selección de los estudios se realizó según el diagrama de flujo PRISMA (Figura 1). La estrategia de búsqueda en las bases de datos permitió identificar inicialmente 198 artículos y se identificaron ocho artículos adicionales mediante la lectura de las referencias de las publicaciones recuperadas y los artículos similares sugeridos por el buscador, para un total de 206, de los cuales 12 cumplieron con los criterios para el análisis final.
Figura 1. Diagrama de flujo PRISMA para la selección de los artículos
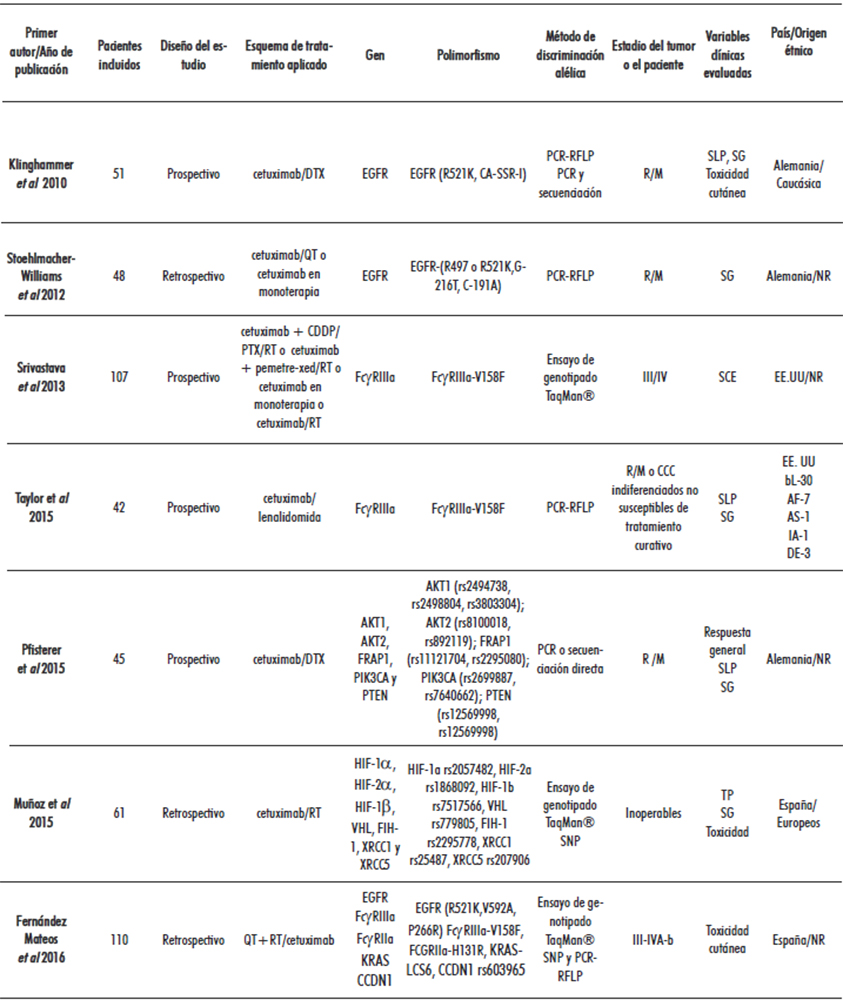
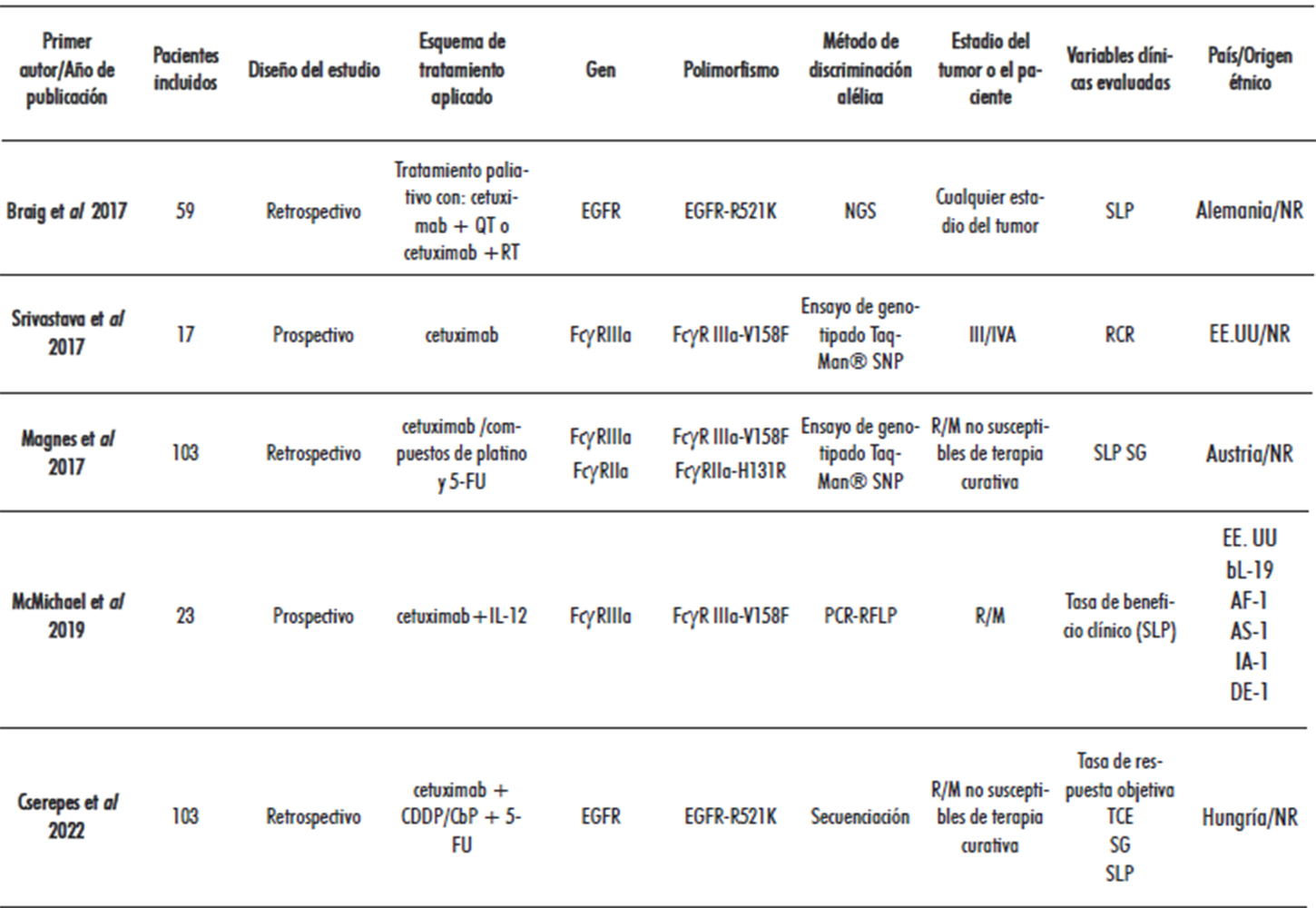
De los 12 trabajos incluidos, ocho fueron estudios de series de casos, de estos, seis presentaban un diseño retrospectivo y dos fueron prospectivos. Los cuatro restantes consistieron en análisis de subgrupos dentro de un EC. Las características de los estudios incluidos se presentan en la Tabla 1.
Tabla 1. Características de los estudios incluidos
Leyenda: DTX: docetaxel; R/M: recurrente y/o metastásico; PCR: reacción en cadena de la polimerasa; RFLP: polimorfismos de longitud de fragmentos de restricción;
CDDP: cisplatino; PTX: paclitaxel; EE. UU: Estados Unidos de América; NR: no referido; SCE: supervivencia por causa específica; NGS: secuenciación de próxima generación;
TP: tiempo hasta la progresión; RCR: respuesta clínica radiológica; IL-12: interleucina-12; CbP: carboplatino; bL: blancos; AF: afroamericanos; AS: asiáticos; IA: indoamericanos;
DE: desconocido
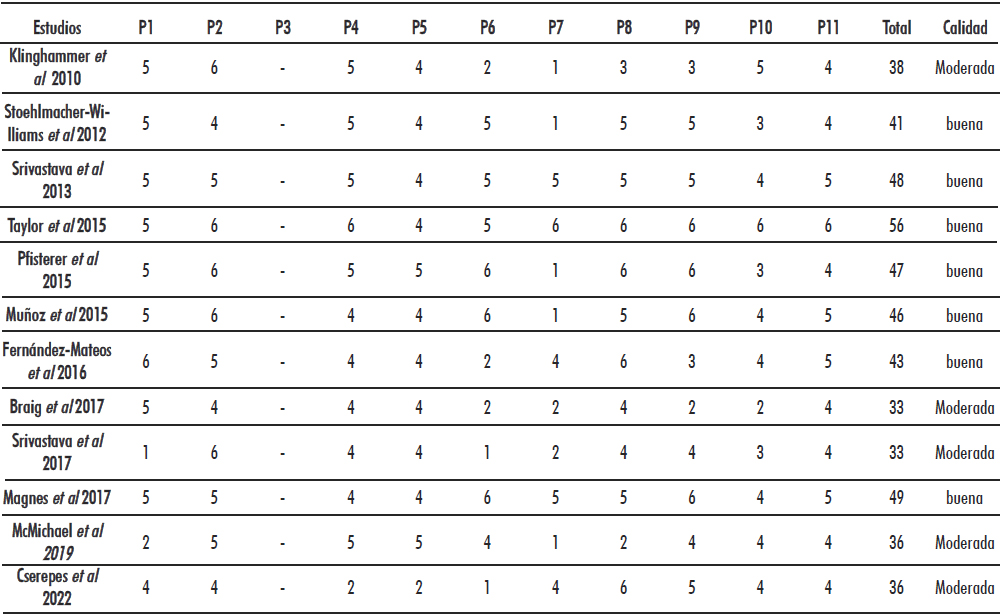
En la evaluación de la calidad se puede constatar que siete artículos son de buena calidad y cinco de calidad moderada (Tabla 2). Ninguno de los estudios tenía grupo control.
El grado de concordancia en la selección de los artículos entre los autores fue muy bueno (κ=0,88).
Tabla 2. Evaluación de la calidad metodológica de los estudios incluidos con la escala Q-Genie
Leyenda: P: pregunta
3.2. Participantes
Se incluyeron un total de 769 pacientes con diagnóstico de CCECC tratados con cetuximab, de estos 415 (54,0%) con carcinoma R/M y el resto de los pacientes se presentaban en: estadio III/IV 234 (30,4%), inoperables 61 (7,9%) o en cualquier estadio del tumor 59 (7,7%). En relación al origen étnico sólo se consideró la etnicidad en cuatro estudios con 177 pacientes: blancos: 161 (91,0%), afroamericanos: ocho (4,5%), asiáticos: dos (1,1%), indoamericanos: dos (1,1%) y desconocidos: cuatro (2,3%).
Polimorfismos del gen EGFR En los estudios incluidos se identificaron seis polimorfismos del gen EGFR que fueron evaluados para su asociación con la respuesta al tratamiento con cetuximab, los cuales son: EGFR- R521K, EGFR-G216T, EGFR-C191A, EGFR-V592A, EGFR-P266R y CA-SSR-1.
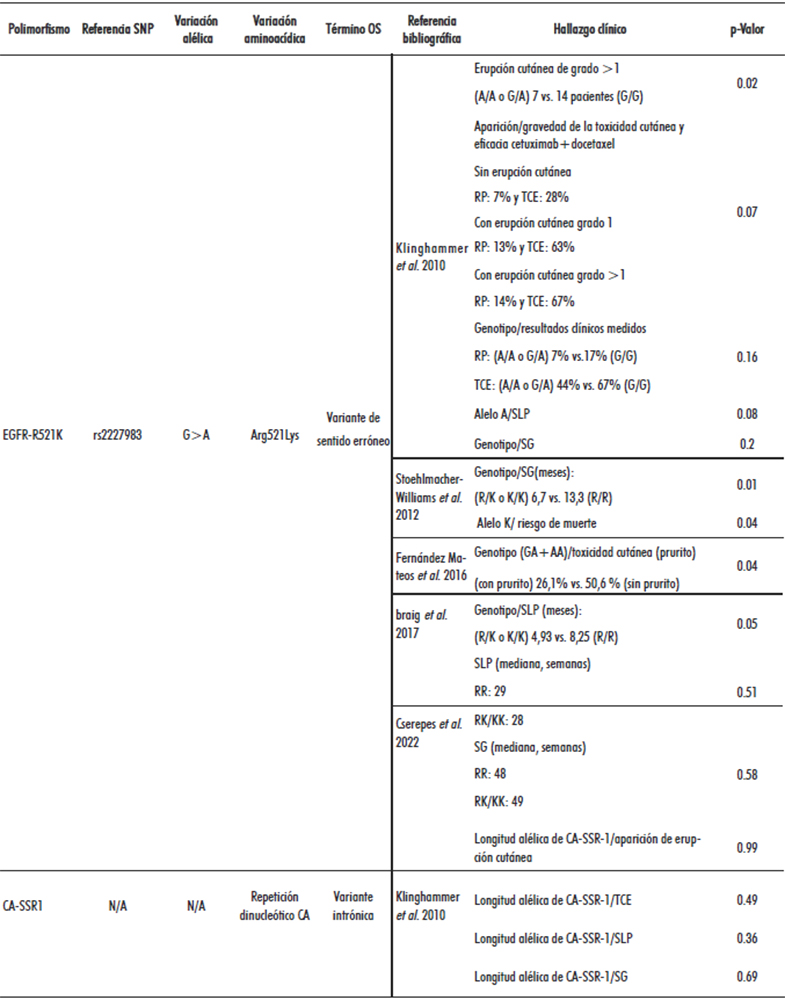
Se encontraron cinco trabajos que evaluaron la influencia del polimorfismo EGFR-R521K en la respuesta del CCECC al cetuximab, de ellos, cuatro [Klinghammer et al., 2010 (27), Stoehlmacher-Williams et al., 2012 (28), Fernández Mateos et al., 2016 (29), braig et al., 2017 (30)] encontraron asociación entre la presencia del polimorfismo y las variables clínicas evaluadas, mientras que en el estudio de Cserepes et al., 2022 (31) no se encontró correlación.
Klinghammer et al., 2010 evaluaron los polimorfismos EGFR-R521K y CA-SSR-1. Se analizó la asociación entre el SNP EGFR-R521K y la toxicidad cutánea, reacción adversa más frecuente del cetuximab cuya presencia es considerada un biomarcador clínico de eficacia al AcM, encontrándose que los pacientes con el genotipo A/A o G/A se asociaron significativamente con una menor incidencia de erupción cutánea de grado >1 en comparación con el genotipo G/G. Además, se constató una tendencia a la asociación entre la aparición/severidad de la toxicidad cutánea y la eficacia del tratamiento (27) (Tabla 3).
También se evaluó la relación entre el SNP EGFR-R521K con los resultados clínicos y se comprobó que los pacientes con el alelo A (A/A o G/A) presentaron una menor respuesta parcial y TCE, así como una tendencia a una SLP más corta. No se observó que el polimorfismo EGFR-R521K influyera en la SG. El polimorfismo CA- SSR-1 no estuvo asociado con la aparición de erupción cutánea, la TCE, SLP o SG (27) (Tabla 3).
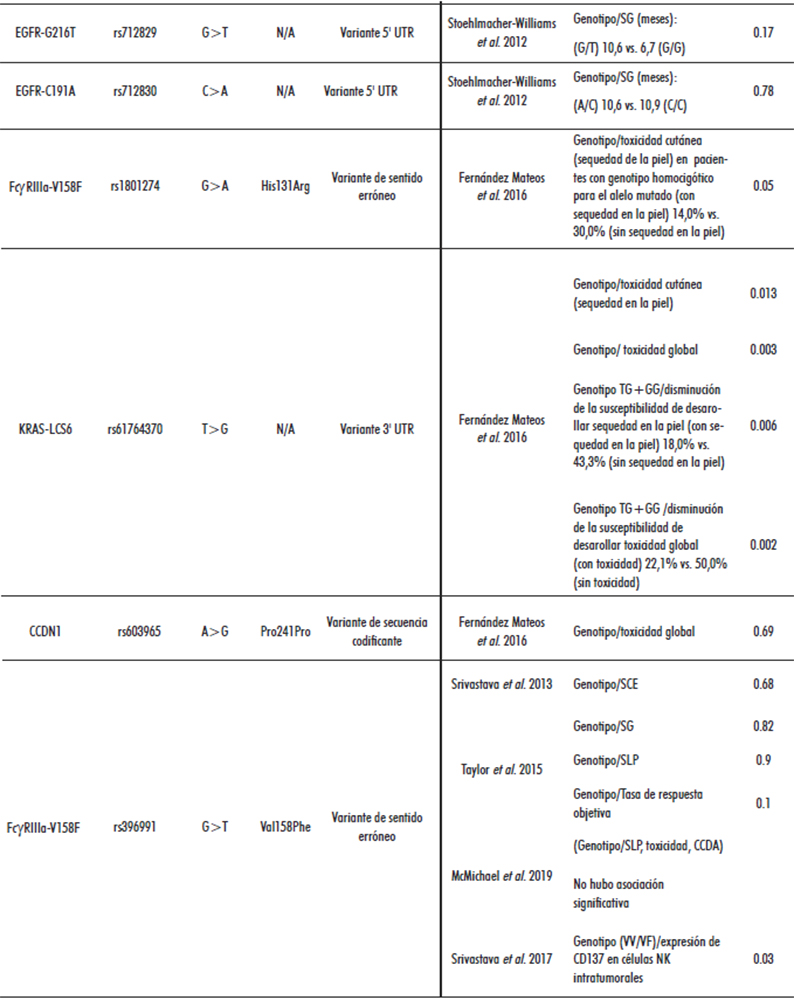
Stoehlmacher-Williams et al., 2012 identificaron los polimorfismos EGFR-R497K (EGFR-R521K), EGFR-G216T y EGFR-C191A. Para el SNP EGFR-R497K (EGFR-R521K) se encontró que los pacientes con al menos un alelo K presentaron una mediana de SG menor en comparación con pacientes homocigóticos para el alelo de tipo silvestre y un riesgo significativamente mayor de muerte independientemente de la edad, el tratamiento y la respuesta. No se observó asociación significativa entre el polimorfismo y la respuesta al tratamiento. En relación a los SNP EGFR-G216T y EGFR-C191A no se halló asociación entre estos polimorfismos y la SG o la respuesta mediante el análisis univariado (28) (Tabla 3).
Fernández Mateos et al., 2016 hallaron asociación entre el SNP EGFR-R521K y la toxicidad cutánea, encontrándose que los portadores del alelo A (GA+ AA) tenían un menor riesgo de sufrir prurito. Los SNP EGFR-V592A y EGFR-P266R no fueron considerados en el análisis porque en la cohorte de pacientes sólo se identificó el genotipo silvestre para ambos polimorfismos (29). En el estudio realizado por Braig et al., 2017 observaron que los pacientes con el polimorfismo EGFR K521 o alelo K (homo y heterocigotos) mostraron una SLP significativamente más corta con respecto a los que no expresaron el alelo K (30). Cserepes et al 2022., compararon la SG y la SLP de los pacientes con los genotipos silvestre y mutado para el SNP EGFR-R521K y no encontraron correlación, concluyendo que el estado del polimorfismo no era predictivo ni pronóstico en la cohorte estudiada (31) (Tabla 3).
Tabla 3. Resultados obtenidos según polimorfismos identificados
Leyenda: N/A: no aplicable; OS: ontología de secuencia génica; RP: respuesta parcial; TCE: tasas de control de la enfermedad; SCE: supervivencia por causa específica; TPE: tiempo hasta la progresión de la enfermedad
3.3. Polimorfismos del receptor del fragmento C gamma (FcγR)
En el trabajo publicado por Srivastava et al., 2013 (32) se encontró que el genotipo FcγRIIIa no se asoció con la supervivencia específica de la enfermedad en la cohorte de pacientes evaluados. Taylor et al., 2015 (33) agruparon los pacientes que eran tanto homocigóticos como heterocigóticos para el alelo V en la posición 158 y se compararon con los que eran homocigóticos para el alelo
F. No se encontró asociación entre el genotipo FcγRIIIa y la SG, SLP o tasa de respuesta objetiva. Fernández Mateos et al., 2016 (29) no constataron asociación entre el SNP FcγRIIIa y la toxicidad cutánea del cetuximab. McMichael et al., 2019 (34) no observaron una asociación significativa entre el estado del polimorfismo FcγRIIIa y el beneficio clínico, evaluado a través de la SLP, la toxicidad o la citotoxicidad celular dependiente de anticuerpos (CCDA) (Tabla 3). Srivastava et al., 2017 (35) constataron una inducción significativa de CD137 [miembro de la familia de receptores del factor de necrosis tumoral y un marcador de activación de las células asesinas naturales (NK) tras la exposición a cetuximab] en las células NK intratumorales, pero no en las circulantes, principalmente en pacientes con FcγRIIIa VV/VF. Por el contrario, no se comprobó un aumento significativo en la expresión de CD137 en células NK intratumorales o de sangre periférica en pacientes con el genotipo FcγRIIIa FF. En el estudio, la expresión de CD137 aumentó significativamente en los pacientes con una buena respuesta clínica radiológica (Tabla 3).
Fernández Mateos et al., 2016 (29) observaron que el genotipo homocigótico para el alelo mutado del SNP FcγRIIa estuvo muy próximo de mostrar asociación con la disminución del riesgo de presentar sequedad en la piel (Tabla 3). Por otro lado, Magnes et al., 2017(36,37) evaluaron la influencia de los polimorfismos FcγRIIIa-V158F y FcγRIIa-H131R en la supervivencia de los pacientes con CCECC recurrente y/o metastásico tratados con cetuximab y obtuvieron que los pacientes con los genotipos FcγRIIIa-158V/V y/o FcγRIIA-131H/H presentaron una mayor SLP y SG en comparación con aquellos con 158 (V/F o F/F) y 131 (H/R o R/R) (Tabla 3).
Los polimorfismos FcγRIIIa-V158F y FCGRIIa-H131R, así como las características del tumor y del paciente fueron evaluados mediante el análisis univariado y multivariado para la SLP. Los genotipos FcγRIIIa-158 V/V, FγRIIA-131H/H y el espacio de tiempo > 12 meses del diagnóstico inicial a la recaída sin posibilidades de terapia curativa, se mantuvieron como los únicos factores pronósticos independientes para la SLP en el análisis de regresión de Cox. También fueron evaluados estos factores para la SG, pero no se observó asociación estadística entre los polimorfismos FcγRIIIa- V158F y FcγRIIA-H131R y la SG (p=0,08) (36,37).
3.4. Variaciones genéticas de la vía PI3K/PTEN/AKT/mTOR
Se identificó un trabajo (Pfisterer et al., 2015) que evaluó algunos polimorfismos de esta vía y la respuesta clínica de los pacientes con CCECC al tratamiento con cetuximab y son los siguientes: AKT1, AKT2, FRAP1, PIK3CA y PTEN (38).
Los investigadores observaron que dos SNP (AKT2:rs8100018 y PTEN: rs12569998) fueron asociados con la variación en el riesgo de progresión. El genotipo homocigótico para el alelo mutado (GG) de AKT2:rs8100018 fue el que presentó un mayor riesgo y en portadores del genotipo TG del PTEN: rs12569998 también se observó un incremento del riesgo. La presencia de ambos polimorfismos calificados como desfavorables (AKT2:rs8100018 y PTEN: rs12569998 variantes) se asociaron con un mayor riesgo de progresión, razón de riesgo de 12,53, mientras que la presencia de uno de los dos a un aumento del riesgo de 4,27 veces en comparación con los pacientes sin ninguna de las dos formas desfavorables (38) (Tabla 3).
Para los SNP PTEN: rs12569998 y AKT2:rs8100018 se observaron diferencias en la mediana de tiempos de SLP. Para AKT2:rs8100018, los pacientes homocigóticos para el alelo silvestre (CC) presentaron una mejor SLP en comparación con aquellos con al menos un alelo mutado. En relación a PTEN:rs12569998, los pacientes homocigóticos con el alelo común (TT) mostraron una mayor SLP con respecto a aquellos con la variante heterocigótica (TG). Los pacientes con los dos genotipos desfavorables mostraron una mediana de SLP más corta en comparación con los portadores de un genotipo desfavorables o aquellos sin la presencia de estos. No se apreciaron diferencias significativas en la mediana de tiempos de SLP para los otros SNP analizados (38) (Tabla 3).
Con respecto a la asociación entre las variantes genotípicas estudiadas y la SG, encontraron que dos de los SNP evaluados estaban significativamente asociados con la supervivencia: AKT1: rs3803304 y AKT2:rs8100018. El genotipo homocigótico para el alelo mutado de AKT2:rs8100018 fue significativamente asociado con un mayor riesgo de muerte y con una mediana de SG más corta. Los pacientes con al menos un alelo mutado de AKT1:rs3803304 mostraron un menor riesgo de muerte y una significativamente mayor SG. No se observaron diferencias significativas en la mediana de tiempos de SG en ninguno de los otros SNP analizados (38) (Tabla 3).
3.5. Variaciones alélicas en los genes relacionados con la hipoxia y la reparación del ADN
Muñoz et al., 2015 (39) seleccionaron siete SNP para el análisis: cinco relacionados con los genes de la hipoxia (HIF-2α rs1868092; HIF-1α rs2057482; HIF-1β rs7517566; VHL rs779805;
FIH-1 rs2295778) y dos en los genes de reparación del ADN (XRCC1 rs25487; XRCC5 rs207906).
Los pacientes con el genotipo CT/TT del SNP HIF-1α presentaron una mayor SG que aquellos con el genotipo homocigótico (CC) para el alelo silvestre, la cual fue todavía más prolongada en los pacientes que presentaban mucositis o foliculitis en relación con los que no tenían toxicidad cutánea (39) (Tabla 3). Para el gen XRCC5, los pacientes homocigóticos para el alelo de tipo silvestre (GG) o mutado (AA) tuvieron un tiempo más prolongado hasta la progresión de la enfermedad (TPE) que los pacientes con el genotipo heterocigótico GA. Tanto el TPE como la SG fueron mayores en pacientes homocigóticos GG/AA con mucositis o foliculitis severa en comparación con aquellos sin toxicidad en la piel (39) (Tabla 3).
3.6. Polimorfismos: KRAS-LCS6 y CCDN1
Fernández Mateos et al., 2016 (29) evaluaron la influencia del SNP KRAS-LCS6 (rs61764370) en la toxicidad cutánea y encontraron asociación significativa entre este polimorfismo con la sequedad en la piel. Además, se observó que los pacientes portadores del alelo mutado G (genotipos TG/GG) tenían una menor susceptibilidad a desarrollar sequedad en la piel y toxicidad global después del tratamiento con cetuximab. Para el SNP CCDN1 rs603965 no se halló asociación entre el polimorfismo y la toxicidad cutánea producida por el cetuximab (Tabla 3).
4. DISCUSIÓN
La presente revisión bibliográfica expone por primera vez la evidencia científica sobre los principales polimorfismos genéticos que han sido asociados a la respuesta del CCECC al cetuximab. Se encontraron SNP que pueden ser usados como biomarcadores apropiados para seleccionar los pacientes con mayor probabilidad de beneficiarse con este antagonista del EGFR.
En relación al gen EGFR se han descrito una multitud de polimorfismos, algunos de estos son funcionales y se asocian con la regulación del EGFR. En la búsqueda realizada se identificaron cinco SNP y un polimorfismo de secuencia repetida.
El SNP EGFR-R521K, es uno de polimorfismos del EGFR mejor investigados y más abordados en la literatura. El EGFR-K521 o alelo K, se expresa en más del 40% de los casos de CCECC y se ha reportado que su presencia produce una variante del EGFR que muestra una función disminuida, al presentar una menor afinidad por sus dos principales ligandos endógenos (factor de c recimiento epidémico y el factor α transformador del crecimiento), así como una disminución en su actividad mitogénica. Los mecanismos moleculares que subyacen a estos resultados no están claramente definidos, pero se ha planteado que el intercambio de aminoácidos produce una reducción en la afinidad y estabilidad del c etuximab en el EGFR K521 (28,30, 40).
En contraste con estos planteamientos, en un estudio realizado por Yu Hsieh et al ., 2012 (41) donde se analizó el efecto de este polimorfismo en 112 pacientes con carcinoma colorrectal metastásico (CCRm) con gen KRAS de tipo salvaje (KRASwt ) tratados con cetuximab como tratamiento de primera línea más el esquema FOLFOX-4 (5-FU, leucovorina y oxaliplatino), se encontró que los pacientes con el alelo K presentaron una marcada disminución en los niveles de expresión del factor de crecimiento endotelial vascular (VEGF) (66,7 % versus 28,9 %, p < 0,01) que se asociaron con una disminución del tamaño del tumor (55,6% versus 31,6%, p= 0,02), buena diferenciación histológica (63,9% versus 85,5%,p=0,01), invasión linfovascular disminuida (69,4% versus 39,5%, p < 0,01) y una mayor tasa de respuesta al tratamiento (55,6 % versus 78,9 %, p= 0,01). Además, este polimorfismo se asoció con un mayor período libre de progresión (p= 0,001) y una mayor SG (p= 0,001). Zhang et al., 2005 (42) encontraron que los pacientes con CCR portadores del genotipo G/G de EGFR-R521K tenían tendencia a un mayor riesgo de recurrencia tumoral local que los pacientes con el genotipo G/A o A/A (p=0,24). Gonçalves et al., 2008 (43), observaron que 11 de 12 pacientes con CCR que presentaron el alelo K alcanzaron respuesta objetiva o enfermedad estable y que tanto la SLP como la SG fueron significativamente mayores en estos pacientes en relación con aquellos homocigóticos para el alelo silvestre (p = 0,041) (p= 0,03), respectivamente. Maeda et al., 2019 (44) hallaron que los pacientes con este mismo tipo de cáncer y homocigóticos para el alelo mutado (KK) presentaron una mayor reducción del tamaño del tumor (p= 0,04). Hasta la fecha no hay una explicación clara para estos resultados contradictorios en ambos tipos de tumores, aunque se plantea que pudieran deberse a las diferentes rutas en la cascada de señalización del EGFR y los diferentes grados de afinidad requeridos por el cetuximab para la inhibición de la señal (44). El polimorfismo CA-SSR-1 implica la repetición del dinucleótido CA (citosina/adenina) en el intrón 1 del EGFR. Se considera que un alelo es corto cuando las repeticiones CA son menores o iguales a 16 y un alelo es largo cuando son mayores a 17 repeticiones CA. La longitud de los alelos de este polimorfismo se correlacionan inversamente con la actividad transcripcional del gen EGFR (45, 46, 47). Graciano et al., 2008 (48) evaluaron el polimorfismo CA-SSR-1 conjuntamente con el SNP 61 A>G del EGF en una cohorte de 110 pacientes con CCRm cuyo estado de la mutación de KRAS era desconocido, tratados con irinotecán más cetuximab y encontraron que los pacientes con dos alelos con menos de 17 repeticiones y un genotipo 61GG presentaron una mayor SLP y SG, en cambio, Loupakis et al., 2014 (49) estudiaron 115 pacientes con CCRm y no hallaron diferencias significativas tanto en la SLP (p= 0,991) como en la SG (p = 0,261) en los portadores de alelos cortos o largos.
Referente a los SNP EGFR-G216T y EGFR-C191A son dos polimorfismos en la región promotora que afectan el sitio de unión de un factor de transcripción Sp1 (50). Dahan et al., 2011 (51) evaluaron el polimorfismo EGFR-C191A en pacientes con CCRm con KRASwt, tratados con irinotecán más cetuximab y observaron asociación entre el SNP-191C > A y el TPE, encontrándose que los pacientes homocigóticos (CC) EGFR-191 presentaron un TPE más largo (p = 0,030).
En relación a los polimorfismos del FcγR, cuando se produce la interacción FcR-AcM, se activan muchas funciones, entre ellas, la CCDA, mecanismo que contribuye al efecto antitumoral de los AcM anti-EGFR. Funcionalmente, estos SNP conducen a variabilidad en la afinidad de la unión a la IgG humana, lo cual podría influir en el potencial citotóxico de las células efectoras del sistema inmune. En función de las diferentes afinidades, se espera que los pacientes con los genotipos FcγRIIa-131H/H y FcγRIIIa-4 158V/V medien una CCDA más eficaz (52).
En este sentido dos estudios (35, 36, 37) muestran una asociación significativa entre las variables clínicas evaluadas y la presencia del polimorfismo FcγRIIIa, mientras que, en otros cuatro (29,32, 33,34) no se encontró que este SNP influyera en la variabilidad de la respuesta mostrada por los pacientes. Para el SNP FcγRIIa-H131R, en un estudio (36,37) se observó asociación entre este polimorfismo y el resultado clínico en los pacientes tratados y en otro trabajo no se constató correlación, pero hubo proximidad de existir asociación entre ambas variables (29).
Zhang et al., 2007 (53) observaron que los polimorfismos FcγRIIa-H131R y FcγRIIIa-V158F se asociaron con una mayor SLP en 39 pacientes con CCRm (p=0,004) mientras que Inoue et al., 2014 (54) evaluaron 57 pacientes también con CCRm y constataron que los portadores de los alelos 131H y 158V presentaron tasas de respuesta inferiores en comparación con aquellos homocigóticos para los alelos mutados (p=0,018). Calemma et al., 2012 (55) encontraron que los polimorfismos del FcγRIIIa se asociaron significativamente con la respuesta a la terapia basada en AcM anti- EGFR en 49 pacientes con CCRm con KRASwt (p=0,035).
Shepshelovich et al., 2018 (56) evaluaron 592 pacientes con CCRm con KRASwt y constataron que aquellos con el genotipo FcγRIIa H/H mostraron una mejor SG (HR: 0,66, p<0,001; mediana del beneficio absoluto, 1,3 meses) en comparación con los que presentaron el alelo R. Liu et al., 2016 (57) realizaron un estudio en pacientes con cáncer en la misma localización tratados con cetuximab en monoterapia y encontraron que los portadores del genotipo FcγRIIa H/H mejoraron sustancialmente tanto la SLP (p= 0,02) como la SG (p = 0,003). Tamura et al., 2011 (58) reportaron que en pacientes con cáncer de mama HER2 (Receptor- 2 del factor de crecimiento epidérmico humano) positivos tratados con trastuzumab, el polimorfismo FcγRIIa-H131H se asoció con una SLP más larga (p=0,034).
Por otro lado, en un análisis retrospectivo publicado por Páez et al., 2010 (59) se concluyó que, el polimorfismo FcγR no era útil para predecir la respuesta al cetuximab o al panitumumab en pacientes con CCRm. El estudio incluyó a 104 pacientes que fueron tratados con cetuximab o panitumumab después de progresar y que recibieron tratamiento previo con al menos un régimen de QT, no observando diferencias significativas en la respuesta tumoral y la SLP entre los subconjuntos con genotipo FcγRIIa y FcγRIIIa.
La vía PI3K/PTEN/AKT/mTOR es una de las vías de señalización celular más importantes, que desempeña un papel crucial en la regulación del crecimiento celular, diferenciación, migración, proliferación, angiogénesis, síntesis de proteínas y metabolismo de la glucosa. Los estudios han señalado que la desregulación de los principales componentes de esta vía de señalización conduce a una activación aberrante de las vías posteriores, promoviendo finalmente la aparición del cáncer. Varias publicaciones reportan que las variaciones genéticas en esta vía están asociadas con los resultados clínicos, propiedades de invasión, resistencia a la QT y complicaciones del tratamiento en diferentes tipos de cáncer incluido el CCECC (60,61, 62).
Los polimorfismos rs17849079 y rs7640662 frecuentes en el gen PIK3CA (que codifica p110α, la subunidad principal de PI3K) ocurren en el 10%-18% de los pacientes con CCR y se conocen como “mutaciones de punto crítico”. Un metaanálisis confirmó que el alelo T rs17849079 y el alelo C rs7640662 predicen una tasa de respuesta objetiva, una SG y una SLP más bajas en pacientes con CCRm tratados con cetuximab y panitumumab (22).
Diferentes autores han demostrado que los SNP de PTEN rs701848 alelo C, rs2735343 alelo G y rs11202586 alelo T se correlacionan con un mayor riesgo de cáncer de células escamosas de esófago y tumores de células germinales testiculares, sin embargo, hasta la fecha no se ha demostrado asociación con la respuesta al tratamiento con AcM anti-EGFR (22).
El microambiente hipóxico es una característica común en los tumores sólidos, incluidos los CCECC (63). Diversas investigaciones revelan que la hipoxia tumoral puede: activar la angiogénesis, lo que aumenta la invasividad, el riesgo de metástasis, la supervivencia del tumor, así como suprime la inmunidad antitumoral y afecta la respuesta terapéutica (64). El cambio molecular más estudiado en la hipoxia tumoral es el aumento en la expresión de los factores inducibles por hipoxia (HIF) que pueden regular la transcripción de una multitud de proteínas críticas que controlan la proliferación, la señalización angiogénica, la metástasis y otros factores oncogénicos (65,66). Los HIF son los marcadores mejor caracterizados que median las respuestas celulares al estrés hipóxico. De los tres miembros de la familia HIF, HIF-1 y HIF-2 son los que mejor se han caracterizado (67).
Qin et al., 2012 (68) constataron que los polimorfismos HIF-1α rs2057482 y VHL rs779805 influyen en la progresión y la supervivencia de pacientes con cáncer de células renales, al observar que, de cada polimorfismo, los pacientes portadores del genotipo con el alelo mutado presentaron una mayor supervivencia a los cinco años en comparación con aquellos que presentan el alelo salvaje (p= 0,045). Guo et al., 2015 (69) evaluaron el SNP rs2057482 en el gen HIF1A en pacientes con carcinoma hepatocelular agresivo y apreciaron que los pacientes con variaciones alélicas (CT/TT) del SNP rs2057482 tenían un riesgo de recurrencia significativamente menor en comparación con los portadores del genotipo CC (p= 0,028), además la mediana de supervivencia libre de recurrencia fue de 14,2 meses en pacientes con el genotipo CC y de 16,9 meses en pacientes con genotipos CT/TT.
Además del gen HIF, otros genes también participan en el proceso de hipoxia y probablemente contribuyen a la supervivencia de los pacientes con cáncer, como los genes implicados en los mecanismos de reparación del ADN. El gen que codifica a la proteína 1 de reparación por escisión del grupo de complementación cruzada de las lesiones por rayos X (XRCC1) es uno de los más estudiados y se ha investigado por su posible asociación con el pronóstico del cáncer. El SNP rs25487 [Arg399Gln, sustitución G>A en la posición 28152, exón 10] se encuentra entre los SNP XRCC1 más investigados, sin embargo, aún no se ha esclarecido su asociación con la respuesta (70).
Nanda et al ., 2018 (71) evaluaron los genotipos del polimorfismo XRCC1 en 101 pacientes con CCECC tratados con quimioradioterapia concurrente y hallaron que los pacientes con variantes polimórficas presentaron tasas más altas de mucositis oral de grado > 2 (p= 0,023), dermatitis (p= 0,003) y toxicidad laríngea (p = 0,006), así como mayores tasas de respuesta completa (p= 0,209), SLP (p=0,077) y SG (p=0,143), en comparación con la variante salvaje.
El gen XRCC5 se encuentra en el cromosoma 2 y codifica la proteína Ku80, una helicasa de ADN dependiente del adenosín trifosfato (ATP) involucrada en la reparación de roturas de doble cadena del ADN, además actúa como un factor de adherencia que participa en la adherencia celular, la migración, así como la invasión de tumores. También se ha demostrado que XRCC5 se sobreexpresa en varios tejidos tumorales, lo que implica q ue XRCC5 es un factor promotor de tumores. Los polimorfismos en XRCC5 se han correlacionado con la susceptibilidad de desarrollar diversas neoplasias malignas. Los polimorfismos XRCC5 rs16855458 y rs92885 provocan cambios en los sitios de unión de los f actores de transcripción y se asocian con el riesgo de desarrollar carcinoma hepatocelular. No se encontraron estudios que e valuaran la asociación de los polimorfismos en el gen XRCC5 y l as terapias empleadas en el tratamiento del cáncer (72).
La familia de miARN let-7 regula múltiples oncogenes, incluido el gen KRAS. La presencia del polimorfismo (rs61764370) en la región LCS-6, sitio de unión del miARN l et-7 en la región 3- UTR del gen KRAS para regular su expresión, conduce a una activación anormal de este, lo que puede inducir la proliferación en algunos tumores sólidos, incluido el CCECC y conferir resistencia a los medicamentos (73,74). Christensen et al., 2009 constataron que este SNP estaba asociado con una menor SG en pacientes con cáncer oral (75). Sebio et al., 2013 (76) evaluaron la asociación del polimorfismo KRAS let-7 LCS6 con la respuesta al tratamiento con AcM anti-EGFR en 100 pacientes con CCRm refractario, encontrándose que los pacientes con el a lelo G no respondieron a estas terapias (p=0,004).
En contraste con estos resultados, Zhang et al., 2011(74) estudiaron 130 pacientes con CCRm inscritos en un EC fase II y tratados con cetuximab en monoterapia y observaron que los pacientes con el genotipo silvestre TT presentaron una menor tasa de respuesta objetiva en comparación con aquellos con el alelo mutado TG o GG (9% vs. 42%) (p=0,02, prueba exacta de Fisher), así como una tendencia a una peor supervivencia, evaluada a través de la SLP y SG (1,3 vs. 3,9 meses) (6,4 vs. 10,7 meses), respectivamente. Saridaki et al., 2014 (77) hallaron que los pacientes con CCRm portadores del genotipo (TG/GG) tuvieron una SLP significativamente mayor durante tratamiento con AcM anti- EGFR en monoterapia (16,85 vs. 7,85 semanas, p= 0,019).
De Ruyck et al., 2014 (78) encontraron que el alelo G se asoció significativamente con una mejor SG (HR = 0,40; p=0,031) en pacientes con cáncer de orofaringe, mientras que en el trabajo publicado por Ulusan et al., 2022 (73) se observó que la variante homocigótica del alelo G estaba significativamente asociada con metástasis en los ganglios linfáticos en pacientes con CCECC. En un metaanálisis publicado por Ying et al., 2014 (79) no se halló una asociación significativa entre el genotipo GT/GG y la respuesta a la terapia con AcM anti-EGFR en pacientes con CCRm (PH = 0,003, OR = 1,18, IC del 95 % CI= 0,34–4,71)
Esta revisión bibliográfica presenta algunas limitaciones. Primero, en la búsqueda sólo se incluyeron los artículos en inglés, lo que puede dar lugar a un sesgo de publicación. Segundo, el número de estudios incluidos para la variedad de polimorfismos identificados es bajo y en algunos casos sólo estuvo disponible una sola publicación debido a la falta de datos publicados o el empleo de dos fuentes de publicación. Tercero, en algunos estudios el tamaño de la muestra es pequeño, lo que limita el poder estadístico de las asociaciones analizadas y además se presentan diferencias en las metodologías utilizadas, pues no todos los artículos son de buena calidad. Cuarto, existe heterogeneidad en los tratamientos indicados y en el origen étnico de los pacientes analizados, aspecto que puede incidir en la variabilidad de los resultados obtenidos. Quinto, existen diferencias en cuanto a la inclusión o no de pacientes con terapias antineoplásicas previas en cada uno de estos estudios. Sexto, las variables para evaluar la respuesta clínica al tratamiento son diferentes. Séptimo, ninguno de los trabajos incluidos tuvo grupo control, lo que limita la robustez metodológica del estudio. Octavo, debido a la falta de homogeneidad de los datos reportados no es posible realizar un análisis estadístico que permita correlacionar la influencia de los polimorfismos analizados en la respuesta a esta terapia.
Esta revisión bibliográfica muestra que de los polimorfismos en el gen EGFR, solamente el SNP EGFR-R521K (AA/GA) se asoció con una reducción de la SG y la SLP, así como con una menor incidencia de toxicidad cutánea, aunque Cserepes et al., 2022 (31) reportaron que no existe correlación entre las variables de supervivencia y la presencia del alelo mutado.
En la evaluación de los polimorfismos del FcγR para el SNP FcγRIIIa-V158F los resultados publicados son contradictorios, mientras que algunos autores refieren que la presencia del alelo V se relacionó con una mayor supervivencia y una mejor respuesta clínica radiológica (35,36), otros no encontraron asociación entre el estado del polimorfismo FcγRIIIa y el beneficio clínico en los pacientes tratados (29, 32, 33,34). Para el SNP FCGRIIa-H131R, los pacientes con el genotipo 131H/H presentaron una mayor SLP y SG, además aquellos con el genotipo homocigótico para el alelo variante presentaron una tendencia a un menor riesgo de sufrir toxicidad en la piel. En relación al SNP KRAS-LCS6, los pacientes portadores del genotipo TG/GG tuvieron una menor susceptibilidad a desarrollar sequedad en la piel y toxicidad global después del tratamiento con cetuximab, lo cual puede relacionarse con una peor respuesta y mayor progresión tumoral.
De los polimorfismos de la vía PI3K/PTEN/AKT/mTOR, se identificaron que AKT2:rs8100018 y PTEN: rs12569998 en sus variantes mutadas, se vincularon con un aumento en el riesgo de progresión y una reducción de la SLP, también la variante homocigótica AKT2:rs8100018 fue asociada significativamente con una mediana de SG más corta mientras que la presencia de al menos un alelo mutado de AKT1:rs3803304 se correlacionó con una SG más larga. De las variaciones alélicas en los genes relacionados con la hipoxia y la reparación del ADN, los polimorfismos HIF-1α (CT/TT) y XRCC5 (GG/AA) se correlacionaron con mayores SG y TPE, respectivamente.
5. CONCLUSIONES
Se han investigado varios polimorfismos que pueden asociarse con la respuesta del carcinoma de cabeza y cuello al tratamiento con cetuximab, siendo EGFR-R521K y FcγR IIIa-V158F los más estudiados. La enorme incertidumbre de los resultados alcanzados no permite alcanzar conclusiones firmes sobre la influencia de los polimorfismos genéticos en la respuesta al cetuximab; sin embargo, pueden convertirse en biomarcadores farmacogenéticos en la práctica clínica como una valiosa herramienta en la medicina personalizada, para predecir la respuesta medicamentosa. Para ello se requiere la realización de ensayos controlados con estratos por genotipo, con asignación aleatoria del tratamiento y el análisis de otras variables con valor pronóstico conocido.
Agradecimientos
A la Lic. Rut Benito Yero por su colaboración en la búsqueda bibliográfica.
6. REFERENCIAS
1. Yan F, Knochelmann HM, Morgan PF, Kaczmar JM, Neskey DM, Graboyes EM, et al. The Evolution of Care of Cancers of the Head and Neck Region: State of the Science in 2020. Cancers (Basel).2020; 12(6):1543. doi: 10.3390/cancers12061543
2. Semidey Raven ME. Evaluación de factores moleculares asociados a la respuesta a cetuximab: integración con tratamiento de radioterapia y quimioterapia en carcinoma escamoso de cabeza y cuello [tesis doctoral]. Barcelona: Departamento de Cirugía y Ciencias Morfológicas, Universitat Autònoma de Barcelona; 2016.
3. Robles Santos J, Pérez García T, Iznaga Marín N, Contreras PJ. Caracterización clínica del cáncer de cabeza y cuello. Rev Cubana de Otorrinolaringol Cirug Cabeza y Cuello. [Internet]. 2020 [citado 24 Oct 2023]; 4 (1) Disponible en: https://revotorrino.sld.cu/index.php/otl/article/view/128
4. Johnson DE, Burtness B, Leemans CR, Lui VWY, Bauman JE, Grandis JR. Head and neck squamous cell carcinoma. Nat Rev Dis Primers. 2020;6(1):92. doi: 10.1038/s41572-020-00224-3
5. Bhat GR, Hyole RG, Li J. Head and neck cancer: Current challenges and future perspectives. Adv Cancer Res. 2021; 152:67-102. doi: 10.1016/bs.acr.2021.05.002
6. Programa Integral para el Control del Cáncer en Cuba. Manual de Diagnóstico y Tratamiento del Cáncer de Cabeza y Cuello. La Habana, Cuba: Ministerio de Salud Pública de Cuba (MINSAP); 2018.
7. Centro de Inmunología Molecular. Protocolo de Ensayo Clínic fase II, aleatorizado, de combinación de poliquimioterapia con platino/5- fluorouracilo y nimotuzumab en Carcinoma de Células Escamosa de Cabeza y Cuello recurrente y/o metastásico; 2010.
8. Singh P, Bennett B, Bailey T, Taylor-Stokes G, Rajkovic I, Contente M. Real-world study of the impact of recurrent/metastatic squamous cell carcinoma of the head and neck (R/M SCCHN) on quality of life and productivity in Europe. BMC Cancer 21. 2021; 854. doi: 10.1186/s12885-021-08557-2.
9. Taberna M, Oliva M, Mesía R. Cetuximab-Containing Combinations in Locally Advanced and Recurrent or Metastatic Head and Neck Squamous Cell Carcinoma. Front Oncol. 2019; 9:383. doi: 10.3389/fonc.2019.00383.
10. Borcoman E, Marret G, Le Tourneau C. Paradigm Change in First-Line Treatment of Recurrent and/or Metastatic Head and Neck Squamous Cell Carcinoma. Cancers (Basel).2021; 13(11):2573. doi: 10.3390/cancers13112573.
11. Rehmani HS, Issaeva N. EGFR in head and neck squamous cell carcinoma: exploring possibilities of novel drug combinations. Ann Transl Med. 2020;8(13):813. doi: 10.21037/atm.2020.04.07.
12. Muraro E, Fanetti G, Lupato V, Giacomarra V, Steffan A, Gobitti C, et al. Cetuximab in locally advanced head and neck squamous cell carcinoma: Biological mechanisms involved in efficacy, toxicity and resistance. Crit Rev Oncol Hematol. 2021; 164:103424. doi: 10.1016/j.critrevonc.2021.103424.
13. Moradi-Kalbolandi S, Hosseinzade A, Salehi M, Merikhian P, Farahmand L. Monoclonal antibody-based therapeutics, targeting the epidermal growth factor receptor family: from herceptin to Pan HER. J Pharm Pharmacol. 2018; 70(7):841-854. doi: 10.1111/jphp.12911.
14. Yang CC, Lien CF, Hwang TZ, Wang CC, Wang CC, Shih YC, et al. Treatment Sequences in Patients with Recurrent or Metastatic Head and Neck Squamous Cell Carcinoma: Cetuximab Followed by Immunotherapy or Vice Versa. Cancers (Basel). 2022; 14(10):2351. doi: 10.3390/cancers14102351.
15. Chapman C H, Saba NF, Yom SS. Targeting epidermal growth factor receptor for head and neck squamous cell carcinoma: still lost in translation? Ann Transl Med. 2016; 4(4):80. doi: 10.3978/j.issn.2305-5839.2016.01.01.
16 Ferris RL, Moskovitz J, Kunning S, Ruffin AT, Reeder C, Ohr J, et al. Phase I Trial of Cetuximab, Radiotherapy, and Ipilimumab in Locally Advanced Head and Neck Cancer. Clin Cancer Res. 2022;28(7):1335-1344. doi: 10.1158/1078-0432.CCR-21-0426.
17. Guigay J, Tahara M, Licitra L, Keilholz U, Friesland S, Witzler P, et al. The Evolving Role of Taxanes in Combination with Cetuximab for the Treatment of Recurrent and/or Metastatic Squamous Cell Carcinoma of the Head and Neck: Evidence, Advantages, and Future Directions. Front Oncol. 2019; 9:668. doi: 10.3389/fonc.2019.00668.
18. Leblanc O, Vacher S, Lecerf C, Jeannot E, Klijanienko J, Berger F. Biomarkers of cetuximab resistance in patients with head and neck squamous cell carcinoma. Cancer Biol Med. 2020; 17 (1): 208-217. doi: 10.20892/j.issn.2095-3941.2019.0153.
19. Picon H, Guddati A. Mechanisms of resistance in head and neck cancer. Am J Cancer Res. 2020;10(9):2742-2751.
20. Novoplansky O, Fury M, Prasad M, Yegodayev K, Zorea J, Cohen L, et al. MET activation confers resistance to cetuximab, and prevents HER2 and HER3 upregulation in head and neck cancer. Int J Cancer. 2019;145(3):748-762. doi: 10.1002/ijc.32170.
21. Ortiz-Cuaran S, Bouaoud J, Karabajakian A, Fayette J, Saintigny P. Precision Medicine Approaches to Overcome Resistance to Therapy in Head and Neck Cancers. Front Oncol. 2021;11:614332. doi: 10.3389/fonc.2021.614332.
22. Shek D, Read SA, Ahlenstiel G, Piatkov I. Pharmacogenetics of anticancer monoclonal antibodies. Cancer Drug Resist. 2019; 2(1):69-8. doi: 10.20517/cdr.2018.20.
23. Reyes Reyes E, Rodeiro Guerra I, Alfredo Herrera J, Cuétara Lugo EB. Polimorfismos en genes de reparación del daño al material genético y cáncer de pulmón. Rev Cub Med Mil. [Internet]. 2021 [citado 29 Oct 2023]; 51 (1) Disponible en: https://revmedmilitar.sld.cu/index.php/mil/article/view/1467
24. Mellor JD, Brown MP, Irving HR, Zalcberg JR, Dobrovic A. A critical review of the role of Fc gamma receptor polymorphisms in the response to monoclonal antibodies in cancer. J Hematol Oncol. 2013; 6:1. doi: 10.1186/1756-8722-6-1.
25. Sohani ZN, Meyre D, de Souza RJ, Joseph PG, Gandhi M, Dennis BB, et al. Assessing the quality of published genetic association studies in meta-analyses: the quality of genetic studies (Q-Genie) tool. BMC Genet. 2015; 16:50. doi: 10.1186/s12863-015-0211-2.
26. Hoxhaj I, Vukovic V, Boccia S, Pastorino R. Single nucleotide polymorphisms and the risk of developing a second primary cancer among head and neck cancer patients: a systematic literature review and meta-analysis. BMC Cancer. 2021; 21(1):660. doi: 10.1186/s12885-021-08335-0.
27. Klinghammer K, Knödler M, Schmittel A, Budach V, Keilholz U, Tinhofer I. Association of epidermal growth factor receptor polymorphism, skin toxicity, and outcome in patients with squamous cell carcinoma of the head and neck receiving cetuximab-docetaxel treatment. Clin Cancer Res. 2010;16(1):304-10. doi: 10.1158/1078-0432.CCR-09-1928.
28. Stoehlmacher-Williams J, Obermann L, Ehninger G, Goekkurt E. Polymorphisms of the epidermal growth factor receptor (EGFR) and survival in patients with advanced cancer of the head and neck (HNSCC). Anticancer Res. 2012;32(2):421-5.
29. Fernández-Mateos J, Seijas-Tamayo R, Mesía R, Taberna M, Pastor Borgoñón M, Pérez-Ruiz E, et al. Epidermal growth factor receptor (EGFR) pathway polymorphisms as predictive markers of cetuximab toxicity in locally advanced head and neck squamous cell carcinoma (HNSCC) in a Spanish population. Oral Oncol. 2016;63:38-43. doi: 10.1016/j.oraloncology.2016.10.006.
30. Braig F, Kriegs M, Voigtlaender M, Habel B, Grob T, Biskup K, et al. Cetuximab Resistance in Head and Neck Cancer is mediated by EGFR- K521 Polymorphism. Cancer Res. 2017; 77(5): 1188-1199. doi: 10.1158/0008-5472.CAN-16-0754.
31. Cserepes M, Nelhűbel GA, Meilinger-Dobra M, Herczeg A, Türk D, Hegedűs Z, et al. EGFR R521K Polymorphism Is Not a Major Determinant of Clinical Cetuximab Resistance in Head and Neck Cancer. Cancers (Basel). 2022;14(10):2407. doi: 10.3390/cancers14102407.
32. Srivastava RM, Lee SC, Andrade Filho PA, Lord CA, Jie HB, Davidson HC, et al. Cetuximab-activated natural killer (NK) and dendritic cells (DC) collaborate to trigger tumor antigen-specific T cell immunity in head and neck cancer patients. Clin Cancer Res. 2013; 19(7): 1858– 1872. doi: 10.1158/1078-0432.CCR-12-2426.
33. Taylor RJ, Saloura V, Jain A, Goloubeva O, Wong S, Kronsberg S, et al. Ex vivo antibody-dependent cellular cytotoxicity inducibility predicts efficacy of cetuximab. Cancer Immunol Res. 2015; 3(5):567-74. doi: 10.1158/2326-6066.CIR-14-0188.
34. McMichael EL, Benner B, Atwal LS, Courtney NB, Mo X, Davis ME, et al. A Phase I/II Trial of Cetuximab in Combination with Interleukin- 12 Administered to Patients with Unresectable Primary or Recurrent Head and Neck Squamous Cell Carcinoma. Clin Cancer Res. 2019 Aug 15;25(16):4955-4965. doi: 10.1158/1078-0432.CCR-18- 2108.
35. Srivastava RM, Trivedi S, Concha-Benavente F, Gibson SP, Reeder C, Ferrone S, et al. CD137 Stimulation Enhances Cetuximab-Induced Natural Killer: Dendritic Cell Priming of Antitumor T-Cell Immunity in Patients with Head and Neck Cancer. Clin Cancer Res. 2017;23(3):707-716. doi: 10.1158/1078-0432.CCR-16-0879.
36. Magnes T, Melchardt T, Hufnagl C, Weiss L, Mittermair C, Neureiter D, et al. The influence of FCGR2A and FCGR3A polymorphisms on the survival of patients with recurrent or metastatic squamous cell head and neck cancer treated with cetuximab. Pharmacogenomics J. 2018;18(3):474-479. doi: 10.1038/tpj.2017.37
37. Magnes T, Melchardt T, Hufnagl C, Weiss L, Mittermair C, Neureiter D, et al . Correction: The influence of FCGR2A and FCGR3A polymorphisms on the survival of patients with recurrent or metastatic squamous cell head and neck cancer treated with cetuximab. Pharmacogenomics J. 2020;20(2):350. doi: 10.1038/s41397-019- 0073-5. Erratum for: Pharmacogenomics J. 2018;18(3):474-479.
38. Pfisterer K, Fusi A, Klinghammer K, Knödler M, Nonnenmacher A, Keilholz U. PI3K/PTEN/AKT/mTOR polymorphisms: association with clinical outcome in patients with head and neck squamous cell carcinoma receiving cetuximab-docetaxel. Head Neck. 2015;37(4):471-8. doi: 10.1002/hed.23604.
39. Muñoz C, Caballero M, Hakim S, Verger E, Grau JJ. Influence of allelic Variations of hypoxia-related and DNA repair genes on patient outcome and toxicity in head and neck cancer treated with radiotherapy plus cetuximab. Eur Arch Otorhinolaryngol. 2016;273(8):2193-9. doi: 10.1007/s00405-015-3740-4.
40. Nelhűbel GA, Cserepes M, Szabó B, Türk D, Kárpáti A, Kenessey I, et al. EGFR Alterations Influence the Cetuximab Treatment Response and c-MET Tyrosine-Kinase Inhibitor Sensitivity in Experimental Head and Neck Squamous Cell Carcinomas. Pathol Oncol Res. 2021; 27:620256. doi: 10.3389/pore.2021.620256.
41. Hsieh YY, Tzeng CH, Chen MH, Chen PM, Wang WS. Epidermal growth factor receptor R521K polymorphism shows favorable outcomes in KRAS wild-type colorectal cancer patients treated with cetuximab- based chemotherapy. Cancer Sci. 2012;103(4):791-6. doi: 10.1111/j.1349-7006.2012.02225.x.
42. Zhang W, Park DJ, Lu B, Yang DY, Gordon M, Groshen S, et al. Epidermal growth factor receptor gene polymorphisms predict pelvic recurrence in patients with rectal cancer treated with chemoradiation. Clin Cancer Res. 2005;11(2 Pt 1):600-5.
43. Gonçalves A, Esteyries S, Taylor-Smedra B, Lagarde A, Ayadi M, Monges G, et al. A polymorphism of EGFR extracellular domain is associated with progression free-survival in metastatic colorectal cancer patients receiving cetuximab-based treatment. BMC Cancer. 2008; 8:169. doi: 10.1186/1471-2407-8-169.
44. Maeda H, Hazama S, Iwamoto S, Oba K, Tsunedomi R, Okayama N, et al. Association between polymorphisms in EGFR and tumor response during cetuximab and oxaliplatin-based combination therapy in metastatic colorectal cancer: Analysis of data from two clinical trials. Oncol Lett. 2019;18(5):4555-4562. doi: 10.3892/ol.2019.10787.
45. Angarita Lozano PA. Análisis de interacción de los polimorfismos CA- SSR-I, -216 G>T y R497K del gen EGFR, estado funcional del gen OIPA y su relación con cáncer gástrico [tesis de maestría]. Chile: Facultad de Medicina, Universidad de Chile; 2014.
46. Costa BM, Viana-Pereira M, Fernandes R, Costa S, Linhares P, Vaz R, et al . Impact of EGFR genetic variants on glioma risk and patient outcome. Cancer Epidemiol Biomarkers Prev. 2011; 20(12):2610-7. doi: 10.1158/1055-9965.EPI-11-0340.
47. Marinović S, Vuković K, Škrtić A, Poljak M, Petek S, Petek L, et al. Epidermal growth factor receptor intron 1 polymorphism and microsatellite instability in sporadic colorectal cancer. Oncol Lett. 2021;21(2):131. doi: 10.3892/ol.2020.12392.
48. Graziano F, Ruzzo A, Loupakis F, Canestrari E, Santini D, Catalano V. Pharmacogenetic profiling for cetuximab plus irinotecan therapy in patients with refractory advanced colorectal cancer. J Clin Oncol. 2008;26(9):1427-34. doi: 10.1200/JCO.2007.12.4602.
49. Loupakis F, Antoniotti C, Cremolini C, Zhang W, Yang D, Wakatsuki T, et al. Prospective study of EGFR intron 1 (CA)n repeats variants as predictors of benefit from cetuximab and irinotecan in chemo- refractory metastatic colorectal cancer (mCRC) patients. Pharmacogenomics J. 2014;14:322–7. doi: 10.1038/tpj.2014.1.
50. Liu W, Innocenti F, Wu MH, Desai AA, Dolan ME, Cook EH Jr, et al. A functional common polymorphism in a Sp1 recognition site of the epidermal growth factor receptor gene promoter. Cancer Res. 2005;65(1):46-53.
51. Dahan L, Norguet E, Etienne-Grimaldi MC, Formento JL, Gasmi M, Nanni I, et al. Pharmacogenetic profiling and cetuximab outcome in patients with advanced colorectal cancer. BMC Cancer. 2011 Nov 25; 11:496. doi: 10.1186/1471-2407-11-496.
52. Mata-Molanes JJ, Rebollo-Liceaga J, Martínez-Navarro EM, Manzano RG, Brugarolas A, Juan M, et al. Relevance of Fc Gamma Receptor Polymorphisms in Cancer Therapy with Monoclonal Antibodies. Front Oncol. 2022; 12:926289. doi: 10.3389/fonc.2022.926289.
53. Zhang W, Gordon M, Schultheis AM, Yang DY, Nagashima F, Azuma M, et al. FCGR2A and FCGR3A polymorphisms associated with clinical outcome of epidermal growth factor receptor expressing metastatic colorectal cancer patients treated with single-agent cetuximab. J Clin Oncol. 2007;25(24):3712-8. doi: 10.1200/JCO.2006.08.8021.
54. Inoue Y, Hazama S, Iwamoto S, Miyake Y, Matsuda C, Tsunedomi R, et al. FcγR and EGFR polymorphisms as predictive markers of cetuximab efficacy in metastatic colorectal cancer. Mol Diagn Ther. 2014; 18(5): 541-8. doi: 10.1007/s40291-014-0103-6.
55. Calemma R, Ottaiano A, Trotta AM, Nasti G, Romano C, Napolitano M, et al. Fc gamma receptor IIIa polymorphisms in advanced colorectal cancer patients correlated with response to anti-EGFR antibodies and clinical outcome. J Transl Med. 2012; 10:232. doi: 10.1186/1479-5876-10-232.
56. Shepshelovich D, Townsend AR, Espin- García O, Latifovic L, O´Callaghan CJ, Jonker DJ, et al. Fc-gamma receptor polymorphisms, cetuximab therapy, and overall survival in the CCTG CO.20 trial of metastic colorectal cancer. Cancer Med.2018; 7(11): 5478-5487. doi: 10.1002/cam4.1819.
57. Liu G, Tu D, Lewis M, Cheng D, Sullivan LA, Chen Z, et al. Fc-γ Receptor Polymorphisms, Cetuximab Therapy, and Survival in the NCIC CTG CO.17 Trial of Colorectal Cancer. Clin Cancer Res. 2016;22(10):2435-44. doi: 10.1158/1078-0432.CCR-15-0414.
58. Tamura K, Shimizu C, Hojo T, Akashi-Tanaka S, Kinoshita T, Yonemori K, et al. FcγR2A and 3A polymorphisms predict clinical outcome of trastuzumab in both neoadjuvant and metastatic settings in patients with HER2-positive breast cancer. Ann Oncol. 2011;22(6):1302- 1307. doi: 10.1093/annonc/mdq585.
59. Paez D, Paré L, Espinosa I, Salazar J, del Rio E, Barnadas A, et al. Immunoglobulin G fragment C receptor polymorphisms and KRAS mutations: are they useful biomarkers of clinical outcome in advanced colorectal cancer treated with anti-EGFR-based therapy? Cancer Sci. 2010;101(9):2048-53. doi: 10.1111/j.1349-7006.2010.01621.x.
60. Ghafouri-Fard S, Noie Alamdari A, Noee Alamdari Y, Abak A, Hussen BM, Taheri M, et al. Role of PI3K/AKT pathway in squamous cell carcinoma with an especial focus on head and neck cancers. Cancer Cell Int. 2022;22(1):254. doi: 10.1186/s12935-022-02676-x.
61. Karar J, Maity A. PI3K/AKT/mTOR Pathway in Angiogenesis. Front Mol Neurosci. 2011; 4:51. doi: 10.3389/fnmol.2011.00051.
62. Moura AC, Assad DX, Amorim Dos Santos J, Porto de Toledo I, Barra GB, Castilho RM, et al. Worldwide prevalence of PI3K-AKT-mTOR pathway mutations in head and neck cancer: A systematic review and meta-analysis. Crit Rev Oncol Hematol. 2021; 160:103284. doi: 10.1016/j.critrevonc.2021.103284.
63. Patel U, Pandey M, Kannan S, Samant TA, Gera P, Mittal N. Prognostic and predictive significance of nuclear HIF1α expression in locally advanced HNSCC patients treated with chemoradiation with or without nimotuzumab. Br J Cancer. 2020;123(12):1757-1766. doi: 10.1038/s41416-020-01064-4.
64. Emami Nejad A, Najafgholian S, Rostami A, Sistani A, Shojaeifar S, Esparvarinha M, et al. The role of hypoxia in the tumor microenvironment and development of cancer stem cell: a novel approach to developing treatment. Cancer Cell Int. 2021;21(1):62. doi: 10.1186/s12935-020-01719-5
65. Roy S, Kumaravel S, Sharma A, Duran CL, Bayless KJ, Chakraborty S. Hypoxic tumor microenvironment: Implications for cancer therapy. Exp Biol Med (Maywood). 2020;245(13):1073-1086. doi: 10.1177/1535370220934038.
66. Muz B, de la Puente P, Azab F, Azab AK. The role of hypoxia in cancer progression, angiogenesis, metastasis, and resistance to therapy. Hypoxia (Auckl). 2015; 3:83-92. doi: 10.2147/HP.S93413.
67. Lee P, Chandel NS, Simon MC. Cellular adaptation to hypoxia through hypoxia inducible factors and beyond. Nat Rev Mol Cell Biol. 2020; 21(5):268-283. doi: 10.1038/s41580-020-0227-y.
68. Qin C, Cao Q, Ju X, Wang M, Meng X, Zhu J, et al. The polymorphisms in the VHL and HIF1A genes are associated with the prognosis but not the development of renal cell carcinoma. Ann Oncol. 2012;23(4):981-9. doi: 10.1093/annonc/mdr325.
69. Guo X, Li D, Chen Y, An J, Wang K, Xu Z, et al. SNP rs2057482 in HIF1A gene predicts clinical outcome of aggressive hepatocellular carcinoma patients after surgery. Sci Rep. 2015; 5:11846. doi: 10.1038/srep11846.
70. Mandal RK, Mittal RD. Genetic Variant Arg399Gln G>A of XRCC1 DNA Repair Gene Enhanced Cancer Risk Among Indian Population: Evidence from Meta-analysis and Trial Sequence Analyses. Indian J Clin Biochem. 2018;33(3):262-272. doi: 10.1007/s12291-017- 0669-y.
71. Nanda SS, Gandhi AK, Rastogi M, Khurana R, Hadi R, Sahni K, et al. Evaluation of XRCC1 Gene Polymorphism as a Biomarker in Head and Neck Cancer Patients Undergoing Chemoradiation Therapy. Int J Radiat Oncol Biol Phys. 2018;101(3):593-601. doi: 10.1016/j.ijrobp.2018.03.039.
72. Zhang Z, Zheng F, Yu Z, Hao J, Chen M, Yu W, et al. XRCC5 cooperates with p300 to promote cyclooxygenase-2 expression and tumor growth in colon cancers. PLoS One. 2017 ;12(10): e0186900. doi: 10.1371/journal.pone.0186900.
73. Ulusan M, Sen S, Yilmazer R, Dalay N, Demokan S. The let-7 microRNA binding site variant in KRAS as a predictive biomarker for head and neck cancer patients with lymph node metastasis. Pathol Res Pract. 2022;239:154147. doi: 10.1016/j.prp.2022.154147.
74. Zhang W, Winder T, Ning Y, Pohl A, Yang D, Kahn M, et al. A let-7 microRNA-binding site polymorphism in 3′-untranslated region of KRAS gene predicts response in wild-type KRAS patients with metastatic colorectal cancer treated with cetuximab monotherapy. Ann Oncol. 2011 Jan;22(1):104-109. doi: 10.1093/annonc/mdq315.
75. Christensen BC, Moyer BJ, Avissar M, Ouellet LG, Plaza SL, McClean MD, et al . A let-7 microRNA-binding site polymorphism in the KRAS 3′ UTR is associated with reduced survival in oral cancers. Carcinogenesis. 2009;30(6):1003-7. doi: 10.1093/carcin/bgp099.
76. Sebio A, Paré L, Páez D, Salazar J, González A, Sala N, et al. The LCS6 polymorphism in the binding site of let-7 microRNA to the KRAS 3′-untranslated region: its role in the efficacy of anti-EGFR-based therapy in metastatic colorectal cancer patients. Pharmacogenet Genomics. 2013 Mar;23(3): 142-7.doi: 10.1097/FPC.0b013e32835d9b0b.
77. Saridaki Z, Weidhaas JB, Lenz HJ, Laurent-Puig P, Jacobs B, De Schutter J, et al. A let-7 microRNA-binding site polymorphism in KRAS predicts improved outcome in patients with metastatic colorectal cancer treated with salvage cetuximab/panitumumab monotherapy. Clin Cancer Res. 2014;20(17):4499-4510. doi: 10.1158/1078- 0432.CCR-14-0348.
78. De Ruyck K, Duprez F, Ferdinande L, Mbah C, Rios-Velazquez E, Hoebers F, et al. A let-7 microRNA polymorphism in the KRAS 3′- UTR is prognostic in oropharyngeal cancer. Cancer Epidemiol. 2014;38(5):591-8. doi: 10.1016/j.canep.2014.07.008.
79. Ying HQ, Wang F, He BS, Pan YQ, Gao TY, Xu YQ. The involvement of Kras gene 3′-UTR polymorphisms in risk of cancer and influence on patient response to anti-EGFR therapy in metastatic colorectal cancer: a meta-analysis. Onco Targets Ther. 2014; 7:1487-96. doi: 10.2147/OTT.S65496.