RESUMEN:
Las proteínas inactivadoras de ribosomas (RIPs del acrónimo “ribosome-inactivating proteins”) son enzimas (N-glicosidasas clasificadas como ARNr N-glicohidrolasas, EC 3.2.2.22), en su mayor parte de origen vegetal, aunque también de origen bacteriano, fúngico y de algas que provocan la inhibición irreversible de la biosíntesis de proteínas llevada a cabo por los ribosomas de organismos superiores y algunas bacterias. La actividad enzimática consiste en la depurinación específica de los ribosomas que los incapacita para interaccionar con el factor de elongación 2 en eucariontes y G en procariontes. Las RIPs más conocidas, ricina y abrina constituidas por dos cadenas polipeptídicas (tipo2), la cadena enzimática y una lectina específica de D-galactosa y galactósidos, son extremadamente tóxicas debido a que pueden internalizarse en las células al unirse a receptores de la membrana plasmática, entrar al citoplasma, alcanzar el aparato de Golgi y después llegar al retículo endoplásmico rugoso, en donde son transferidas al citosol y allí inactivar a los ribosomas. Las RIPs más abundantes solo poseen una cadena enzimática (tipo 1) y no poseen la toxicidad de ricina y abrina. En la actualidad se desconoce su función biológica en vegetales, aunque se han propuesto distintas hipótesis sobre su mediación en respuesta de estrés vegetal y frente a patógenos. Las RIPs han encontrado una aplicación muy interesante en la construcción de inmunotoxinas y conjugados en la terapia experimental del cáncer. La formulación de diversas RIPs en nanopartículas se ha empleado para optimizar sus propiedades biofarmacéuticas y farmacocinéticas y también con fines diagnósticos.
Palabras clave: RIP; enzima; función; inmunotoxina; vectorización
ABSTRACT:
Ribosome-inactivating proteins (RIPs) are enzymes (N-glycosidases classified as rRNA N-glycohydrolases, EC 3.2.2.22), mostly of plant origin, but also of bacterial, fungal and algal origin, which cause irreversible inhibition of protein biosynthesis carried out by ribosomes of superior organisms and some bacteria. The enzymatic activity consists of specific depurination of ribosomes that renders them unable to interact with elongation factor 2 in eukaryotes and elongation factor G in prokaryotes. The best-known RIPs, ricin and abrin, consisting of two polypeptide chains (type 2), an enzymatic chain and a lectin chain specific for D-galactose and galactosides, are extremely toxic because they can be internalised in cells by binding to plasma membrane receptors, entering the cytoplasm, reaching the Golgi apparatus, and then reaching the rough endoplasmic reticulum, where they are transferred to the cytosol and there inactivate ribosomes. The most abundant RIPs possess only one enzymatic chain (type 1) and do not possess the toxicity of ricin and abrin. Their biological function in plants is currently unknown, although different hypotheses have been proposed for their mediation in plant stress response and against pathogens. RIPs have found an interesting application in the construction of immunotoxins and conjugates in experimental cancer therapy. The formulation of RIPs into nanoparticles, has been used to optimize their biopharmaceutical and pharmacokinetic properties and for diagnostic purposes.
Keywords: RIP; enzyme; function; inmunotoxin; drug targeting
1. INTRODUCCIÓN
Las proteínas inactivadoras de ribosomas (RIPs) constituyen un campo de investigación floreciente que se ha disparado exponencialmente en los últimos cuarenta años. El primer contacto del laboratorio de Valladolid con dichas proteínas se realizó en 1987, cuando después de estudiar una revisión de Antonio Jiménez y David Vázquez en el Annual Review of Microbiology de 1985 me pareció un buen tema de investigación para desarrollar en el incipiente laboratorio experimental que estaba organizando. En dicha revisión se abordaban ricina y abrina, dos poderosas toxinas inhibidoras de la biosíntesis de proteínas, así como otras proteínas que podría serlo también (1). Me animó el hecho de que durante mis estudios sobre la biosíntesis de proteínas en Escherichia coli en el laboratorio de David Vázquez, tuve la ocasión de conocer y tratar en 1974 al Prof. Sjur Olness, probablemente el mayor conocedor de la ricina en ese momento. En 1988, contacté con el Prof. Fiorenzo Stirpe del Departamento de Patología Experimental de la Universidad de Bolonia, que estaba en esos momentos en una fructífera investigación sobre proteínas inhibidoras de la biosíntesis de proteínas de una cadena y me invitó a su laboratorio un par de meses en 1988. Esto me inclinó a implicarme totalmente en el estudio de este tipo de proteínas, su distribución en vegetales, su estructura y función y sus aplicaciones.
En los últimos veinte años, han aparecido una serie de revisiones exhaustivas (2-11) que cubren la investigación central sobre las RIPs. A pesar de su distribución aparentemente amplia, especialmente en el reino vegetal, no hay datos de detección sistemáticos que respalden su ubicuidad. Las primeras RIPs identificadas fueron la ricina y la abrina, dos toxinas potentes estudiadas por Paul Ehrlich, pero solo en 1971 se encontró que la ricina inhibe la síntesis de proteínas eucariotas dañando los ribosomas (1, 12). Desde entonces se han identificado y aislado muchas otras proteínas con propiedades aparentemente idénticas, que desde un punto de vista estructural, se dividieron en dos grandes grupos: RIP tipo 1 y tipo 2 (2-5). El tipo 1, más numeroso, son proteínas de cadena única, fuertemente básicas, de 30 kDa, aproximadamente, que tienen actividad enzimática. Inhiben la síntesis de proteínas libres de células, pero son relativamente no tóxicas para las células y los animales. Las RIPs de tipo 2 son proteínas de 60-65 kDa, de un mínimo de dos cadenas polipeptídicas (AB) en las que una cadena enzimática A, similar a las RIPs de tipo 1, está unida a una cadena B ligeramente más grande con actividad de lectina específica para azúcares generalmente con la estructura de D-galactosa libre terminal (4); algunas como SNA I y las proteínas tetraméricas relacionadas (AB)2 se unen a terminaciones de ácido siálico (5-6). La cadena B se une a las membranas celulares, facilitando así la entrada de las RIPs AB o (AB)2 completas en las células. Por lo tanto, este grupo incluye ricina, abrina y otras toxinas potentes encontradas posteriormente, pero también nigrina b de Sambucus nigra L. (13), ebulina l de Sambucus ebulus L. (14) y otras proteínas identificadas más recientemente, que tienen una estructura similar y propiedades enzimáticas, y son mucho menos tóxicas que la ricina. Por otro lado, algunos autores han postulado la creación de una nueva categoría, las RIPs tipo 3, para incluir algunas proteínas como la RIP b-32 o la JIP60. La primera de ellas se aísla del maíz como proenzima y se activa después de la eliminación de un segmento peptídico interno corto que libera dos segmentos de 16,5 y 8,5 kDa (15). Por su parte, la JIP60 es una RIP de cebada, en el que un segmento similar al RIP tipo 1 continúa con otro segmento de tamaño similar sin una función conocida (16). Parece injustificado definir un nuevo tipo de proteínas sobre la base de casos únicos y diferentes, y por el momento parece preferible considerar estas dos proteínas como RIPs peculiares de tipo 1.
Los dos tipos de RIP 1 y 2 podrían diferenciarse más adecuadamente sobre la base de la ausencia (tipo 1) o la presencia (tipo 2) de una cadena de lectinas, lo que marca una diferencia significativa en su modo de interaccionar con las células. Esta clasificación permite encajar fácilmente otros ejemplos de RIP. Desde este punto de vista, las RIPs de cuatro cadenas como la SNA I (17) y la SNA I’ (18) deben clasificarse como RIP de tipo 2. Además, algunas proteínas también pueden incluirse en la categoría RIP tipo 2, como SNRLP 1 y 2 (19) y la nigrina b básica (20), que exhiben la estructura clásica de dos cadenas AB, pero no pueden aglutinar los glóbulos rojos, debido muy probablemente a algún tipo de defecto en los dominios de unión de azúcar en la cadena B.
2. DISTRIBUCIÓN ENTRE PLANTAS, HONGOS, ALGAS Y BACTERIAS
El trabajo realizado en los últimos años reveló que las RIPs, están ampliamente distribuidos entre plantas, hongos, algas y bacterias (2-7, 11), y recientemente también se ha descrito una actividad de tipo RIP en tejidos de mamíferos (21). Las RIPs están presentes en una gama bastante amplia de familias aparentemente no relacionadas desde un punto de vista filogenético. La investigación sobre las RIPs hasta ahora se ha centrado en algunos pocos géneros y familias. Las familias más investigadas han sido: Caryophyllaceae, Cucurbitaceae, Fabaceae, Euphorbiceae, Phytolaccaceace, Poaceae, Sambucaceae, Viscaceae, en algas Laminariaceae y en basiodiomicetos, Pleurotaceae y Tricholomataceae (5, 6). En estas familias se han identificado diversas RIPs, en su mayoría isoformas, aunque muchas de ellas poseen actividades específicas distintas. Esto no indica una distribución limitada de estas proteínas, ni un papel relativamente poco importante en las plantas. Esto podría ser simplemente una consecuencia del procedimiento de cribado que se llevó a cabo para encontrar plantas con altas concentraciones de actividad RIP precisamente en aquellas familias que anteriormente se había demostrado contener esta actividad en lugar de analizar de manera más amplia su distribución en todos los reinos, como se hace en la actualidad. Con este nuevo enfoque, se ha puesto de manifiesto que aproximadamente un tercio de todos los órdenes de plantas con flores contienen RIPs y que éstas, se pueden agrupar en quince categorías con diferentes características y propiedades, atendiendo a sus diversos dominios proteicos (2-6, 10, 11; estudios no publicados de Stirpe y Girbés). Nuestro laboratorio en la Universidad de Valladolid ha contribuido con el aislamiento de 24 RIPs de tipo 1 y 2 en 5 especies de Sambucus, así como otras 10 RIPs de tipo 1 en endemismos del norte de España del género Petrocoptis, en Beta vulgaris, en Marah oreganus, en Trichosanthes kirilowii y en Muscari armeniacum (2, 6). La baja actividad, si la hay, observada en muchos extractos de plantas puede deberse a una baja concentración de RIP en un extracto en particular o simplemente a la baja actividad del RIP presente. Probablemente, el uso de los fluidos supercríticos a baja temperatura como tecnología alternativa de extracción, permitiría aislar nuevas RIPs o RIPs funcionales en estos substratos. Incluso no puede excluirse que existan proteínas originalmente con actividad RIP que hayan perdido esta actividad y por lo tanto no pueden detectarse solo buscando la actividad enzimática. Esto podría haber restringido el enfoque del estudio a un número reducido de familias de plantas que se sabe desde hace muchos años que incluyen plantas que contienen RIP en varios de sus tejidos y, en algunos casos, en grandes cantidades, por ejemplo, Saponaria officinalis (22) y Sambucus nigra (13, 17-20), dos especies que contienen numerosas RIPs en hojas semillas y frutos, habiéndose aislado un gran número de estas proteínas de un solo tejido de la planta. Esto también podría haber reducido la visión de las RIPs como componentes potenciales de mecanismos aún desconocidos o no bien aclarados ampliamente representados en las plantas y tal vez en otros organismos como, por ejemplo, el papel antipatogénico en plantas desempeñado por las RIPs (23). El hecho de que algunas RIP puedan ser inducidas por diferentes factores como la senescencia (24), la infección por virus (25) y el estrés (26) apoya esta creencia. En nuestra opinión, probablemente las RIPs reproducen efectos pleiotrópicos que pueden estar orquestados por mecanismos aún desconocidos. Claramente, se necesita más investigación sobre ellas para determinar una imagen más apropiada sobre la distribución y la importancia de las RIPs en todos los reinos (7, 11).
Los pesos moleculares de las RIPs tipo 1 se encuentran normalmente en el rango de 21-38.000 Da, excepto una serie de proteínas de bajo peso molecular que se alega que también son RIPs, con masas relativas aparentes en el rango de 8-13.500 Da. En lo que respecta a las RIPs de tipo 2, los pesos moleculares de estas proteínas se encuentran en el rango de masas relativas aparentes de 56-69.000 Da para las de tipo AB y de 120-240 kDa para las de tipo (AB)2. Se sabe desde hace muchos años que las RIPs también están presentes en bacterias. Así, las RIPs denominadas Stx1 y Stx2, se han aislado de Escherichia coli y actúan enzimáticamente como las RIPs de las plantas (27, 28). Por otro lado, las RIPs de tipo 1 se han encontrado también en los últimos años en hongos (29, 30) y en el alga Laminaria japonica A (31). Todos estos hallazgos favorecen la hipótesis generalmente aceptada de que las RIPs están ampliamente representadas y, por lo tanto, deben desempeñar funciones biológicas importantes, aunque en general aún poco definidas.
De los estudios realizados hasta ahora parece que las RIPs tipo 1 podrían ser más abundantes que las RIPs tipo 2. De hecho, hasta el descubrimiento de las RIPs tipo 2 en Sambucus (2), solo se habían descrito la ricina, la abrina, la viscumina, la módeccina y volkensina, las cinco altamente tóxicas para células animales y roedores (4, 5). En los primeros tiempos de la investigación sobre la distribución de las RIPs de tipo 2 se seguía el principio de que estas proteínas eran extremadamente tóxicas. Ello limitaba el cribado de las especies a las plantas venenosas o muy tóxicas, como Ricinus communis, Abrus precatorius, etc. Muy probablemente esto ha llevado a subestimar la presencia de estas proteínas en plantas no tóxicas, por lo que probablemente muchas de estas RIPs de tipo 2 han pasado desapercibidas, como tuvimos ocasión de demostrar con el descubrimiento del grupo especial de RIPs de tipo 2 no tóxicas respecto a la ricina en el género Sambucus constituido por nigrinas y ebulinas (16, 17). Las proteínas de estos subgrupos tienen la característica extraordinaria de que, aunque son aún más activos que la ricina a nivel molecular, es decir sobre la inactivación enzimática del ribosoma, carecen de la alta toxicidad de la ricina hacia las células animales cultivadas e in vivo en roedores. Por ejemplo, la concentración que provoca el 50% de inhibición de la síntesis de proteínas (IC50) en un sistema libre de células (lisado de reticulocitos de conejo) de nigrina b es de 0,03 nM, en células HeLa es de 27,6 nM y la dosis letal 50% (DL50) en ratones es de 12 mg por kg de peso corporal (38). Estos valores significan que la nigrina b es tres veces más activa a nivel ribosómico que la ricina, pero es 25.000 veces menos tóxica en células animales cultivadas y 1.500 veces menos tóxica para ratones que la ricina (39).
El reconocimiento de que las plantas pueden tener RIP de tipo 2 no tóxicas en el género Sambucus (2, 13, 14) animó el estudio de este género en profundidad. Los estudios realizados indicaron que tanto nigrina b como ebulina l eran proteínas del tipo AB en donde A es una cadena polipeptídica con actividad enzimática, el mismo tipo de actividad que la ricina capaz de promover la depuración de la A4324 del ARNr 28S del ribosoma hepático de rata, lo que provoca la inactivación inmediata del ribosoma y por lo tanto la inhibición de la síntesis de proteínas (2, 13, 14). La cadena B es un polipéptido con actividad de lectina y afinidad por D-galactosa y galactósidos (2). Los experimentos de captación e internalización de la cadena A de ricina y la ebulina en cultivos celulares sugieren que la cadena A en general no tiene señales de translocación especiales para interaccionar con la membrana plasmática, en cambio, las cadenas B si las tienen (32). Por lo tanto, la citotoxicidad reducida de la ebulina l en comparación con la ricina podría deberse a deficiencias de la cadena B (figura 1).
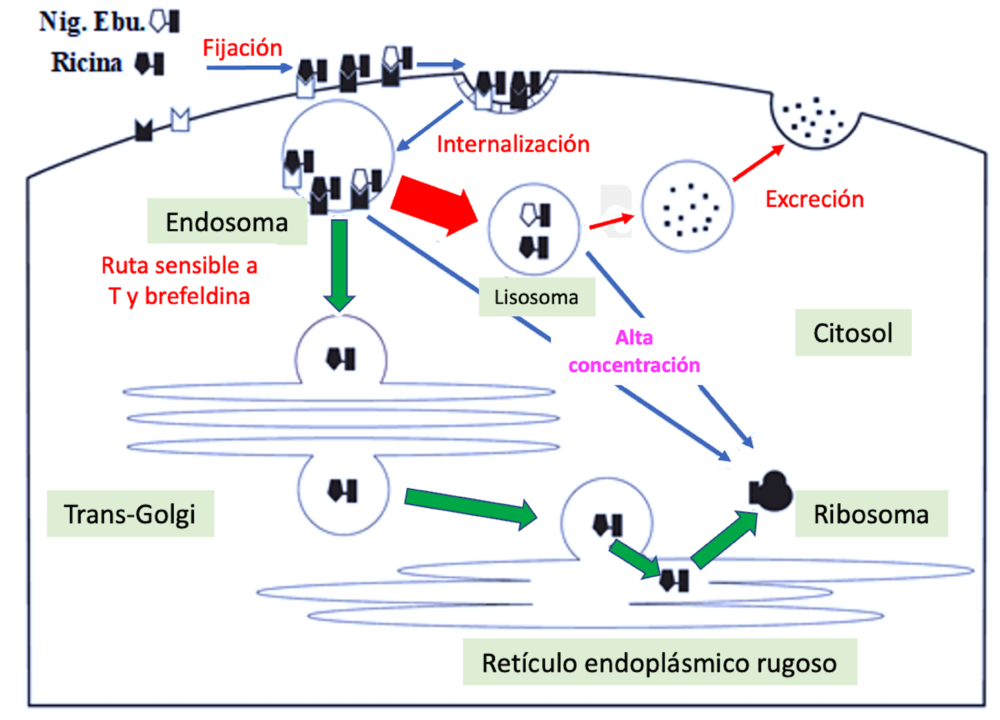
Figura 1. Esquema general de las rutas intracelulares seguidas por la ricina, nigrina y ebulina. Las flechas indican las rutas de tránsito.
La clonación molecular, el estudio de la secuencia de aminoácidos y de la estructura cristalina por difracción de rayos X a la resolución de 2.8-A de la ebulina l revelaron cambios en los aminoácidos clave de los subdominios de unión al azúcar de la cadena B, especialmente en el subdominio de alta afinidad de unión al azúcar 2γ en el que el Tyr249 de ricina se cambia a Phe en la ebulina l (33). Esto reduce la afinidad de la ebulina por los galactósidos y, por lo tanto, por las glicoproteínas que contienen galactosa ubicadas en la superficie de la membrana plasmática.
3. COEXISTENCIA DE RIPS DE TIPO 1 Y 2
Las RIPs tipo 1 pueden coexistir con las de tipo 2 como se demostró en al menos en tres especies de Sambucus y posteriormente en Cinnamomum camphora, e Iris hollandica. Ello sugiere que esta circunstancia podría ser reflejo de un fenómeno más general. Los frutos de S. nigra contienen las nigritinas tipo 1 f1 de naturaleza constitutiva y f2 de naturaleza inducible por maduración (34). Los estudios preliminares indicaron que ambas nigritinas se acumulan en la piel de los frutos, mientras que la nigrina RIP tipo 2 f está en el interior acuoso del fruto. Por otro lado, las hojas de S. ebulus contienen las ebulitinas tipo 1, α, β y γ (35). Dicha coexistencia de las RIPs de tipo 1 y tipo 2 se ha descrito en Cinnamomum camphora en la que se han descrito las RIPs cinamomina (tipo 2) y canforina (tipo 1), pero a diferencia de nigritinas y ebulitinas, parece ser que canforina es de hecho la cadena A del cinamomina (36). Iris hollandica también contiene RIP de tipo 1 (IRIP 1,2,3) y tipo 2 (IRAb e IRAr, dos aglutininas con afinidad por Gal/N-acetilgalactosamina y manosa) (37). En la actualidad aún no se conoce el significado de la presencia simultánea de RIPs tipo 1 y tipo 2 en el mismo tejido.
4. ACTIVIDADES ENZIMÁTICAS DE RIPS
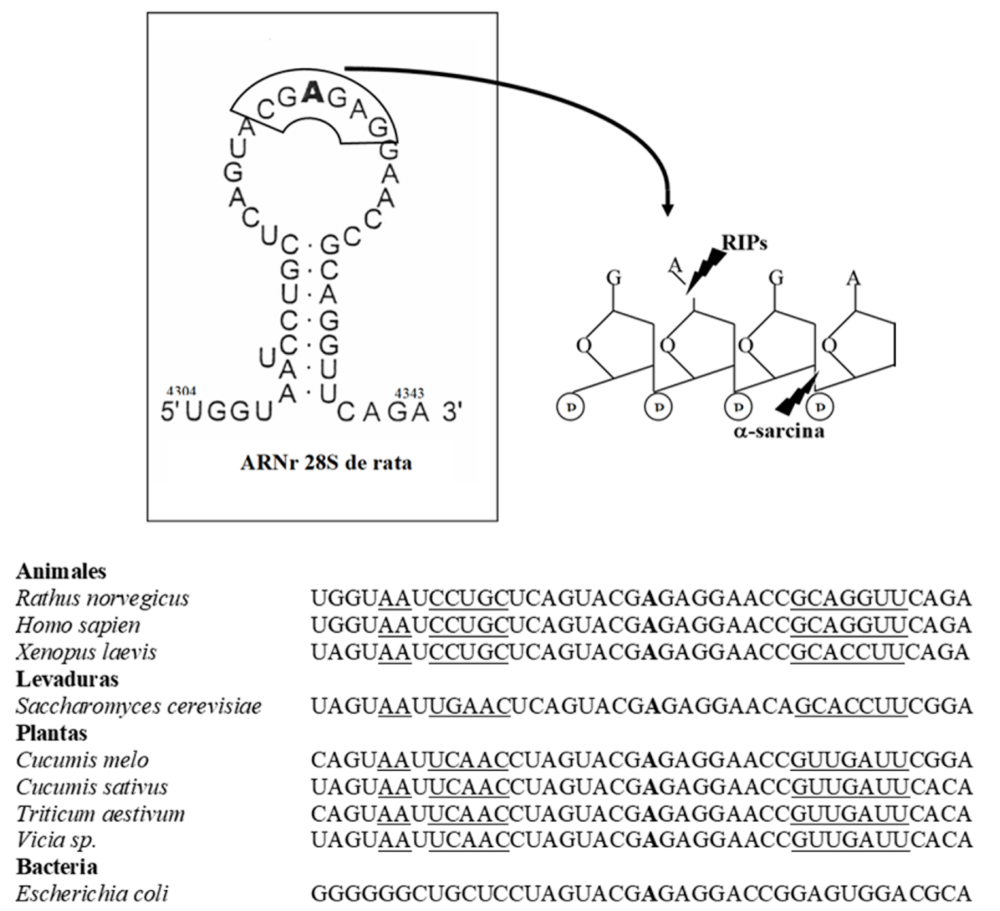
Las RIPs muestran una serie de actividades enzimáticas, la mayoría de ellas pueden ser simplemente un reflejo de su actividad en el ARN y otras completamente nuevas. Estas actividades han sido revisadas recientemente (7,8,10). A pesar de la mayor parte de la investigación realizada sobre RIPs desde el descubrimiento de la ricina (3, 4) fue en 1987, cuando el grupo de Endo publicó el mecanismo molecular de acción de la ricina involucrado en la inhibición de la síntesis de proteínas (38) (figura 2).
Figura 2. Secuencia de nucleótidos del bucle altamente conservado del ARNr sobre el que actúan las RIPs. En la parte superior izquierda se muestra la estructura secundaria del ARNr 28S de rata en el cual la adenina (señalada en negrita) se elimina por acción de la RIP, En la parte inferior de la figura se muestran las secuencias homólogas pertenecientes a ribosomas sensibles a la acción de las RIPs. Los nucleótidos subrayados a la derecha de la adenina son complementarios de los situados a su izquierda (39).
Las RIPs son N-glicosidasas que rompen el enlace N-glicosídico que une el A4324 con la cadena de polifosfato del ARNr 28S del ribosoma hepático de rata (Figura 2 superior) (38, 40). Esta adenina se encuentra en un bucle que está altamente conservado en las diferentes especies, llamado sitio de ricina/α-sarcina que es esencial para la interacción de los factores de elongación G y 2 con los ribosomas procariota y eucariota, respectivamente, por esta razón la inactivación del ribosoma deja a la cadena polipeptídica naciente en el sitio A (2, 40). En esta línea, se ha demostrado que la estabilización de EF-G sobre ribosomas de Escherichia coli con el antibiótico ácido fusídico protege al ribosoma de la depuración por crotina 2, una RIP que inactiva los ribosomas bacterianos (41).
Muchas RIP inactivan también los ribosomas de insectos (42), hongos (43), bacterias (44-45) y plantas (46), a través de un mecanismo de acción similar que actúa en mamíferos (2). Aunque no hay estudios sistemáticos publicados sobre la sensibilidad de los ribosomas a las RIP, los datos disponibles indican que los ribosomas de mamíferos son mucho más sensibles que los ribosomas vegetales y bacterianos (2). Un examen de la sensibilidad relativa de los ribosomas de Escherichia coli, Agrobacterium tumefaciens, Brevibacterium lactofermentum y Streptomyces lividans reveló que son sensibles a unos pocas RIP de tipo 1, pero no a otras, o a las RIPs de tipo 2 intactas (2, 45-47). Esto ha obstaculizado la clonación molecular y la expresión de tales RIP inhibitorias en los sistemas comunes de expresión bacteriana (48, 49).
El bucle blanco para las RIPs es también el objetivo para otros inhibidores de la síntesis de proteínas como las RNasas de la familia de la α-sarcina, a saber, α-sarcina, restrictocina y tricolina (38, 40, 50, 51), que hidrolizan el enlace fosfodiéster en el extremo 3’ del G4325 del 28S ARNr. Algunas RIP promueven la liberación de una adenina por ribosoma, suficiente para inactivarlo, mientras que otros pueden liberar también otras adeninas del ribosoma, proceso este que no parece estar correlacionada con la capacidad inhibitoria de la RIP (52).
Se ha encontrado un nuevo tipo de enzima en algunas especies de plantas que producen RIPs, la llamada RALyasa. Esta enzima promueve de manera específica la hidrólisis del enlace fosfodiéster en el extremo 3’-terminal del sitio apurinico generado por la acción de la RIP autóloga. Se ha sugerido que este enzima podría formar parte de algún sistema de reparación del ARNr similar o equivalente al encontrado para la reparación del ADN (53-56), y que actuaría en los ribosomas que resultasen inactivados accidentalmente por las RIPs autólogas. Las RIP pueden eliminar las adeninas no solo de los ribosomas sino también de cualquier tipo de ácido nucleico, a saber, ARNr, ARNt, ARNm, ARN viral e incluso ADN (57, 58). Tal actividad llevó a cambiar el nombre de las RIPs con la denominación más significativa y sistemática de las glicosilasas polinucleótidos de adenina (2). Esta actividad enzimática de sustrato amplio podría ser responsable de algunas propiedades que se han atribuido a las RIPs durante los últimos años, como la actividad antiviral, la promoción de la senescencia y la apoptosis por modificación del ADN (2, 7).
5. ACTIVIDADES ENZIMÁTICAS NO ANTI-RIBOSOMICAS
5.1. Acción específica sobre el ARNm
Efectos específicos de las RIPs sobre el ARNm diferentes de la actividad de la adenina polinucleótido glicosilasa se han descrito recientemente para la PAP. PAP es capaz de unirse a las estructuras “cap” del mensajero y luego promover la depuración de ARNm en un sentido de 5’- 3’, inactivando así el ARNm (59). Esto permite que la PAP también regule el nivel de expresión de su propio ARNm (60).
5.2. Actividades de lipasa y trastorno de membranas
Las interacciones entre las RIPs y las membranas biológicas se han descrito en los últimos años. Las investigaciones más intensas se han realizado con ricina y proteínas relacionadas. Se ha descrito que la cadena A de ricina se une a vesículas de fosfolípidos cargadas negativamente y desestabiliza las bicapas lipídicas (61). El trabajo realizado con el cinnamomum RIP tipo 2 indica que la proteína forma canales catiónicos en las bicapas lipídicas (62). Estudios más recientes indicaron que la ricina muestra actividad de lipasa, que podría estar relacionada con la translocación a través de las membranas intracelulares (63). Tal actividad de lipasa parece estar relacionada con residuos específicos de aminoácidos pertenecientes tanto a la cadena A como a la B, y se ha argumentado que simplemente la mutación a alanina de la serina catalítica 221 en la cadena A redujo fuertemente la actividad de la lipasa de ricina (64). La ausencia de aminoácidos equivalentes en algunos RIP tipo 2 como la ebulina l y el VVA podría ser la base molecular de la falta o la menor toxicidad de tales proteínas en comparación con la ricina (64).
5.3. Enzimas bifuncionales
Se ha descrito que los cultivos celulares de Trichosanthes kirilowii producen enzimas bifuncionales de defensa vegetal con actividades de quitinasa y RIP (65). Hasta la fecha no hay más estudios sobre la aparición de las proteínas bifuncionales a pesar de la relevancia potencial de este nuevo tipo de proteína que podría desempeñar un papel en la resistencia a los patógenos fúngicos.
5.4. Inhibidores de otras enzimas
Las RIPs se han citado también como inhibidores de otras enzimas. Entre ellos se ha reportado la inhibición de la poli (ADP-ribosa) polimerasa involucrada en la reparación del ADN (66). Tal inhibición se ejerce sobre la fracción poli (ADP-ribosa) unida a la enzima, y se ha sugerido que tal efecto podría ser la base de las actividades transformadoras, apoptóticas y quizás antivirales atribuidas a algunos RIP. Otra enzima cuya actividad es inhibida por las RIPs es la integrasa del VIH-1. Lee-Huang y sus colegas encontraron que algunos RIP son capaces de inhibir las tres reacciones parciales involucradas en la actividad del VIH-1, a saber: i) procesamiento 3’-terminal; ii) Integración del ADNc vih-1 en el ADN aceptor; iii) desintegración del ADN pro-viral. (67-68). Otras actividades enzimáticas, que se ha informado que son sensibles a RIP como la telomerasa (69) y la subperóxido dismutasa (70). Deben realizarse estudios adicionales para determinar si tales efectos son un atributo general de las RIPs o, en cambio, son simplemente peculiaridades.
6. INMUNOTOXINAS Y CONJUGADOS CONSTRUIDOS CON RIPS PARA LA TERAPIA DEL CANCER
La cirugía, radioterapia y quimioterapia constituyen las herramientas de la terapia clásica para el cáncer, pero resultan métodos muy agresivos a causa de los efectos colaterales. Para que estas terapias sean efectivas se requiere que las células malignas difieran suficientemente de las células normales, en su crecimiento o en su metabolismo, lo que no siempre sucede. Sin embargo, incluso en estos casos más desfavorables, las células tumorales expresan receptores de superficie u otras moléculas que las distinguen de los tejidos sanos. El objetivo de la terapia con inmunotoxinas es precisamente enviar selectivamente un agente citotóxico a estas moléculas de superficie con la finalidad de que las internalicen y provoquen la muerte celular (71-74).
Las inmunotoxinas químicas son conjugados formados por un ligando capaz de reconocer específicamente un tipo de células, y una toxina capaz de matar las células a las que se une el ligando. El ligando puede ser un anticuerpo monoclonal, una lectina, un factor de crecimiento, una hormona, un carbohidrato, o antígenos y receptores. En la figura 3 se representan esquemas de las inmunotoxinas, conjugados y especies recombinantes conteniendo RIPs o fragmentos activos de las mismas.
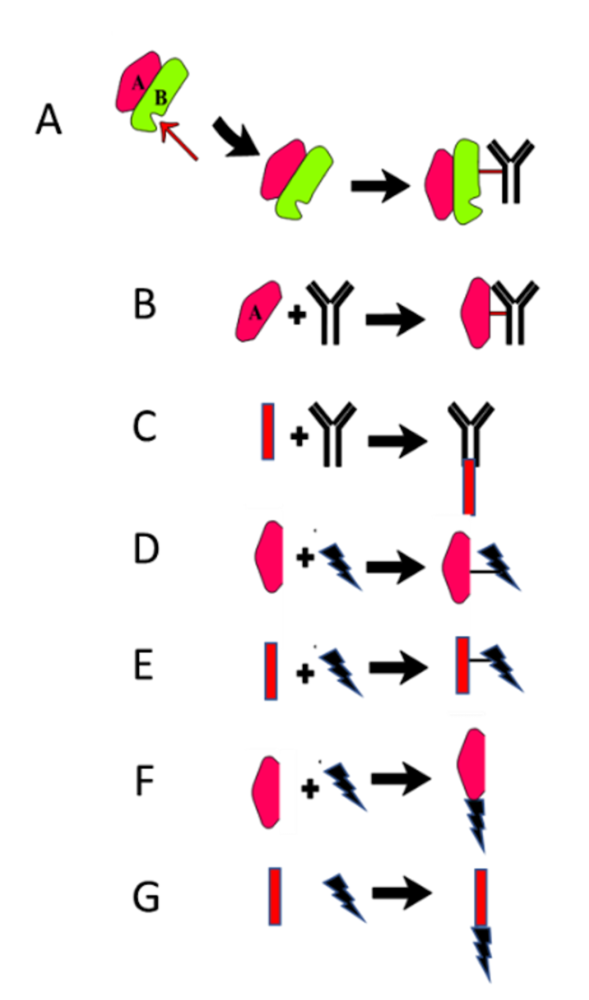
Figura 3. Inmunotoxinas y conjugados con RIPs intactas o sus fragmentos. A) inmunotoxinas con RIPs de tipo 2; B) inmunotoxinas con RIPs de una cadena; C) inmunotoxinas con fragmentos activos de RIPs; D) conjugado químico con moléculas conductoras, factores de crecimiento, etc.; E) Conjugados con moléculas conductoras, factores de crecimiento, etc.; F) Especies recombinantes con RIP intacta y moléculas conductoras, factores de crecimiento, etc.; G) Especies recombinantes con fragmentos activos de RIP y moléculas conductoras, factores de crecimiento, etc
Las inmunotoxinas químicas se construyen uniendo el ligando y la toxina mediante reacción química. La conjugación química de la RIP y la molécula conductora se realiza, por lo general, a través de puentes disulfuro reducibles insertados mediante agentes ligantes homobifuncionales (como el glutaraldehído o el dietilmalonimidato) o heterobifuncionales, como por ejemplo succinimidil-piridil-ditiopropionato (SPDP) y el 2-iminotiolano (2-IT). Con independencia del agente ligante empleado, la unión entre el ligando y la toxina debe ser suficientemente estable para permanecer intacta hasta que la inmunotoxina alcance las células blanco (75). Por otro lado, se han construido inmunotoxinas por vía de ADN recombinante en sus múltiples variantes (76), con RIPs clonadas en bacterias o levaduras, RIPs recombinantes con deleciones en la cadena, RIPs enteras o fragmentos derivados fundidas a moléculas conductoras, etc. Las moléculas conductoras pueden ser hormonas, factores de crecimiento, fragmentos de anticuerpos monoclonales, etc. Aprovechando estas tecnologías se están mejorando las actividades farmacológicas de las RIPs (9, 10), así como las formas de administración para mejorar la interacción con las células (77).
El mecanismo general de acción en las inmunotoxinas que tienen como blanco la biosíntesis de proteínas es el mismo en todas ellas. La inmunotoxina o conjugado se une a la célula blanco, se internaliza por endocitosis mediada por receptor y la parte toxica (RIP) es liberada al citosol donde inactiva los ribosomas provocando la muerte de la célula (75). Cada inmunotoxina o conjugado sigue una ruta intracelular distinta en función del elemento conductor que utilice, hasta el punto de que una misma inmunotoxina puede seguir diferentes rutas dependiendo de la línea celular tratada, lo que se traduce en distintas tasas de degradación del conjugado y, por tanto, en distinta toxicidad (78, 79). Además, la ruta puede ser independiente de la toxina, es el caso de algunos compuestos dirigidos frente al receptor de transferrina (80).
La eficacia de una inmunotoxina depende del antígeno de la superficie celular al que va dirigida. En primer lugar, para que el conjugado sea lo más específico posible, se eligen antígenos que tengan una expresión elevada en tumores y una expresión restringida en tejidos normales. Estos blancos deben ser accesibles al conjugado y no encontrarse en forma soluble o tratarse de moléculas minoritarias, con el fin de que se fije la máxima cantidad de inmunotoxina a las células blanco (75). En la eficacia de una inmunotoxina también influye su estabilidad y la capacidad para penetrar en el tumor (81). Los compuestos de menor tamaño acceden con mayor facilidad al interior del tumor, pero se eliminan fácilmente a través del riñón (75). Por otro lado, la vida media de las inmunotoxinas en el suero está muy relacionada con su tasa de eliminación por el sistema retículo-endotelial, la cual se ve favorecida por la presencia de carbohidratos en la proteína (75).
Recientemente, se han obtenido conjugados con diversas RIPs de tipo 1, tanto nativas como recombinantes. Como ejemplos se pueden citar entre otras saporin 6, momordin I, PAP, gelonin, RIP de cebada, bryodin, etc., (82-84). Entre las inmunotoxinas construidas podemos destacar las inmunotoxinas contra la neovasculatura tumoral que tienen la ventaja de atacar los vasos que nutren el tumor sólido en vez de las células tumorales. La destrucción de estos vasos promueve un efecto multiplicador destructivo que aumenta la eficacia de la inmunotoxina. Entre las RIPs utilizadas se encuentran ricina (85) y RIPs de tipo 2 del género Sambucus (86). Las inmunotoxinas también se han empleado en la terapia experimental contra el SIDA dirigiéndolas hacia diferentes regiones de la cubierta glicoproteínica del VIH o hacia antígenos de superficie (87, 88).
La nanoencapsulación de diversas RIPs en distintos nanovectores bioselectivos, permite aumentar la biodistribución de la toxina, alcanzando biofase con menor riesgo de efectos adversos incontrolados y facilitar la posología en pacientes oncológicos, mediante el control de la liberación. Aunque el mayor número de estudios con estos sistemas se centran en cáncer, es de destacar su uso en patologías infecciosas (SIDA) o autoinmunes. En este sentido, nuestro grupo de investigación ha desarrollado un vector oral para la optimización del tratamiento con corticoides de la enfermedad de Crohn, tanto en fase de brote como en periodos intermedios. Dicho sistema ha sido formulado en base a diversos polímeros bioadhesivos para aumentar su tiempo de residencia en la mucosa intestinal. Además, debido a su funcionalización con cadenas con actividad lectina (B), es capaz de dirigirse de manera específica a tejido inflamado mediante el reconocimiento específico a receptores celulares sobreexpresados en células afectadas (datos no publicados).
7. INVESTIGACIÓN FUTURA
La investigación sobre las RIPs ha recibido especial atención en los últimos años debido a su interés intrínseco como componentes de mecanismos biológicos desconocidos y también a su uso como partes tóxicas de conjugados e inmunotoxinas para la terapia dirigida de enfermedades importantes. Hay una serie de preguntas abiertas sobre el papel desempeñado por las RIPs en los organismos productores y sobre su uso para aplicaciones médicas. El papel recientemente descrito para PAP como un componente del sistema de lectura compleja de ARNm en plantas, junto con el papel intrínseco antipatógeno atribuido a las RIPs en los últimos tiempos añaden más interés aún a estas enzimas. La variación de las concentraciones de RIP en diferentes tejidos y la presencia de isoformas de RIP podría tener relación en el desarrollo de las plantas. En cuanto al mecanismo de acción molecular de las RIPs, la amplia especificidad de muchos RIP en ribosomas de mamíferos, plantas y bacterias plantea la cuestión de si las RIPs contienen dominios catalíticos y de unión específicos para los diferentes ribosomas, ARNm e incluso ADN. Dado que muchos RIP no inhiben la síntesis de proteínas en bacterias y en plantas, su clonación y expresión y la construcción de plantas transgénicas resistentes a hongos y virus facilitarán la elucidación de las relaciones estructura-función RIP. La investigación futura intentará abordar todas estas (y otras) preguntas relacionadas con la función biológica de las RIPs.
8. BIBLIOGRAFÍA
1. Abrams P, Blaivas JG, Stanto SL, Andersen JT. The standardization of terminology of lower urinary tract function. Scand J Urol Nephrol Suppl 1988; 114:5-19.
1. Jiménez A, Vázquez D. Plant and fungal protein and glycoprotein toxins inhibiting eukaryote protein synthesis. Annu Rev Microbiol 1985; 39: 649-72.
2. Girbes T, Ferreras JM, Arias FJ, et al. Description, distribution, activity and phylogenetic relationship of ribosome-inactivating proteins in plants, fungi and bacteria. Mini-Reviews Med Chem 2004; 4(5): 461-76.
3. Stirpe F. Ribosome-inactivating proteins: From toxins to useful proteins. Toxicon 2013; 67: 12-6.
4. Bolognesi A, Bortolotti M, Maiello S, et al. Ribosome-Inactivating Proteins from Plants: A Historical Overview. Molecules 2016; 21(12): art. 1627.
5. Fabbrini MS, Katayama M, Nakase I, et al. Plant Ribosome-Inactivating Proteins: Progresses, Challenges and Biotechnological Applications (and a Few Digressions). Toxins 2017; 9(10): art. 314.
6. Schrot J, Weng A, Melzig MF. Ribosome-Inactivating and Related Proteins. Toxins 2015; 7(5): 1556-15.
7. Akkouh O, Ng TB, Cheung RCF, et al. Biological activities of ribosome-inactivating proteins and their possible applications as antimicrobial, anticancer, and anti-pest agents and in neuroscience research. Appl Microbiol Biotecnol 2015; 99(23): 9847-63.
8. Jain M, Amera GM, Muthukumaran J, et al. Insights into biological role of plant defense proteins: A review. Biocatalysis Agric Biotechnol 2022; 40: art. 102293.
9. Lu JQ, Zhu ZN, Zheng YT, et al. Engineering of Ribosome-inactivating Proteins for Improving Pharmacological Properties. Toxins 2020; 12(3): art. 167.
10. Sharma A, Gupta S, Sharma NR, et al. Expanding role of ribosome-inactivating proteins: From toxins to therapeutics. IUBMB Life. Disponible en: (https://iubmb.onlinelibrary.wiley.com/doi/10.1002/iub.2675)
11. Dougherty K, Hudak KA. Phylogeny and domain architecture of plant ribosome inactivating proteins. Phytochemistry 2022; 202: 113337.
12. Montanaro L, Sperti S, Stirpe F. Inhibition by ricin of protein synthesis in vitro. Ribosomes as the target of the toxin. Biochem J 1973; 136(3): 677-83.
13. Girbés T, Citores L, Ferreras JM, et al. Isolation and partial characterization of nigrin b, a non-toxic novel type 2 ribosome-inactivating protein from the bark of Sambucus nigra L. Plant Mol Biol 1993; 22: 1181-6.
14. Girbés T, Citores L, Iglesias R, et al. Ebulin 1, a nontoxic novel type 2 ribosome-inactivating protein from Sambucus ebulus L. leaves. J Biol Chem 1993; 268(24): 18195-9.
15. Walsh TA, Morgan AE, Hey TD. Characterization and molecular cloning of a proenzyme form of a ribosome-inactivating protein from maize. Novel mechanism of proenzyme activation by proteolytic removal of a 2.8-kilodalton internal peptide segment. J Biol Chem 1991; 266(34): 23422-7.
16. Reinbothe S, Reinbothe C, Lehmann J, et al. JIP60, a methyl jasmonate-induced ribosome-inactivating protein involved in plant stress reactions. Proc Natl Acad Sci USA. 1994; 91(15): 7012-6.
17. Mishra V, Mishra R, Shamra RS. Ribosome inactivating proteins – An unfathomed biomolecule for developing multi-stress tolerant transgenic plants. Int J Biol Macromol 2022; 210: 107-22.
18. Van Damme EJ, Roy S, Barre A, et al. Elderberry (Sambucus nigra) bark contains two structurally different Neu5Ac(alpha2,6)Gal/GalNAc-binding type 2 ribosome-inactivating proteins. Eur J Biochem 1997; 245(3): 648-55.
19. Van Damme EJ, Barre A, Rougé P, et al. Isolation and molecular cloning of a novel type 2 ribosome-inactivating protein with an inactive B chain from elderberry (Sambucus nigra) bark. J Biol Chem 1997; 272(13): 8353-60.
20. de Benito FM, Citores L, Iglesias R, et al. Isolation and partial characterization of a novel and uncommon two-chain 64-kDa ribosome-inactivating protein from the bark of elder (Sambucus nigra L.). FEBS Lett 1997; 413(1): 85-91.
21. Barbieri L, Valbonesi P, Bondioli M, et al. Adenine glycosylase activity in mammalian tissues: an equivalent of ribosome-inactivating proteins. FEBS Lett 2001; 505(1): 196-7.
22. Ferreras JM, Barbieri L, Girbés T, et al. Distribution and properties of major ribosome-inactivating proteins (28 S rRNA N-glycosidases) of the plant Saponaria officinalis L. (Caryophyllaceae). Biochim Biophys Acta 1993; 1216(1): 31-42.
23. Zhu F, Zhou YK, Ji ZL, et al. The plant ribosome-inactivating proteins play important roles in defense against pathogens and insect pest attacks. Front Plant Sci 2018; 9: 146. Disponible en: https://www.frontiersin.org/articles/10.3389/fpls.2018.00146/full.
24. Stirpe F, Barbieri L, Gorini P, et al. Activities associated with the presence of ribosome-inactivating proteins increase in senescent and stressed leaves. FEBS Lett 1996; 382(3): 309-12.
25. Girbés T, de Torre C, Iglesias R, et al. RIP for viruses. Nature 1996, 379: 777-8.
26. Rippmann JF, Michalowski CB, Nelson DE, et al. Induction of a ribosome-inactivating protein upon environmental stress. Plant Mol Biol 1997 Dec; 35(6): 701-9.
27. Sandvig K, van Deurs B. Transport of protein toxins into cells: pathways used by ricin, cholera toxin and Shiga toxin. FEBS Lett 2002; 529(1): 49-53.
28. Chan YS, Ng TB. Shiga toxins: from structure and mechanism to applications. Appl Microbiol Biotechnol 2016; 100(4): 1597-610
29. Wang HX, Ng TB. Isolation of pleuturegin, a novel ribosome-inactivating protein from fresh sclerotia of the edible mushroom Pleurotus tuber-regium. Biochem Biophys Res Commun 2001; 288(3): 718-21.
30. Lam SK, Ng TB. Hypsin, a novel thermostable ribosome-inactivating protein with antifungal and antiproliferative activities from fruiting bodies of the edible mushroom Hypsizigus marmoreus. Biochem Biophys Res Commun 2001; 285(4): 1071-5.
31. Liu RS, Yang JH, Liu WY. Isolation and enzymatic characterization of lamjapin, the first ribosome-inactivating protein from cryptogamic algal plant (Laminaria japonica A). Eur J Biochem 2002; 269(19): 4746-52.
32. Svinth M, Steighardt J, Hernandez R, et al. Differences in cytotoxicity of native and engineered RIPs can be used to assess their ability to reach the cytoplasm. Biochem Biophys Res Commun 1998; 249(3): 637-42.
33. Pascal JM, Day PJ, Monzingo AF, et al. 2.8-Å crystal structure of a nontoxic type-II ribosome-inactivating protein, ebulin l. Proteins 2001; 43: 319-26.
34. de Benito FM, Iglesias R, Ferreras JM, et al. Constitutive and inducible type 1 ribosome-inactivating proteins (RIPs) in elderberry (Sambucus nigra L.). FEBS Lett 1998; 428(1-2): 75-9.
35. de Benito FM, Citores L, Iglesias R, et al. Ebulitins: a new family of type 1 ribosome-inactivating proteins (rRNA N-glycosidases) from leaves of Sambucus ebulus L. that coexist with the type 2 ribosome-inactivating protein ebulin 1. FEBS Lett 1995; 360(3): 299-302.
36. Ling J, Liu WY, Wang TP. Simultaneous existence of two types of ribosome-inactivating proteins in the seeds of Cinnamonum camphora–characterization of the enzymatic activities of these cytotoxic proteins. Biochim Biophys Acta 1995, 1252(1): 15-22.
37. Hao Q, Van Damme EJ, Hause B, et al. Iris bulbs express type 1 and type 2 ribosome-inactivating proteins with unusual properties. Plant Physiology 2001;1 25(2): 866-76.
38. Endo Y, Tsurugi K. The RNA N-glycosidase activity of ricin A-chain. The characteristics of the enzymatic activity of ricin A-chain with ribosomes and with rRNA. J Biol Chem 1988; 263(18): 8735-9.
39. Girbes T. Lectinas: herramientas para el tercer código biológico. Discurso de ingreso en la Real Academia Nacional de Farmacia como Académico de Número. Disponible en: https://ranf.com/wp-content/uploads/academicos/discursos/numero/girbes.pdf.
40. Endo Y, Mitsui K, Motizuki M, et al. The mechanism of action of ricin and related toxic lectins on eukaryotic ribosomes. The site and the characteristics of the modification in 28 S ribosomal RNA caused by the toxins. J Biol Chem 1987; 262(12): 5908-12.
41. Iglesias R, Escarmis C, Alegre C, et al. Fusidic acid-dependent ribosomal complexes protect Escherichia coli ribosomes from the action of the type 1 ribosome-inactivating protein crotin 2. FEBS Lett 1993; 318(2): 189-92.
42. Ferrari C, Barbieri L, Stirpe F. Effects of plant ribosome-inactivating proteins on ribosomes from Musca domestica. Comp Biochem Physiol 1991; 100B(2): 223-7.
43. Park SW, Stevens NM, Vivanco JM. Enzymatic Specificity of Three Ribosome-Inactivating Proteins against Fungal Ribosomes, and Correlation with Antifungal Activity. Planta 2002, 216(2): 227–34.
44. Girbés T, Barbieri L, Ferreras M, et al. Effects of ribosome-inactivating proteins on Escherichia coli and Agrobacterium tumefaciens translation systems. J Bacteriol 1993, 175(20): 6721-4.
45. Ferreras JM, Iglesias R, Barbieri L, et al. Effects and molecular action of ribosome-inactivating proteins on ribosomes from Streptomyces lividans. Biochim Biophys Acta 1995; 1243(1): 85-93.
46. Iglesias R, Arias FJ, Rojo MA, et al. Molecular action of the type 1 ribosome-inactivating protein saporin 5 on Vicia sativa ribosomes. FEBS Lett 1993; 325(3): 291-4.
47. Ferreras JM, Alegre C, Iglesias R, et al. Characterization of a cell-free translation system from Agrobacterium tumefaciens. Biochem Soc Trans 1992; 20(4): 314S.
48. Habuka N, Akiyama K, Tsuge H, et al. Expression and secretion of Mirabilis antiviral protein in Escherichia coli and its inhibition of in vitro eukaryotic and prokaryotic protein synthesis. J Biol Chem 1990; 265(19): 10988-92.
49. Hartley MR, Legname G, Osborn R, et al. Single-chain ribosome inactivating proteins from plants depurinate Escherichia coli 23S ribosomal RNA. FEBS Letters 1991; 290(1-2): 65-68.
50. Endo Y, Wool IG. The site of action of alpha-sarcin on eukaryotic ribosomes. The sequence at the alpha-sarcin cleavage site in 28S ribosomal ribonucleic acid. J Biol Chem 1982; 257(15): 9054-60.
51. López-Otín C, Barber D, Fernández-Luna JL, et al. The primary structure of the cytotoxin restrictocin. Eur J Biochem 1984; 143(3): 621-34.
52. Barbieri L, Ferreras JM, Barraco A, et al. Some ribosome-inactivating proteins depurinate ribosomal RNA at multiple sites. Biochem J 1992; 286: 1-4.
53. Sawasaki T, Morishita R, Ozawa A, et al. Mechanism of ribosome RNA apurinic site specific lyase. Nucleic Acids Symp Ser 1999; 42: 257-8.
54. Ogasawara T, Sawasaki T, Morishita R, et al. A new class of enzyme acting on damaged ribosomes: ribosomal RNA apurinic site specific lyase found in wheat germ. The EMBO Journal 1999; 18(22): 6522-31.
55. Ito Y, Ozawa A, Sawasaki T, et al. OsRALyase1, a putative F-box protein identified in rice, Oryza sativa, with enzyme activity identical to that of wheat RALyase. Biosci Biotech Biochem 2002; 66(12): 2727-31.
56. Pelosi E, Lubelli C, Polito L, et al. Ribosome-inactivating proteins and other lectins from Adenia (Passifloraceae). Toxicon 2005; 46(6): 658-63.
57. Barbieri L, Gorini P, Valbonesi P, et al. Unexpected activity of saporins. Nature 1994; 372(6507):624.
58. Brigotti M, Alfieri R, Sestili P, et al. Damage to nuclear DNA induced by Shiga toxin 1 and ricin in human endothelial cells1. The FASEB Journal 2022; 16: 365-72.
59. Hudak KA, Bauman JD, Tumer NE. Pokeweed antiviral protein binds to the cap structure of eukaryotic mRNA and depurinates the mRNA downstream of the cap. RNA. 2002; 8(9): 1148-59.
60. Parikh BA, Coetzer C, Tumer NE. Pokeweed antiviral protein regulates the stability of its own mRNA by a mechanism that requires depurination but can be separated from depurination of the alpha-sarcin/ricin loop of rRNA. J Biol Chem 2002; 277(44): 41428-37.
61. Day PJ, Pinheiro TJ, Roberts LM, et al. Binding of ricin A-chain to negatively charged phospholipid vesicles leads to protein structural changes and destabilizes the lipid bilayer. Biochemistry 2002; 41(8): 2836-43.
62. Zhang GP, Shi YL, Wang WP, et al. Cation channel formed at lipid bilayer by Cinnamomin, a new type II ribosome-inactivating protein. Toxicon 1999; 37(9): 1313-22.
63. Lombard S, Helmy ME, Piéroni G. Lipolytic activity of ricin from Ricinus sanguineus and Ricinus communis on neutral lipids. Biochem J 2001; 358: 773-81.
64. Morlon-Guyot J, Helmy M, Lombard-Frasca S, et al. Identification of the ricin lipase site and implication in cytotoxicity. J Biol Chem 2003; 278(19): 17006-11.
65. Remi Shih N, McDonald K, Jackman A, et al. Bifunctional plant defence enzymes with chitinase and ribosome inactivating activities from Trichosanthes kirilowii cell cultures. Plant Sci 1997; 130(2): 145-50.
66. Barbieri L, Brigotti M, Perocco P, et al. Ribosome-inactivating proteins depurinate poly(ADP-ribosyl)ated poly(ADP-ribose) polymerase and have transforming activity for 3T3 fibroblasts. FEBS Letters 2003; 538(1-3): 178-82.
67. Lee-Huang S, Kung HF, Huang PL, et al. A new class of anti-HIV agents: GAP31, DAPs 30 and 32. FEBS Lett 1991; 291(1): 139-44.
68. Lee-Huang S, Huang PL, Kung HF, et al. TAP 29: an anti-human immunodeficiency virus protein from Trichosanthes kirilowii that is nontoxic to intact cells. Proc Natl Acad Sci USA 1991; 88(15): 6570-4.
69. Lyu SY, Choi SH, Park WB. Korean mistletoe lectin-induced apoptosis in hepatocarcinoma cells is associated with inhibition of telomerase via mitochondrial controlled pathway independent of p53. Arch Pharm Res 2002; 25: 93-101.
70. Li XD, Chen WF, Liu WY, et al. Large-scale preparation of two new ribosome-inactivating proteins – Cinnamomin and camphorin from the seeds of Cinnamomum camphora. Prot Express Purific 1997; 10(1): 27-31.
71. Gilabert-Oriol R, Weng A, von Mallinckrodt B, et al. Immunotoxins Constructed with Ribosome-Inactivating Proteins and their Enhancers: A Lethal Cocktail with Tumor Specific Efficacy. Current Pharmaceutical Design 2014; 20(42): 6584-43.
72. Pizzo E, di Maro A. A new age for biomedical applications of Ribosome Inactivating Proteins (RIPs): from bioconjugate to nanoconstructs. J Biomed Sci 2016; 23: art 54.
73. Temaj G, Chichiarelli S, Eufemi M, et al. Ribosome-Directed Therapies in Cancer. Biomedicines 2022; 10; 2088.
74. Flavell DJ, Flavell SU. Plant-Derived Type I Ribosome Inactivating Protein-Based Targeted Toxins: A Review of the Clinical Experience 2022; 14; art. 563.
75. Brinkmann U, Pastan I. Immunotoxins against cancer. Biochim Biophys Acta 1994; 1198(1): 27-45.
76. Mahmoudi R, Dianat-Moghadam H, Poorebrahim M, et al. Recombinant immunotoxins development for HER2-based targeted cancer therapies. Cancer Cell Int 2021; 21: 470. art. 470.
77. Chen YZ, Zhang M, Min KA, et al. Improved Protein Toxin Delivery Based on ATTEMPTS Systems. J Drug Targ 2018; 19(4); 380-92.
78. Battelli MG, Citores L, Buonamici L, et al. Toxicity and cytotoxicity of nigrin b, a two-chain ribosome-inactivating protein from Sambucus nigra: comparison with ricin. Arch Toxicol 1997; 71(6): 360-4.
79. Muñoz R, Arias Y, Ferreras JM, et al. Sensitivity of cancer cell lines to the novel non-toxic type 2 ribosome-inactivating protein nigrin b, Cancer Letters 2001; 167(2): 163-9.
80. Citores L, Ferreras JM, Muñoz R, et al. Targeting cancer cells with transferrin conjugates containing the non-toxic type 2 ribosome-inactivating proteins nigrin b or ebulin l. Cancer Letters 2002; 184(1): 29-35.
81. Rostaing-Capaillon O, Casellas P. In vivo cytotoxic efficacy of immunotoxins prepared from anti-CD5 antibody linked to ricin A-chain. Cancer Immunol Immunother 1991; 34(1): 24-30.
82. Filpula D. Antibody engineering and modification technologies. Biomol Eng 2007; 24(2): 201-15.
83. Khirehgesh MR, Sharifi J, Safari F, et al. Immunotoxins and nanobody-based immunotoxins: review and update. J Drug Targ 2021; 29(8), 848-62.
84. Kreitman RJ, Pastan I. Immunotoxins: From Design to Clinical Application. Biomolecules 2021; 11(11): 1696
85. Seon BK, Matsuno F, Haruta Y, et al. Long-lasting complete inhibition of human solid tumors in SCID mice by targeting endothelial cells of tumor vasculature with antihuman endoglin immunotoxin. Clin Cancer Res 1997; 3(7): 1031-44.
86. Ferreras JM, Citores L, Iglesias R, et al. Use of Ribosome-Inactivating Proteins from Sambucus for the Construction of Immunotoxins and Conjugates for Cancer Therapy. Toxins 2011; 3(5): 420-4.
87. Zarling JM, Moran PA, Haffar O, et al. Inhibition of HIV replication by pokeweed antiviral protein targeted to CD4+ cells by monoclonal-antibodies. Nature 1990; 347(6288): 92-95.
88. Pincus SH, Song K, Maresh GA, et al. Identification of Human Anti-HIV gp160 Monoclonal Antibodies That Make Effective Immunotoxins. J Virol 2017; 91(3). art. 01955.