1. INTRODUCCIÓN
El conocimiento de Los β-lactámicos surge con el descubrimiento de la penicilina por Alexander Fleming en 1929, cuándo varias placas que contenían colonias de Staphylococcus se encontraban contaminadas por un moho, actualmente caracterizado cómo Penicillium notatum (Alexander Fleming 1929). A pesar de los esfuerzos realizados por Fleming para conseguir concienciar al mundo científico de la necesidad de aislar el compuesto no es hasta 1940 cuando Howard Florey y Ernest Chain publican un método para poder purificar la penicilina de manera eficaz para ser utilizada en medicina clínica (Aminov 2010). Este suceso marcó un antes y un después en el desarrollo de la humanidad. A partir de este momento, desde un punto de vista científico, numerosos grupos se interesan por los β-lactámicos, intentando entender su mecanismo de acción para poder realizar modificaciones que mejoraran su actividad terapéutica. Por otro lado, a nivel médico, se comienzan a utilizar este grupo de antibióticos como una herramienta eficaz en la lucha contra las enfermedades microbianas, destacando el papel sanitario que tuvieron durante la 2ª Guerra Mundial (Luepke, Suda et al. 2017).
Actualmente estos compuestos son muy utilizados debido a diferentes características antibióticas cómo su heterogeneidad, su amplio espectro y su gran poder bactericida. Tanto es así que, según datos de la OMS, el 63,8% de todos los antibióticos que se consumieron cada día en España durante 2015 fueron β-lactámicos (WHO 2018). Históricamente, este tipo de antibióticos se han dividido en cinco subgrupos diferentes: penicilinas, cefalosporinas, carbapenémicos, monobactámicos e inhibidores de las β-lactamasas. Todos ellos se caracterizan por tener una estructura química compuesta por una amida cíclica muy reactiva, a la que se denomina anillo β-lactámico, que sufre diferentes modificaciones para dar lugar a los distintos subgrupos mencionados anteriormente (Fernandes, Amador et al. 2013).
El mecanismo de acción de la mayoría de los antibióticos β-lactámicos consiste en la inhibición de la formación de la pared celular bacteriana. En concreto, impiden la correcta formación del peptidoglucano, componente esencial del espacio periplasmático que confiere rigidez a la célula a la vez que la protege del estrés osmótico interior (Ur Rahman, Ali et al. 2018). Esta molécula se forma mediante el entrecruzamiento de largas cadenas de glúcidos (N-acetilmurámico, NAM, y N-acetilglucosamina, NAG), también conocidas como cadenas de mureína, entrelazadas mediante pentapéptidos, conformando una estructura en forma de malla. Cabe destacar que los pentapéptidos se encuentran unidos al glúcido NAM y se caracterizan por ser estructuralmente diferentes entre las especies bacterianas. La estructura general del péptido se caracteriza por tener una Alanina en 1ª posición, exceptuando algunas especies que en su lugar contienen Glicina o Serina. En 2ª posición una Glutamina común para todas las bacterias. La 3ª posición que conforma el péptido es la más variada y dependiendo de la especie se pueden identificar un tipo u otro de aminoácidos, en general, las gram negativas se caracterizan por tener m-DAP (ácido meso-diaminopimelico), mientras que las gram positivas suelen tener D-Lisina. Los aminoácidos en la 4ª y 5ª posición generalmente son dos D-Alanina y suelen añadirse de forma conjunta en forma de dímero que se une al tripéptido (Vollmer, Blanot et al. 2008).
El último paso esencial en la formación de un peptidoglucano estable y funcional es el proceso denominado “cross-linking”, donde las cadenas de glúcidos se unen unas a otras dando lugar a una conformación biológicamente funcional. En este paso, es necesario que unas transpeptidasas y carboxypeptidasas, pertenecientes al grupo de proteínas PBPs (penicillin-binding proteins), se encarguen de realizar la unión de dos cadenas de mureína (Zapun, Contreras-Martel et al. 2008). En el caso de las bacterias gram-negativas, estas PBPs conectan dos cadenas adyacentes realizando un enlace peptídico entre la penúltima D-Alanina del péptido de una de las cadenas y el m-DAP del péptido adyacente, a la vez que una D-Alanina carboxypeptidasa se encarga de eliminar el último aminoácido del primer pentapéptido (Figura 1). Asimismo, en gram-positivas, ocurre un proceso similar, en el que, en vez de un enlace peptídico, se forma un puente peptídico (formado por 5 glicinas) entre la penúltima D-Alanina de una cadena y el 3ª aminoácido perteneciente al pentapéptido de la cadena adyacente. En conclusión, se crea un peptidoglucano estable debido a la unión de dos cadenas de mureína a nivel de sus pentapéptidos (Pazos, Peters 2019; Kong, Schneper et al. 2010).
Figura 1: Formación de la pared celular mediante la actuación de una PBP (izquierda). Inhibición de la formación del peptidoglucano por la acción de distintos tipos de β-lactámicos que se unen al sitio activo de la enzima mimetizando los dos últimos aminoácidos (D-Ala-D-Ala) del pentapéptido.(derecha). Figura Modificada de Usman Umar Zango, Munir Ibrahim et al. 2019.
Según la teoría más aceptada que Tipper y Strominger desarrollaron en 1965, los β-lactámicos, en especial la penicilina, forman una estructura tridimensional muy similar a la conformación que adoptan los aminoácidos terminales (D-Ala-D-Ala) del pentapéptido, pudiendo ser esta la razón de la inhibición de las PBPs (Donald J Tipper, Jack L. Strominger 1965). Investigaciones más recientes demuestra que la inhibición de distintas PBPs puede dar lugar a diferentes situaciones dentro de la célula, como: la lisis celular por la activación de unas proteínas denominadas autolisinas que se encargan de destruir el peptidoglucano mal formad; La inhibición de la división celular, debido a la inestabilidad periplásmica; la filamentación de la célula (protuberancias que aparecen en la célula e impiden su correcto funcionamiento; Bush, Karen, Bradford 2016).
Además, dentro del amplio grupo de los β-lactámicos, existe un subgrupo denominado “inhibidores de las β-lactamasas”. Este se compone de una serie de moléculas con estructura similar a los β-lactámicos, pero cuya función, en vez de ser antimicrobiana es la de inhibir el funcionamiento de las enzimas bacterianas (mediante una unión no competitiva al sitio activo) que se encargan de desactivar los antibióticos β-lactámicos. Estas enzimas son las β-lactamasas (Neelanjana Pandey, Marco Cascella 2019).
Las β-lactamasas son enzimas capaces de inactivar los antibióticos β-lactámicos mediante dos tipos diferentes de acción hidrolítica. El primer gran grupo son las β-lactamasas con serina que hidrolizan el anillo β-lactámico y que contienen una secuencia de aminoácidos muy variada entre ellas, pero un centro catalítico muy conservado con una serina responsable de la catálisis (Parveen Salahuddin, Amit Kumar et al. 2018). El segundo grupo se caracteriza por catalizar la reacción de hidrólisis mediante la utilización de iones de zinc que se encuentran en los sitios activos de estas enzimas, y que por su mecanismo de acción se denominan MBLs (Zhang, Hao 2011; del inglés metallo-betalactamases).
Estos dos grandes grupos, se dividen en 4 clases diferentes de β-lactamasas (A, B, C y D) según la clasificación de Ambler (R. P. Ambler 1980). Esta clasificación se basa en la homología o similitud de las secuencias de aminoácidos, donde las enzimas de los grupos A, C y D son β-lactamasas que presentan una serina catalítica, mientras que las del grupo B utilizan iones metálicos para realizar la catálisis (Melise Chaves Silveira, Rangeline Azevedo da Silva et al. 2018). Sin embargo, la clasificación de las β-lactamasas más utilizada en el diagnostico de infecciones es la de Bush-Jacoby-Medeiros (Bush, K., Jacoby et al. 1995). El uso popular en medicina clínica de esta clasificación se debe a los criterios utilizados en la misma para identificar cada proteína: Primero, las β-lactamasas se agrupan dependiendo de que agente es el que produce su inhibición (ácido etilendiaminotetraacético, el ácido clavulánico, etc.) y, en segundo lugar, se agrupan de acuerdo con su capacidad y eficacia a la hora de hidrolizar distintos compuestos β-lactámicos (David L. Paterson, Robert A. Bonomo 2005).
La diversificación de las β-lactamasas es un evento reciente que surge como un mecanismo de defensa bacteriano ante la alta presión terapéutica ejercida por el uso popular de los β-lactámicos sintéticos o semi-sintéticos en diversos sectores como la medicina, la ganadería, la cosmética o la agricultura. Sin embargo, se han encontrado genes de resistencia en sedimentos de permafrost con más de 30,000 años (Crofts, Gasparrini et al. 2017) y se estima el origen de las β-lactamasas en más de 2000 millones de años (Risso, Gavira et al. 2013).
En 2018 se habían descrito más de 2.770 β-lactamasas. A lo largo de los últimos 20 años, desde el año 2000 hasta 2018, el número de nuevas β-lactamasas identificadas ha aumentado un 500% con respecto a las que se habían caracterizado en el periodo anterior desde el descubrimiento de la penicilina en 1929 (Karen Bush 2018). En todos estos estudios se destaca que las β-lactamasas son el principal mecanismo de resistencia que presentan las bacterias frente a los antibióticos β-lactámicos.
En relación con este tema, cabe resaltar que, en Europa, el coste total debido al aumento de la resistencia a los antibióticos, en términos de hospitalización y pérdida de producción laboral, se estima en 1,500 millones de euros cada año (Karen Bush, Patrice Courvalin et al. 2011). Además, la OMS publicó en 2019 un informe realizado por la Interagency coordination group en el que se estima que para 2030, los costes debido a la resistencia microbiana podrían llevar a 24 millones de personas a la pobreza extrema en el mundo. Por otro lado, la resistencia a antibióticos no produce solamente un problema económico, sino que la OMS resalta que en 2019 hubo 700.000 muertes debido a enfermedades infecciosas resistentes y estima que para 2050 la resistencia microbiana a antibióticos provocará 10 millones de muertes cada año (IACG 2019). Por ende, se hace incuestionable la necesidad de estudiar estas proteínas para poder luchar contra ellas y así disminuir este coste sanitario y económico. Más aún, cuando se conoce que la aparición de resistencia a un nuevo antibiótico ocurre a los 3 años de su aprobación por los organismos reguladores (Barlow, Hall 2003). Siendo esta una de las principales razones por las que la industria farmacéutica solamente consiguió sacar al mercado 12 nuevos antibióticos en el periodo comprendido entre 1999 y 2014 (Kållberg, Årdal et al. 2018).
Una de las posibles soluciones planteadas ante esta situación de emergencia ha sido la reestructuración y limitación del uso de estos fármacos para intentar frenar el aumento de resistencias, sin embargo, se ha demostrado que aunque se reduzca el número de prescripciones de antibióticos, no se consigue reducir la resistencia, sino que en algunos casos incluso se puede aumentar, como fue el aumento de bacterias nosocomiales súper resistentes, debido a su uso necesario en un lugares muy concretos y de manera continuada (Livermore 2004). Por lo tanto, parece necesario comprender las características biológicas de las β-lactamasas, desde el origen de su promiscuidad hasta su alta capacidad para evolucionar ante el estrés, para poder prever que tipo de mutaciones sufrirán los genes de resistencia ante el uso continuo de un fármaco o ante los nuevos fármacos que pudieran salir al mercado (Hall 2004).
En este sentido, el grupo del profesor Barry G. Hall ha descrito un modelo de evolución dirigida de proteínas que mimetiza la evolución natural en el laboratorio utilizando como objeto de estudio el gen TEM-1 que codifica para una β-lactamasa que hidroliza penicilinas y algunas cefalosporinas(Barlow, Hall 2003b). Una de las conclusiones de este estudio resaltaba que a la hora de predecir futuras mutaciones de un linaje uno de los métodos más efectivos es mimetizar la evolución natural mediante una evolución in vitro de proteínas ancestrales reconstruidas.
La reconstrucción ancestral de proteínas (Ancestral Sequence Reconstruction, o ASR), es una técnica basada en la obtención de proteínas del pasado utilizando una combinación de herramientas filogenéticas y de biología molecular. Su uso principal es poder estudiar en el laboratorio la evolución natural de una familia de proteínas y deducir su futuro linaje a través del análisis y detección de los cambios estructurales que ha tenido que sufrir un gen para dar lugar a sus homólogos actuales (Thornton 2004). El estudio y caracterización de estas proteínas ha permitido, por ejemplo, comprender características como la relación existente entre la secuencia de aminoácidos de una proteína y su posible función, así como descifrar posibles características proteicas (estabilidad a altas temperaturas, promiscuidad, etc.) inferidas por el estrés medioambiental de hace millones de años (Gumulya, Gillam 2017).
El proceso de obtención de una proteína ancestral consta de una serie de pasos:
a) Hacer un alineamiento de múltiples β-lactamasas actuales homólogas a partir de las cuales quiera inferirse su secuencia ancestral. Estas secuencias se obtienen de bases de datos como UniProt o NCBI; b) Estimar la filogenia de estas secuencias mediante algún método de inferencia estadístico como la máxima parsimonia, la máxima verosimilitud o la inferencia bayesiana (MP, ML e IB, respectivamente); c) generar la secuencia más probable para cada nodo del árbol filogenético; d) Producir y expresar la proteína codificada por la secuencia ancestral seleccionada (Gumulya, Gillam 2017). Todos los pasos desde el “a)” hasta el “c)” precisan el uso de distintas herramientas computacionales. Actualmente, existen diversos programas gratuitos con los que poder realizar todo el proceso de reconstrucción ancestral de proteínas de manera eficaz (Arenas, Weber et al. 2017).
Esta técnica científica ya se ha utilizado para el estudio y la mejora de proteínas actuales. Se pueden encontrar diversos ejemplos como puede ser la utilización de la reconstrucción ancestral de la proteína ancestral de factor VIII de coagulación, para producir una proteína con más estabilidad, menos inmunogénica y con una mejora de la actividad funcional (Zakas, Brown et al. 2017); o la obtención, mediante ASR de mutantes de la enzima RUBISCO mucho más eficientes a la hora de fijar el CO2 (Gómez-Fernández, García-Ruiz et al. 2018).
Con el fin de profundizar en el conocimiento de la acción y el origen de actividad hidrolítica de las β-lactamasas de clase A (atendiendo a la nomenclatura de Ambler) en este trabajo hemos caracterizado el perfil de resistencia de 4 β-lactamasas de espectro extendido y de un ancestro común reconstruido por ASR frente a 8 antibióticos β-lactámicos de uso clínico.
2. MATERIALES Y MÉTODOS
2.1 Reactivos
A partir de bacterias E. coli JM109 (endA1 glnV44 thi-1 relA1 gyrA96 recA1mcrB+Δ(lac–proAB)e14- [F’ traD36 proAB+ lacIq lacZΔM15] hsdR17(rK-mK+), cedidas por la Dra. María Isabel Martínez Jiménez (CBMSO); y E. coli DH5E (F- φ80dlacZΔM15 Δ(lacZYA-argF) U169 endA1 recA1 hsdR17(r– m+) deoR thi-1 phoA supE44 λ– gyrA96 relA1 gal–; ThermoFisher Scientific) se obtuvieron células electrocompetentes utilizando un protocolo estandarizado (Sambrook, Green 2012). El vector pACSE3 fue cedido por el Dr. Barry Hall (Barlow, Hall 2002). Los compuestos Aztreonam (ATM), Cefotaxima (CTX), Cefepima (FEP), Cefuroxima (CXM), Ceftazidima (CAZ), Ceftriaxona (CRO) e Imipenem (IMP) se obtuvieron de la empresa MedChemExpress.
El medio de cultivo en polvo LB, ampicilina (AMP), tetraciclina (TET), glicerol y el isopropil-β-D-1- tiogalactopiranósido (IPTG) se consiguieron a través de la empresa AppliChem. La solución stock de cada uno de los antibióticos se realizó utilizando 0.1M de tampón NaPO4 a pH 7.0 y se almacenó a -80 ºC en estas concentraciones: AMP, CTX y CXM a 128 g/L; TET a 15 g/L; FEP e IMP a 8 g/L; ATM, CAZ y CRO a 4 g/L. Las enzimas de restricción SacI y BspHI, la DNA polimerasa Phusion® High-Fidelity (HF), la ligasa T4 y la enzima fosfatasa alcalina (CIP proveniente del inglés “Calf intestine phosphate”) se adquirieron de la empresa New England Biolabs. El SYBR® Green II fluorescent dye, el medio de cultivo en polvo Müler-Hinton y los oligonucleótidos utilizados se compraron a la casa comercial µ-Aldrich. Los kits de purificación de ADN (NucleoSpin™ Gel and PCR Clean-up Kit) y de aislamiento de plásmido se consiguieron a través de la empresa Macherey-Nagel.
2.2 Obtención y selección de secuencias de β-lactamasas ancestrales
2.2.1 Selección de secuencias de β-lactamasas actuales
Para este trabajo, se decidió estudiar la actividad hidrolítica de 4 enzimas actuales [caracterizadas por ser β-lactamasas de espectro extendido (ESBLs del inglés extended expectrum β-lactamases) y carbapenemasas]. En concreto se escogieron cuatro β- lactamasas, que de acuerdo con la clasificación de Ambler son: Una β-lactamasa de clase A capaz de hidrolizar el antibiótico β-lactamámico carbapenem, denominada “SME-1” proveniente de Serratia marcescens S6 (Thierry Naas, Laurence Vandel et al. 1994); Una ESBL de clase A denominada “KLUG-1” ó “imiA” proveniente de Kluyvera georgiana (Poirel, Kampfer et al. 2002); Una carbapenemasa de clase A denominada “BKC-1” que procede de Klebsiella pneumoniae (Nicoletti, Marcondes et al. 2015); Y otra ESBL de clase A capaz de hidrolizar carbapenem denominada “KPC-1” proveniente de una cepa resistente, Klebsiella pneumoniae 1534 (Yigit, Queenan et al. 2001).
2.2.2 Obtención de secuencias ancestrales
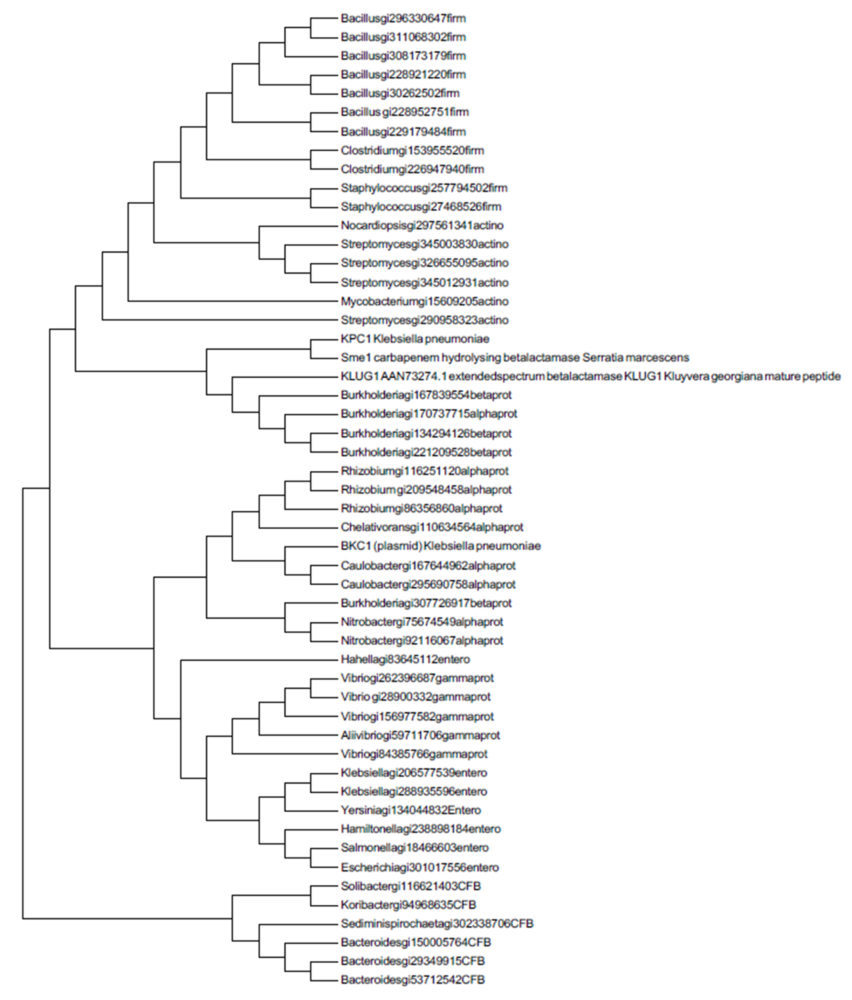
Para la selección de las secuencias ancestrales que han sido sintetizadas en el proyecto se contó con la colaboración del Dr. Barry G. Hall (Bellingham Research Institute), experto a nivel mundial en el estudio de la evolución experimental de β-lactamasas. Estas secuencias se obtuvieron a partir de 2 alineamientos: Uno construido a partir de las secuencias peptídicas de 48 β-lactamasas actuales utilizadas previamente en el trabajo realizado por Risso et al. en 2014 en combinación con las secuencias de las 4 enzimas nombradas en el apartado anterior, y otro alineamiento construido a partir de 16 secuencias de β-lactamasas de clase A e incluyendo las 4 proteínas actuales escogidas para el estudio, que pueden tener capacidad de hidrolizar carbapenémicos. Cabe destacar, que este segundo alineamiento se realizó para obtener un modelo más concreto de la filogenia de las β-lactamasas de clase A y su evolución hacia la resistencia hacia antibióticos carbapenémicos. La herramienta informática utilizada para alinear las secuencias fue el programa gratuito Guidance 2.
A partir de este alineamiento, se estimó la filogenia de estas secuencias mediante el método de máxima verosimilitud ML utilizando el programa informático gratuito MEGA. Finalmente, la obtención de las secuencias de ADN y proteínas más probables correspondientes a cada uno de los nodos del árbol filogenético se obtuvieron mediante la herramienta informática diseñada por el Dr. Barry G. Hall denominada ExtAnceSeqProt.
Tras obtener las secuencias ancestrales, el Dr. Barry G. Hall seleccionó aquellas situadas en los nodos que correspondían a los ancestros más comunes recientes (MRCA de sus siglas en inglés). En el caso de la filogenia construida con las 48 secuencias provenientes del trabajo de Risso et al., se seleccionaron los MRCAs de las Betaproteobacteria, Alphaproteobacteria, Proteobacteria (gram negativas): nodos 75, 92 y 90, respectivamente. En el caso de la filogenia obtenida utilizando el conjunto de 16 β-lactamasasde clase A, se seleccionó el nodo perteneciente al MRCA más antiguo: nodo 26. (Anexo ML-phylo). Finalmente, se seleccionó un MRCA de β-lactamasas pertenecientes a bacterias gram negativas denominado GNCA (del inglés Gram negative Common Ancestor) que había sido caracterizado anteriormente (Risso, Gavira et al. 2013) para ser utilizado como control.
2.2.3 Síntesis de secuencias ancestrales y actuales
Las secuencias pertenecientes a los nodos 75, 90, 92 y 26 (denominadas Anc 75, Anc 90, Anc 92 y Anc 26, respectivamente), así como la secuencia ancestral GNCA y la secuencia del gen actual BKC-1 se obtuvieron mediante síntesis química (Eurofins Genomics) los genes actuales KLUG-1 (imi A), KPC-1 y SME-1 se obtuvieron mediante amplificación por PCR a partir de muestras obtenidas del ADN bacteriano correspondiente. Este material genético se extrajo mediante el método fenol/cloroformo (Sambrook, Green 2012) a partir de las bacterias Kluyvera georgiana y Serratia marcescens S6, cedidas por el Dr. Patrice Nordmann, y Klebsiella pneumoniae, cedida por la Dra. Ana Cristina Galesa.
2.3 Clonaje de las secuencias ancestrales y actuales en el vector pACSE3
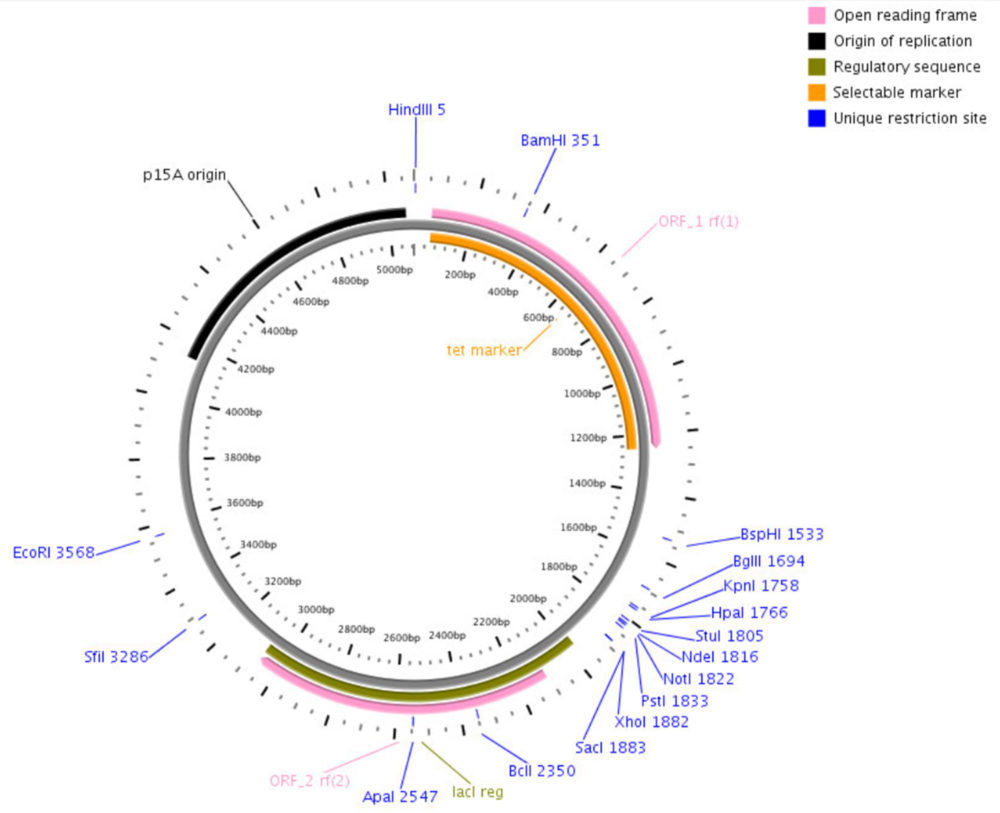
Todos los genes codificantes de β-lactamasas utilizados en este trabajo se clonaron dentro del vector pACSE3 (Figura 2).
Figura 2: Plásmido pACSE3 utilizado para el clonaje de los genes utilizados en este estudio. Se muestra muestra el sitio de policlonaje con las dianas de restricción utilizadas (Bsp HI y Sac I), el gen de resistencia Tet (en naranja) y la región lac (en verde).
Tanto los genes obtenidos por síntesis química, como el ADN purificado de las bacterias Kluyvera georgiana, Klebsiella pneumoniae y Serratia marcescens S6 se amplificaron por PCR utilizando parejas de iniciadores específicos (Tabla A1). durante esta amplificación, se introdujeron por mutagénesis dirigida las dianas de las enzimas de restricción SacI y BspHI en los extremos 5’ y 3’, respectivamente, de los genes de interés. Los amplicones obtenidos se purificaron con el kit NucleoSpin™ Gel and PCR Clean-up y se digirieron utilizando las enzimas de restricción SacI y BspHI durante 1 h a 37 ºC para obtener el ADN utilizado como inserto. Todo el producto de digestión se cargó en un gel de agarosa (1% p/v), y tras electroforesis se aisló del gel mediante el kit. Por otro lado, el vector pACSE3 purificado mediante el kit NucleoSpin™ Plasmid Miniprep, se digirió utilizando las mismas enzimas de restricción en presencia de CIP para eliminar los fosfatos de los extremos 5’de las cadenas de ADN.
Una vez realizada la digestión tanto el vector como los como insertos se purificaron y se ligaron (utilizando una relación molar entre el vector y el inserto de 1:7) con ligasa T4 durante 1 h a 25 ºC. Tras la ligación, se procedió a la purificación utilizando el kit correspondiente.
Finalmente, 2 μL del producto purificado de la ligación se transformaron en 50μL de bacterias E. Coli DH5E electrocompetentes utilizando un electroporador Gene Pulser® II Electroporation System siguiendo las indicaciones del fabricante. Este proceso se realizó con cada una de las construcciones de interés, obteniendo así 9 transformantes diferentes que contenían en su interior el plásmido con el gen de interés. Estas bacterias se multiplicaron y una alícuota de cada una de ellas se conservaron a -80 ºC en presencia glicerol al 20% para su posterior uso. Asimismo, se comprobó mediante secuenciación la correcta estructura de cada una de las construcciones.
2.4 Puesta a punto del método para medir la funcionalidad de las β-lactamasas seleccionadas
Para medir la actividad de las β-lactamasas estudiadas en este trabajo, se decidió utilizar el método de la mínima concentración inhibitoria (MICs del inglés Minimal inhibitory concentrations), utilizado históricamente para la detección de resistencia de una bacteria ante un antibiótico (Christiane Bébéar, Janet A. Robertson 1996). Se utilizó una placa de 96 pocillos (M-96, 8 filas por 12 columnas) como soporte para medir la resistencia de cada uno de las β-lactamasa los 8 antibióticos utilizados en este estudio: ampicilina, aztreonam, cefotaxima, ceftazidima, ceftriaxona, cefepimac, cefuroxima e imipenem. Se añadieron, en cada una de las 8 filas de la placa, diferentes concentraciones de cada compuesto. Desde la columna 12 a la columna 2, la concentración de antibiótico en cada uno de los pocillos fue la mitad de la del pocillo anterior. De este modo, el segundo pocillo de cada fila contendrá la mínima concentración de antibiótico y el último la máxima concentración de antibiótico. El primer pocillo de cada fila se mantuvo libre de antibiótico y se utilizó como control negativo del efecto inhibitorio de estos compuestos. Las concentraciones de antibiótico utilizadas en cada placa se indican en la sección correspondiente de Resultados. Después de incubar la placa durante 20 horas a 30 ºC, se midieron la densidad óptica (O.D.) de cada uno de los pocillos utilizando un espectrofotómetro lector de placas FLUOstar® Omega (BMG labtech) a una longitud de onda de 600 nm.
3. RESULTADOS
3.1. Puesta a punto del método para el análisis de las secuencias ancestrales
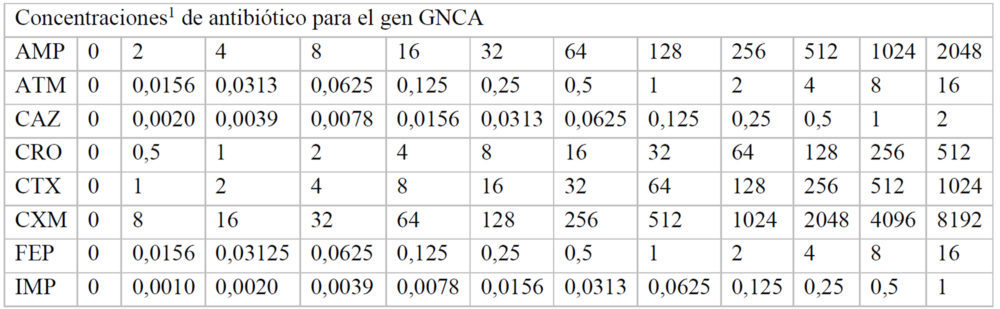
El objetivo de este trabajo es medir y comparar la actividad enzimática de varias β-lactamasas frente a un grupo de antibióticos. Asimismo, se decidió medir la concentración mínima de antibiótico que era capaz de inhibir el crecimiento de cultivos de E. coli que expresaban los genes recombinantes de β-lactamasas. De modo que en una única placa M-96 se analiza el efecto β-lactámico de una única enzima frente a 8 antibióticos diferentes. Para ello, se crece un cultivo de bacterias transformadas con plásmidos que portaban el gen de interés en 10 mL medio Müller-Hinton (M-H) durante toda la noche a 37 ºC en presencia de 0.1 mM IPTG y 10 μg/mL de TET. Esta incubación se realiza en tubos de 15 mL y cerrados para limitar la aireación del cultivo. Al día siguiente, se diluye el cultivo para que contenga una concentración de 5×105 unidades formadoras de colonia (CFU) por mL. Se añaden 200 μL de esta disolución en cada uno de los pocillos de una placa M-96 y 500 μL en 8 tubos eppendorf. En cada tubo eppendorf se diluye cada uno de los antibióticos utilizados en el estudio. La concentración de cada antibiótico en el tubo es igual al doble de la concentración máxima utilizada en el ensayo (Tablas A2-A8). En el pocillo duodécimo de cada fila de la placa M-96 se añaden 200 μL del tubo eppendorf que contiene el antibiótico correspondiente (Tabla 1). A partir del último pocillo de cada fila de la M96 se realizaron diluciones seriadas añadiendo a cada pocillo 200 μL del pocillo anterior hasta el segundo pocillo de cada fila. De este modo, el primer pocillo de cada fila se utiliza como control positivo de crecimiento bacteriano en ausencia de antibiótico. Se incuba la placa M-96 a 30 ºC con aireación durante 20-22h. y transcurrido este periodo, se mide la absorbancia de cada pocillo a una longitud de onda de 600 nm para determinar el crecimiento bacteriano.
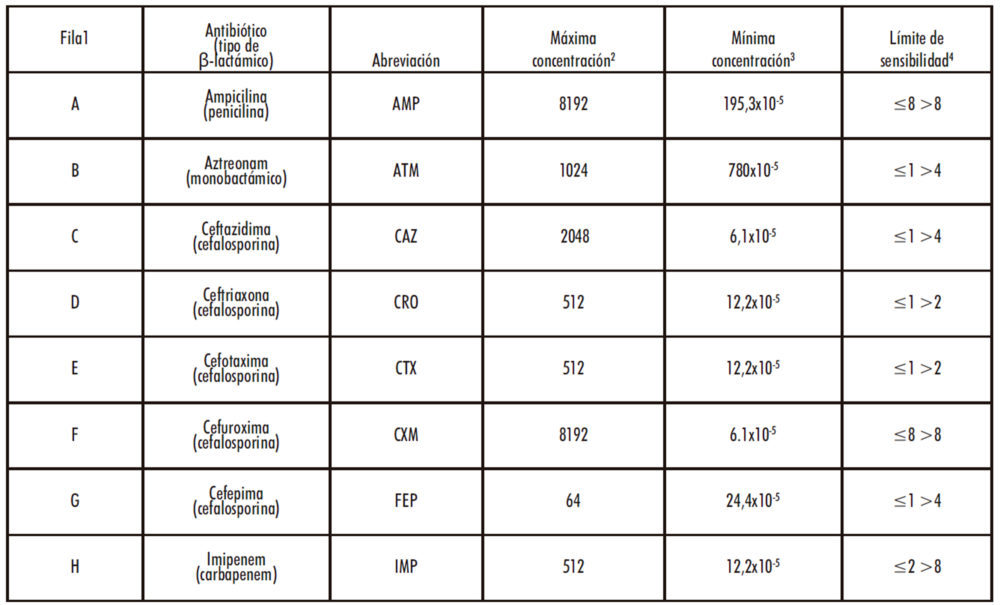
De acuerdo con el Dr. Barry Hall y la literatura consultada (Barlow, Hall 2002); (Thierry Naas, Laurence Vandel et al. 1994) se establecieron, para cada proteína recombinante, las concentraciones máxima y mínima de cada uno de los antibióticos utilizados para medir la actividad hidrolítica (Tabla 1). Además, se establecieron las concentraciones de fármaco a partir de las cuales se considera que E. coli es sensible o resistente al efecto antibiótico (Tabla 1).
Tabla 1: Resumen de las concentraciones utilizadas en el trabajo junto con los criterios de sensibilidad y resistencia establecidos.
1 Se índica la fila que corresponde a cada fila de la placa M-96.
2,3 Indica la concentración máxima (2) y mínima (3) en μg/mL de inhibidor utilizada en cada caso
4 Se indican (en μg/mL) las concentraciones de antibiótico por debajo de la cual (izquierda) o por encima de la cual (derecha) se considera que una población de E. coli es sensible o resistente, respectivamente, a un antibiótico.
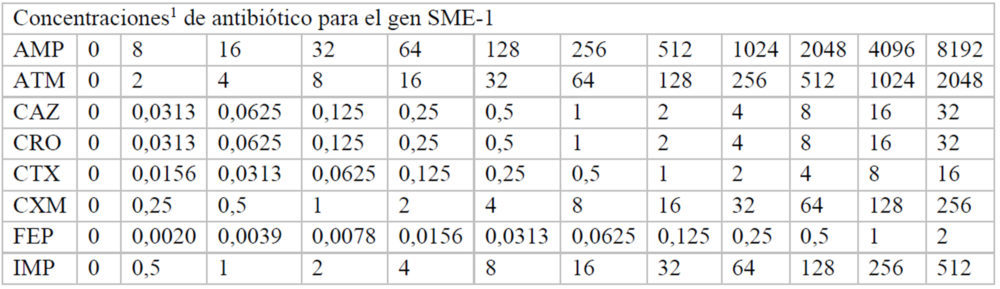
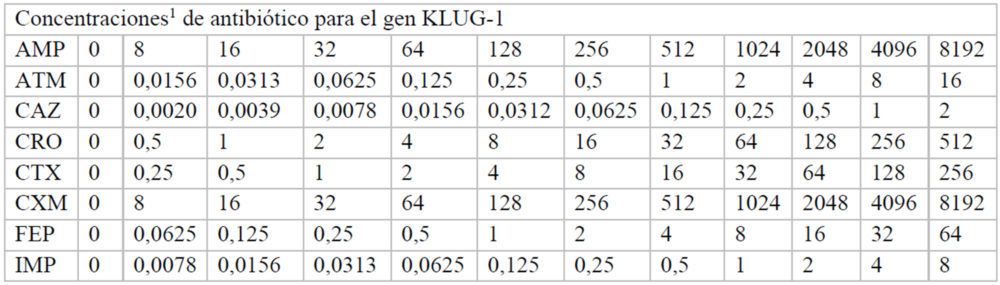
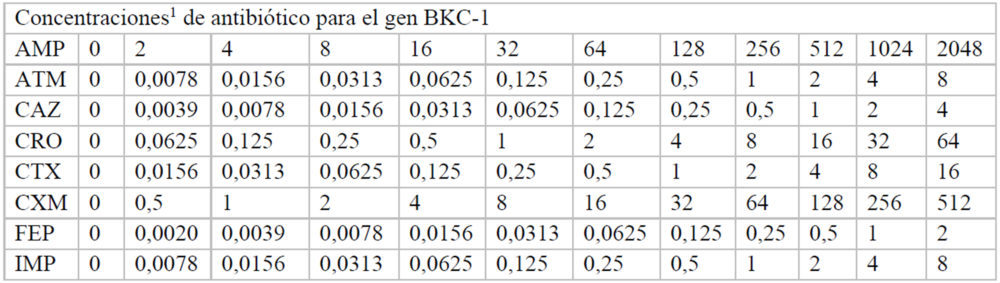
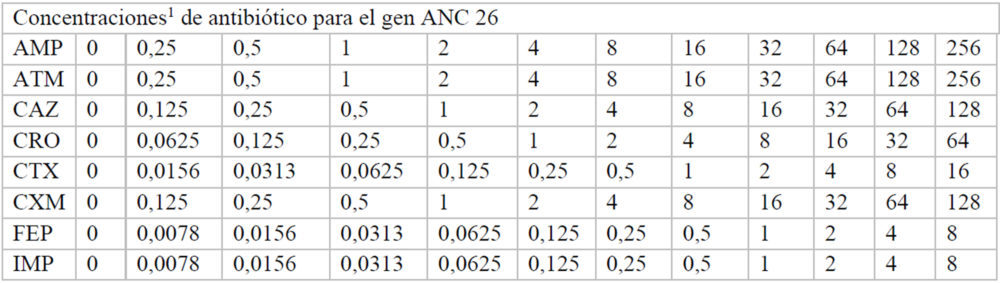
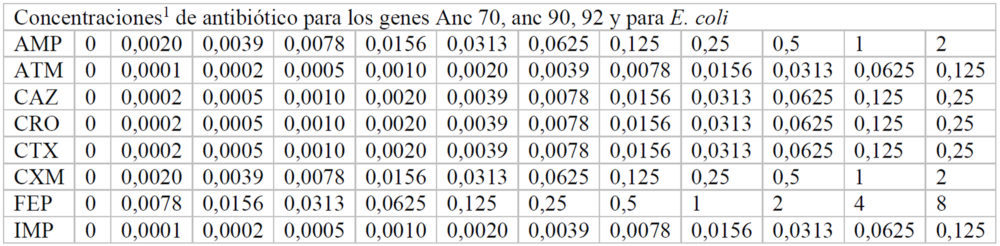
Para hallar las concentraciones máxima y mínima utilizadas, inicialmente se realizó un experimento en el que se usaron las mismas concentraciones de antibiótico en cada una de las placas correspondiente a cada β-lactamasa. A partir de estos datos, se pudieron ajustar las concentraciones adecuadas de cada antibiótico para calcular de manera fiable, a cada β-lactamasas, los valores de inhibición de crecimiento. Las concentraciones finales de cada fármaco utilizadas en cada caso se indican en el Anexo (Tablas A2-A8).
Tablas A2-A8: Concentraciones utilizadas de cada antibiótico en cada uno de los pocillos de la placa M-96
A2
A3
A4
A5
A6
A7
A8
3.2. Comprobación del sistema biológico escogido para analizar las β-lactamasas ancestrales
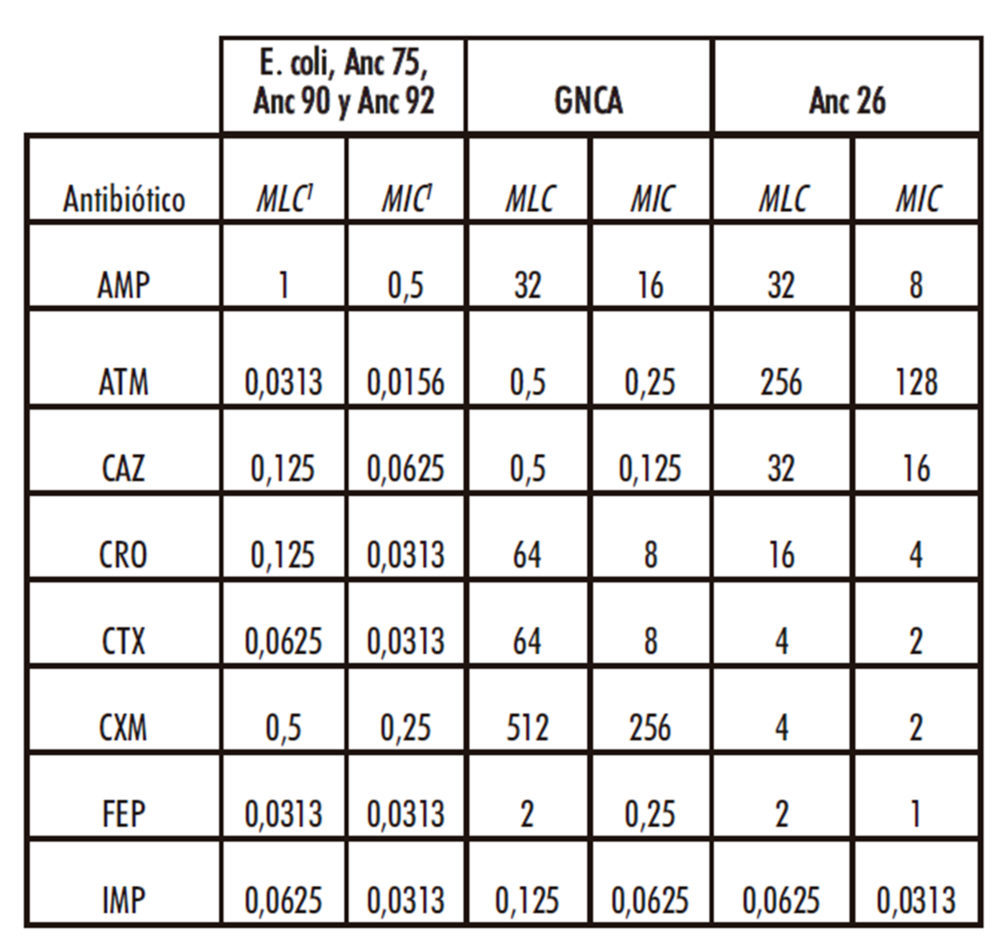
Para comprobar la resistencia de un gen ancestral ya caracterizado, se ha decidido medir los valores de MICs del gen reconstruido GNCA frente a los 8 antibióticos utilizados en este ensayo. Estos valores nos dan información de la actividad de este gen expresado a partir del vector que se utiliza en nuestro estudio (el pACSE3), confirmando que el vector es viable y permite la funcionalidad de β-lactamasas ancestrales. Asimismo, se calculan las MICs de bacterias E. Coli DH5E que carecen del plásmido pACES3, como control para verificar que las bacterias utilizadas no presentan resistencia a los fármacos utilizados y en el caso de que la presenten, establecer esos valores como base.
Los resultados obtenidos demuestran en nuestro sistema, que el gen GNCA se expresa como una proteína que presenta resistencia a los antibióticos AMP, CAZ, CRO y CTX, tal como se observó anteriormente (Risso, Gavira et al. 2013). Además, las bacterias E. coli no transformadas son susceptibles a todos los compuestos utilizados en este trabajo, por lo tanto, la resistencia a los antibióticos se debe específicamente a las β-lactamasas recombinantes expresadas por el plásmido pACSE3 (Tabla 2). Estos datos demuestran que nuestro sistema es efectivo para poder determinar la actividad hidrolítica de distintas β-lactamasas.
3.3. Obtención de MIC y MLC de cada gen
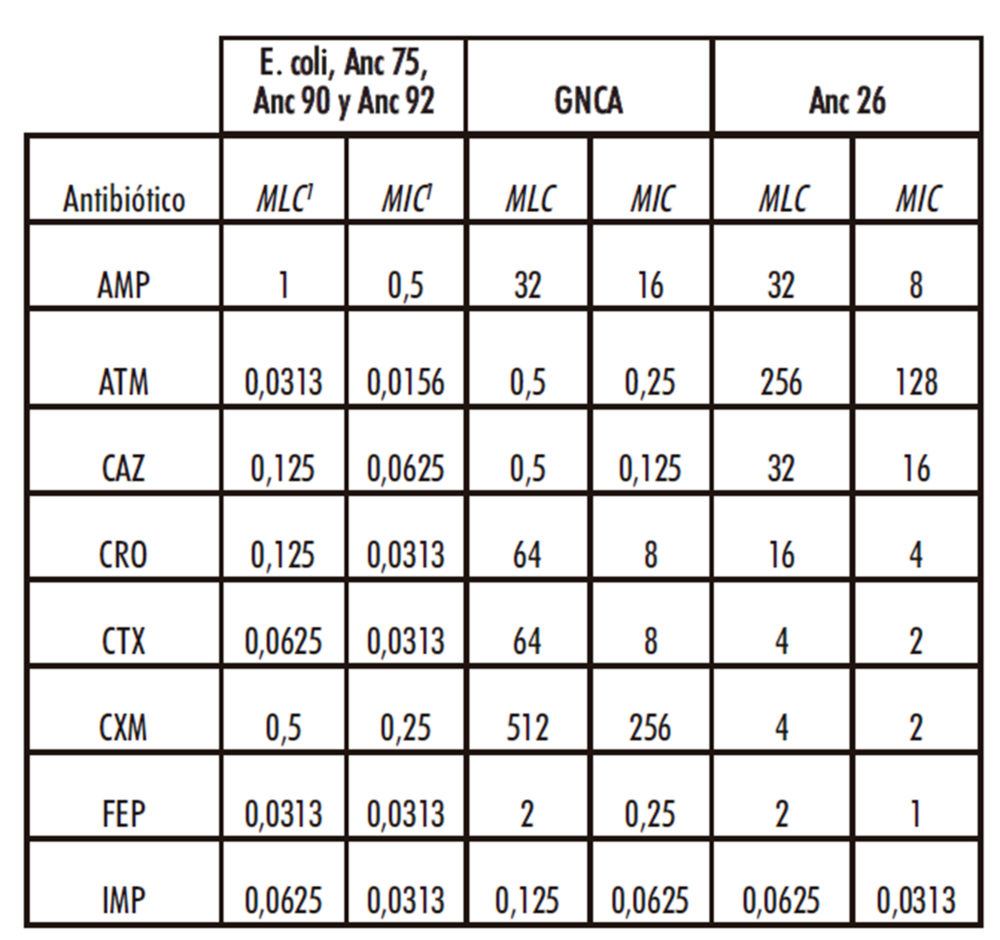
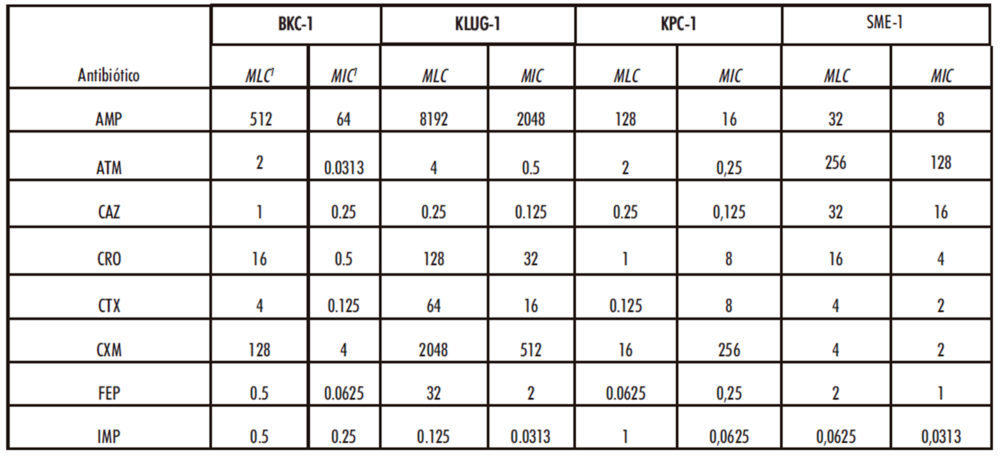
A partir de los datos obtenidos se obtiene los valores de MICs de cada una de las β-lactamasas estudiadas frente a cada uno de los antibióticos utilizados. Se determinó también de manera separada el valor de la mínima concentración letal de antibiótico (MLC del inglés “minimal lethal concentration”) ya que es un error frecuente considerar los valores de MIC y MLC equivalentes, cuando en realidad el valor de MIC corresponde a la concentración de antibiótico que inhibe el crecimiento bacteriano y que no tiene por qué coincidir con el valor MLC. Los valores MIC y MLC de cada uno de las β-lactamasas utilizadas, así como el control de E. coli no transformada y controles aparecen en las tablas 2 y 3.
Tabla 2: Concentraciones inhibitorias y letales de los distintos antibióticos a E. coli, GNCA, Anc 26, Anc 75, Anc 90 y Anc 92
1 los valores MIC y MLC se muestran en μg/mL
Tabla 3: Concentraciones inhibitorias y letales de los distintos antibióticos frente a las β-lactamasas actuales
Los valores MIC y MLC se muestran en μg/mL
Los resultados muestran que las cuatro β-lactamasas utilizadas en este estudio presentan distintos grados de resistencia a los antibióticos analizados, mientras que, de las β- lactamasas ancestrales observados en este estudio, sólo el producto de expresión de Anc26 muestra resistencia a todos los antibióticos utilizados excepto frente a IMP, mientras que el resto no resultaron activas.
3.4. Obtención del mejor generalista y el mejor especialista
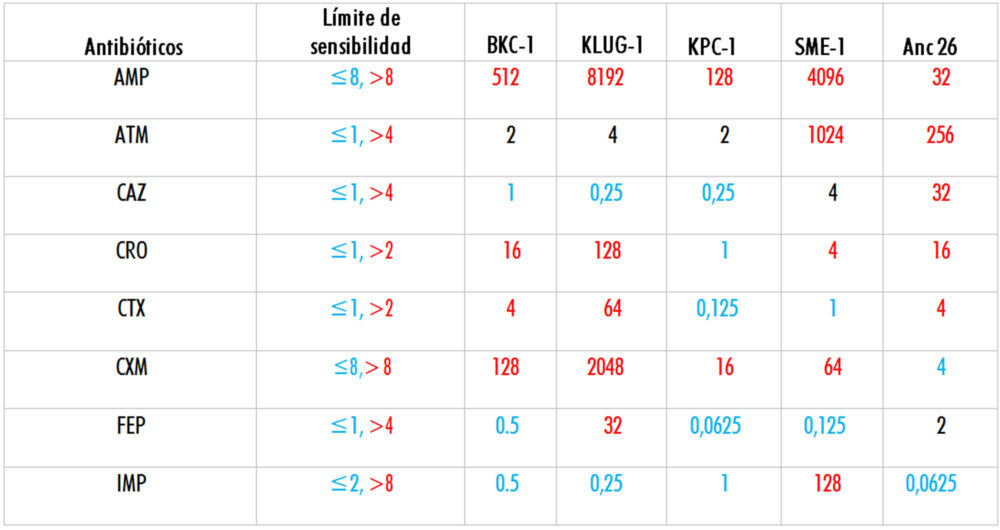
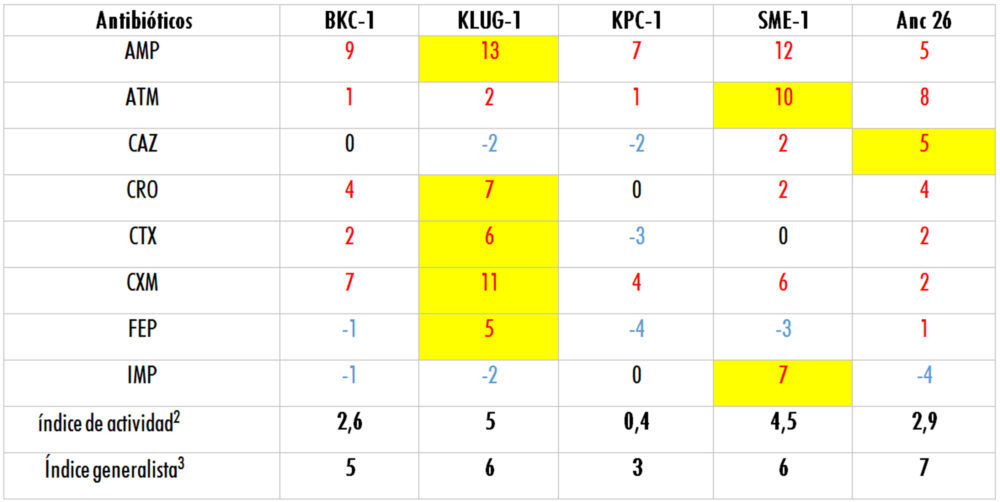
Para tener una mejor comprensión de los resultados, se decidió analizar que enzima presenta resistencia a mayor número de antibióticos (más generalista) y cual es el más resistente a cada uno de los β-lactámicos (más especialista). Para este análisis se calcula el logaritmo en base 2 de los valores MLC obtenidos en cada caso (Tabla 4). De este modo se obtiene un valor arbitrario adimensional que sirve para comparar fácilmente la actividad de los distintos genes estudiados en este trabajo (Tabla 5).
Tabla 4: Resumen de los valores de MLC obtenidos1
1Los valores MLC se muestran en μg/mL. Se indica en color rojo los valores que están por encima del límite de sensibilidad (resistentes), en negro los valores que están en el límite de sensibilidad y en azul los valores que están por debajo del límite de sensibilidad (sensibles).
En relación a las β-lactamasas actuales, los resultados demuestran que KLUG-1 es la β-lactamasa más generalista de las examinadas en este trabajo, además, presenta los valores de resistencia más altos frente a 5 de los 8 antibióticos estudiados (AMP, CRO, CTX, CXM y FEP), mientras que SME-1 es la que presenta los valores más altos de resistencia frente a ATM e IMP, siendo la única enzima de las seleccionadas que presenta resistencia a este carbapenem. Finalmente, el producto del gen Anc 26 presenta el mayor nivel de resistencia frente a ceftazidima de todas las β-lactamasas estudiadas y se comporta como un generalista intermedio.
Tabla 5: valores de los Logaritmos obtenidos a partir de los datos de MLC mostrados en la Tabla 1
1Se índica en color rojo los valores que corresponden con MLCs que están por encima del límite de sensibilidad (resistentes), en negro los valores que se corresponden con los MLCs que están en el límite de sensibilidad y en azul los valores que se corresponden con los MLCs que están por debajo del límite de sensibilidad (sensibles). Se remarca en amarillo el valor más alto de resistencia para cada antibiótico.
2 El índice de actividad es el promedio de los valores de logaritmo obtenidos.
3 El índice generalista señala el número de antibióticos frente a los cuales la β-lactamasa correspondiente muestra resistencia
4. DISCUSIÓN
En este trabajo se han obtenido diferentes secuencias proteicas de β-lactamasas ancestrales para analizar su actividad y posteriormente compararlas con secuencias actuales. Como se establecía en la introducción, estas enzimas son de vital interés, debido a los estragos sanitarios que producen y pueden producir en el futuro. Es por ello, que en este trabajo se caracterizó las diferentes actividades hidrolíticas que presentan las β-lactamasas estudiadas y sirve para validar la metodología de reconstrucción de proteínas ancestrales.
La clasificación de las enzimas estudiadas según el índice de actividad establecido en este trabajo es KLUG-1 > SME-1> Anc 26 > BKC-1 > KPC-1. Sin embargo, aunque KLUG-1 es la β-lactamasas que presenta los valores más altos de resistencia frente al mayor número de antibióticos, es el producto génico ancestral Anc 26 el más generalista al mostrar resistencia a mayor número de antibióticos (Tabla 5), siendo sensible únicamente a imipenem, un antibiótico altamente resistente a la acción de las β-lactamasas (Finch, Whitley et al. 2010). Aunque es cierto que los antibióticos utilizados en este estudio son sintéticos o semi-sintéticos, se considera que los genes ancestralescodifican proteínas más generalistas que pueden evolucionar mediante selección convirtiéndose en genes especialistas (Siddiq, Hochberg et al. 2017; Zou, Risso et al. 2015).
Esta idea se sustenta aún más cuando existen ciertos estudios que demuestran que las β-lactamasas evolucionaron directamente de las PBPs (enzimas contra las que actúanlos β-lactámicos; Meroueh, Minasov et al. 2003).
Se observa en los resultados que el producto génico de Anc 26, es el más resistente frente a ceftazidima (CAZ). Esta cefalosporina se caracteriza por ser un antibiótico estratégico, debido a que no se utiliza a gran escala en el ámbito hospitalario. Es posible que este hecho haya impedido que las β-lactamasas actuales analizadas en este estudio no hayan estado suficientemente expuestas a este antibiótico para adquirir una resistencia mayor que la de la secuencia ancestral. Por el contario, la resistencia mostrada por Anc26 a este compuesto se debería a su promiscuidad inherente. En un estudio en el que se evolucionó in vitro la ESBL CTX-M9 (Delmas, Robin et al. 2006), una β-lactamasa filogenéticamente muy cercana a KLUG-1, se obtuvieron diferentes mutantes seleccionando contra CAZ tras varias rondas de selección. En dicho estudio, la variante que más resistencia mostró frente a CAZ (MIC = 128 μg/mL, con respecto a MIC 1μg/mL mostrado por la proteína parental), presenta 5 sustituciones con respecto a CTX-M9: P167S, A109T, G146R, T227A, Q254P (variante A6B1C1). La proteína derivada de Anc 26 presenta 3 de estos cambios (T180, A198 y P221 correspondientes a los cambios A109T, T227A, Q254P que aparecen en A6B1C1), además de contener una sustitución conservativa (K117 en la posición correspondiente G146R que aparece en A6B1C1). El resto de las proteínas actuales (BKC-1, KLUG-1, KPC-1 y SME-1) solamente presentan una o dos de estas mutaciones. La presencia de estos cambios en la estructura de Anc 26 podría ser responsable de la mayor resistencia frente CAZ. Este hecho refuerza el poder predictivo que pueden tener las proteínas ancestrales a la hora de mostrar residuos que pudieran ser determinantes de resistencia a un antibiótico.
En este estudio hemos obtenido distintas secuencias ancestrales que representaran diferentes nodos del árbol filogenético perteneciente a la historia evolutiva de las β-lactamasas. Sin embargo, solamente una de ellas (Anc 26) ha resultado ser funcional. Una característica que presenta la β-lactamasa Anc 26 es un extremo N-terminal unos 10 aminoácidos más largo que el resto de las proteínas ancestrales inferidas en este estudio.
Las β-lactamasas se caracterizan por tener una región muy variable en este extremoterminal (Philippon, Slama et al. 2016), que es imprescindible para que la enzima sea exportada al espacio periplásmico donde realizan su función (Abraham Minsky, Richard G. Summers et al. 1986), lo que dificulta el alineamiento de secuencia, la obtención de una filogenia fiable y por lo tanto la inferencia de la secuencia ancestral correspondiente.
Estos problemas se traducen en la obtención de proteínas truncadas o directamente mal plegadas y podrían ser la causa de la falta de actividad de las proteínas obtenidas a partir de Anc 75, Anc 90 y Anc 92. En este trabajo se ha obtenido una secuencia ancestral funcional para poder comparar su actividad con la de sus homólogos actuales bajo las mismas condiciones experimentales. Los resultados obtenidos están en concordancia con los resultados previamente publicados que demuestran la relevancia de la reconstrucción de proteínas ancestrales para caracterizar y entender el comportamiento de sus homólogos actuales (Risso, Gavira et al. 2013; Novais, Comas et al. 2010).
Un objetivo adicional de este trabajo es establecer un punto de partida para la realización de experimentos de evolución dirigida de proteínas en paralelo tanto de las β-lactamasas ancestrales como de las actuales. Este tipo de estudios podrían proporcionar una gran información sobre la evolución natural de las β-lactamasas que nos podría permitir anticipar y predecir el tipo mutaciones de resistencia antes de su aparición.
5. CONCLUSIÓN
Los datos presentados demuestran que la proteína obtenida a partir de la secuencia ancestral reconstruida Anc 26 es activa y muestra resistencia a varios antibióticos. Esta secuencia muestra niveles de resistencia frente a CAZ mayores que cualquiera de las β-lactamasas actuales estudiadas. Los datos obtenidos confirman que las proteínas ancestrales son más generalistas que las proteínas actuales, esta estrategia podría servir como un punto de partida prometedor para la obtención de proteínas de interés biotecnológico, ya que la promiscuidad es una de las principales características a la hora de producir nuevas proteínas mediante evolución in vitro de proteínas (Alcalde 2017).
6. BIBLIOGRAFÍA
- Abraham Minsky, Richard G. Summers , Jeremy R. Knowles,1986. Secretion of β -lactamase into the Periplasm of Escherichia coli: Evidence for a Distinct Release Step Associated with a Conformational Change. Proceedings of the National Academy of Sciences of the United States of America, 83(12), pp. 4180-4184.
- Alcalde, M., 2017. When directed evolution met ancestral enzyme resurrection. Microbial Biotechnology, 10(1), pp. 22-24.
- Alexander Fleming, 1929. On the antibacterial action of cultures of a Penicillium, with special reference to their use in the isolation of B. influenzae.
- Aminov, R.I., 2010. A Brief History of the Antibiotic Era: Lessons Learned and Challenges for the Future. Frontiers in microbiology, 1, pp. 134.
- Arenas, M., Weber, C.C., Liberles, D.A. , Bastolla, U., 2017. ProtASR: An Evolutionary Framework for Ancestral Protein Reconstruction with Selection on Folding Stability. Systematic biology, 66(6), pp. 1054-1064.
- Barlow, M. , Hall, B.G., 2003a. Experimental Prediction of the Natural Evolution of Antibiotic Resistance. Genetics, 163(4), pp. 1237-1241.
- Barlow, M. , Hall, B.G., 2003b. Experimental Prediction of the Natural Evolution of Antibiotic Resistance. Genetics, 163(4), pp. 1237-1241.
- Barlow, M. , Hall, B.G., 2002. Predicting evolutionary potential: in vitro evolution accurately reproduces natural evolution of the tem beta-lactamase. Genetics, 160(3), pp. 823-832.
- Bush, K., Jacoby, G.A. , Medeiros, A.A., 1995. A functional classification scheme for beta-lactamases , its correlation with molecular structure. Antimicrobial agents and chemotherapy, 39(6), pp. 1211-1233.
- Bush, K. , Bradford, P.A., 2016. β-Lactams and β-Lactamase Inhibitors: An Overview. Cold Spring Harbor perspectives in medicine, 6(8), pp. a025247.
- Christiane Bébéar , Janet A. Robertson, 1996. Determination Of Minimal Inhibitory Concentration. In: ACADEMIC PRESS INC, ed, Molecular and Diagnostic Procedures in Mycoplasmology.
- Crofts, T.S., Gasparrini, A.J. , Dantas, G., 2017. Next-generation approaches to understand and combat the antibiotic resistome. Nature reviews. Microbiology, 15(7), pp. 422-434.
- David L. Paterson, Robert A. Bonomo, 2005. Extended-Spectrum-Lactamases: a Clinical Update.
- Delmas, J., Robin, F., Carvalho, F., Mongaret, C. , Bonnet, R., 2006.Prediction of the Evolution of Ceftazidime Resistance in Extended-Spectrum-Lactamase CTX-M-9. Antimicrobial Agents and Chemotherapy, 50(2), pp. 731-738.
- Donald J Tipper , Jack L. Strominger, 1965. Mechanism of action of Penicillins: a proposal based on their structural similarity to acyl-d-alanyl-d-alanine*.
- Fernandes, R., Amador, P. , Prudêncio, C., 2013. β-Lactams. Reviews in Medical Microbiology, 24(1), pp. 7-17.
- Finch, R.G., Whitley, R.J., Norrby, S.R. , Greenwood, D., 2010. Antibiotic and chemotherapy: anti-infective agents and their use in therapy. Saunders.
- Gomez-Fernandez, B.J., Garcia-Ruiz, E., Martin-Diaz, J., Gomez De Santos, P., Santos-Moriano, P., Plou, F.J., Ballesteros, A., Garcia, M., Rodriguez, M., Risso, V.A., Sanchez-Ruiz, J.M., Whitney, S.M. , Alcalde, M., 2018. Directed -in vitro- evolution of Precambrian and extant Rubiscos. Scientific reports, 8(1), pp. 5532-11.
- Gumulya, Y. , Gillam, E.M.J., 2017. Exploring the past and the future of protein evolution with ancestral sequence reconstruction: the ‘retro approach to protein engineering. The Biochemical journal, 474(1), pp. 1-19.
- Hall, B.G., 2004. Predicting the evolution of antibiotic resistance genes. Nature Reviews Microbiology, 2(5), pp. 430-435.
- IACG. NO PODEMOS ESPERAR: Asegurar el futuro contra las infecciones farmacorresistentes. Madrid: Prisacom; 2019 Mar 15.
- Kållberg, C., Årdal, C., Salvesen Blix, H., Klein, E., M Martinez, E., Lindbæk, M., Outterson, K., Røttingen, J. , Laxminarayan, R.,2018. Introduction and geographic availability of new antibiotics approved between 1999 and 2014. PloS one, 13(10), pp. e0205166.
- Karen Bush, 2018. Past and Present Perspectives on β-Lactamases. Antimicrobial Agents and Chemotherapy. 62. pp. 1076-18.
- Karen Bush, Patrice Courvalin, Gautam Dantas, Julian Davies, Barry Eisenstein, Pentti Huovinen, George A. Jacoby, Roy Kishon, Barry N. Kreiswirth , Elizabeth Kutter, 2011. Tackling antibiotic resistance. BMJ (Clinical research ed.), 340(may18 2),.
- Kong, K., Schneper, L. , Mathee, K., 2010. Beta-lactam antibiotics: from antibiosis to resistance and bacteriology. Apmis, 118(1), pp. 1-36.
- Livermore, D., 2004. Can better prescribing turn the tide of resistance? Nature Reviews Microbiology, 2(1), pp. 73-78.
- Luepke, K.H., Suda, K.J., Boucher, H., Russo, R.L., Bonney, M.W., Hunt, T.D. , Mohr, J.F., 2017. Past, Present, and Future of Antibacterial Economics: Increasing Bacterial Resistance, Limited Antibiotic Pipeline, and Societal Implications. Pharmacotherapy: The Journal of Human Pharmacology and Drug Therapy, 37(1), pp. 71-84.
- Melise Chaves Silveira, Rangeline Azevedo Da Silva, Fábio Faria Da Mota, Marcos Catanho, Rodrigo Jardim, Ana Carolina,R. Guimarães, Antonio B De Miranda, 2018. Systematic Identification and Classification of β-Lactamases Based on Sequence Similarity Criteria: β-Lactamase Annotation. , pp. 1-11.
- Meroueh, S.O., Minasov, G., Lee, W., Shoichet, B.K. , Mobashery, S., 2003. Structural Aspects for Evolution of β-Lactamases from Penicillin-Binding Proteins. Journal of the American Chemical Society, 125(32), pp. 9612-9618.
- Neelanjana Pandey , Marco Cascella, 2019. Beta Lactam Antibiotics. StatPearls, 20(10) pp. 20.
- Nicoletti, A.G., Marcondes, M.F.M., Martins, Willames M B S, Almeida, L.G.P., Nicolás, M.F., Vasconcelos, A.T.R., Oliveira, V. , Gales, A.C., 2015. Characterization of BKC-1 Class A Carbapenemase from Klebsiella pneumoniae Clinical Isolates in Brazil. Antimicrobial agents and chemotherapy, 59(9), pp. 5159-5164.
- Novais, A., Comas, I., Baquero, F., Cantón, R., Coque, T.M., Moya, A., González-Candelas, F. , Galán, J., 2010. Evolutionary Trajectories of Beta-Lactamase CTX-M-1 Cluster Enzymes: Predicting Antibiotic Resistance. PLoS pathogens, 6(1), pp. e1000735.
- Arveen Salahuddin, Amit Kumar , Asad U. Khan, 2018. Structure, Function of Serine and Metallo-β-lactamases and their Inhibitors. Current Protein & Peptide Science, 19(2), pp. 130-144.
- Pazos, M. , Peters, K., 2019. Peptidoglycan. Sub-cellular biochemistry, 92, pp. 127-168.
- Philippon, A., Slama, P., Dény, P. , Labia, R., 2016. A Structure-Based Classification of Class A β-Lactamases, a Broadly Diverse Family of Enzymes. Clinical microbiology reviews, 29(1), pp. 29-57.
- Poirel, L., Kampfer, P. , Nordmann, P., 2002. Chromosome-Encoded Ambler Class A -Lactamase of Kluyvera georgiana, a Probable Progenitor of a Subgroup of CTX-M Extended-Spectrum -Lactamases. Antimicrobial Agents and Chemotherapy, 46(12), pp. 4038-4040.
- R. P. Ambler, 1980. The structure of β-lactamases. The Royal Society.
- Risso, V.A., Gavira, J.A., Mejia-Carmona, D.F., Gaucher, E.A. , Sanchez-Ruiz, J.M., 2013. Hyperstability and Substrate Promiscuity in Laboratory Resurrections of Precambrian β-Lactamases. Journal of the American Chemical Society, 135(8), pp. 2899-2902.
- Sambrook, J. , Green, M.R., 2012. Molecular cloning: a laboratory manual. 4th edn. United States: Cold Spring Harbor Laboratory Press.
- Siddiq, M.A., Hochberg, G.K. , Thornton, J.W., 2017. Evolution of protein specificity: insights from ancestral protein reconstruction. Current Opinion in Structural Biology, 47, pp. 113-122.
- Thierry Naas, Laurence Vandel, Wladimir Sougakoff, David M. Livermore , Patrice Nordmann`, 1994. Hydrolyzing Class A PLactamase, Sme-1, from.
- Thornton, J.W., 2004. Resurrecting ancient genes: experimental analysis of extinct molecules. Nature Reviews Genetics, 5(5), pp. 366-375.
- Ur Rahman, S., Ali, T., Ali, I., Khan, N.A., Han, B. , Gao, J., 2018. The Growing Genetic and Functional Diversity of Extended Spectrum Beta-Lactamases. BioMed research international, 2018, pp. 9519718-14.
- Usman Umar Zango, Munir Ibrahim, Sadiq Abdurrahman, Abubakar Shawai , Ibrahim Muhammad Shamsuddin, 2019. A review on β-lactam antibiotic drug resistance. MOJ Drug Des Develop Ther., pp. 52-58.
- Vollmer, W., Blanot, D. , De Pedro, M.A., 2008. Peptidoglycan structure and architecture. FEMS Microbiology Reviews, 32(2), pp. 149-167.
- WHO, 2018. WHO Report on Surveillance of Antibiotic Consumption
- Yigit, H., Queenan, A.M., Anderson, G.J., Domenech-Sanchez, A., Biddle, J.W., Steward, C.D., Alberti, S., Bush, K. , Tenover, F.C., 2001. Novel Carbapenem-Hydrolyzing β-Lactamase, KPC-1, from a Carbapenem-Resistant Strain of Klebsiella pneumoniae. Antimicrobial Agents and Chemotherapy, 45(4), pp. 1151-1161.
- Zakas, P.M., Brown, H.C., Knight, K., Meeks, S.L., Spencer, H.T.,Gaucher, E.A. , Doering, C.B., 2017. Enhancing the pharmaceutical properties of protein drugs by ancestral sequence reconstruction. Nature biotechnology, 35(1), pp. 35-37.
- Zapun, A., Contreras-Martel, C. , Vernet, T., 2008. Penicillin-binding proteins and β-lactam resistance. FEMS Microbiology Reviews, 32(2), pp. 361-385.
- Zhang, G. , Hao, Q., 2011. Crystal structure of NDM‐1 reveals a common β‐lactam hydrolysis mechanism. The FASEB Journal, 25(8), pp. 2574-2582.
- Zou, T., Risso, V.A., Gavira, J.A., Sanchez-Ruiz, J.M. and Ozkan, S.B., 2015. Evolution of Conformational Dynamics Determines the Conversion of a Promiscuous Generalist into a Specialist Enzyme. Molecular biology and evolution, 32(1), pp. 132-143.
ANEXO
Anexo ML-phylo
Anexo ML-phylo
Tabla A1: Primers utilizados en el clonaje