1. VITALISMO Y BIOQUÍMICA
Hasta la mitad del siglo XIX muchos biólogos creían que los fenómenos vitales característicos de los seres vivos eran debidos a fuerzas especiales distintas de las de la Física y la Química. Estas fuerzas se denominaron “fuerza vital” y se pensaba que solo estaba presente en los organismos vivos. Las ideas del vitalismo pueden parecernos extrañas en la actualidad, pero los científicos de entonces habrían encontrado muy difícil explicar las actividades que tienen lugar en los seres vivos, solamente en términos de fuerzas físicas y químicas.
La idea de que muchas de las actividades de la vida pueden ser entendida en términos de química tiene sus orígenes en los estudios de la fermentación. Antoine Lavoisier (1743-1794) fue uno de los fundadores de la Química moderna, y se interesó entre otras cosas en la fermentación, una práctica empleada desde tiempos remotos por la que algunas frutas fermentan y producen alcohol.
Observando que el principal componente del jugo de uva era azúcar y que el principal producto de la fermentación era etanol Lavoisier propuso que “la fermentación era una reacción química en la que el azúcar del jugo de la uva se convertía en el etanol del vino producido”. Investigando más mostró que el “fermento” presente durante la fermentación, era el elemento que jugaba un papel clave en la reacción química. Ahora bien, si se sustituía el jugo de uva por glucosa y se añadía una pequeña cantidad de fermento se producía también etanol, igual que durante una fermentación normal. Exactamente lo que hacía el fermento no estaba claro entonces, aunque un poco más tarde, Theodor Schawn el famoso autor de la teoría celular y otros, especulaban ya con que el fermento era la levadura.
La claridad llegó un cuarto de siglo después con el trabajo de Louis Pasteur, el gran científico francés. Solicitado por la industria del vino para investigar porqué algunas veces las fermentaciones se estropeaban, mostró que en algunos casos la fermentación producía ácido láctico en lugar de etanol. El examen microscópico de los sedimentos de las vasijas de fermentación reveló que las que producían etanol contenían levaduras, en tanto que las que daban lugar a ácido láctico contenían otro microorganismo, en lugar de levadura. Pasteur propuso entonces que la vida microbiana de la levadura era la responsable de la producción de etanol y que el otro microorganismo era el que producía ácido láctico. El aspecto más importante para la Biología de este descubrimiento fue la conclusión de que el crecimiento de una célula viva daba lugar a la producción de una sustancia química. Esto llevo a Pasteur a concluir que “las reacciones químicas son la expresión de la vida de las células”. Para confirmar esta idea Pasteur inoculó sedimentos de ambos tipos de vasijas en botellas que contenían azúcares y demostró que las levaduras producían etanol y el otro microorganismo, ácido láctico. Estos experimentos y el estudio de otras fermentaciones le llevaron a proponer más adelante que las reacciones químicas eran “actos fisiológicos” que daban lugar a múltiples productos, los cuales eran necesarios para las células.
El siguiente avance, fue la demostración de que las células vivas contenían sustancias que podían llevar a cabo reacciones químicas análogas a las que se producían durante la fermentación. Así, algo más tarde, el también científico francés Berthelot (1827-1907), tras triturar células de levaduras obtuvo un extracto soluble que purificado y separado de las células fue capaz de hidrolizar el azúcar sacarosa en sus componentes glucosa y fructosa. La sustancia responsable de esta actividad la denominó invertasa y concluyó, que las células no eran necesarias para que las reacciones químicas de la fermentación tuvieran lugar sino que contenían sustancias que eran “activas” por sí mismas y que estas podían llevar a cabo la fermentación aunque las células de las que procedían no estuvieran presentes.
Aproximadamente 30 años más tarde, al principio del siglo XX, estas observaciones fueron continuadas por dos hermanos alemanes, Hans y Eduard Buchner, los cuales extrajeron de las células de levadura un enzima que resultó ser responsable de las reacciones químicas. Los Buchner llevaron a cabo la rotura de células de levadura moliéndolas en un mortero y filtrando después los restos celulares para obtener un extracto celular. Este extracto era capaz de fermentar azúcares y producir alcohol demostrando que dicha reacción química podía tener lugar “in vitro”. El trabajo de los Buchner permitió concluir que las células de levadura contenían una sustancia, llamada zymasa (un enzima) y que esta sustancia era la responsable de la reacción química que convertía el azúcar en alcohol.
Se puede decir que los resultados anteriores formaron la piedra angular de la Bioquímica. Mostraron que la fermentación, un fenómeno asociado con la vida, podía ser reducida a reacciones químicas catalizadas por sustancias intracelulares llamadas enzimas.
2. DE LAS REACCIONES ENZIMÁTICAS A SU REGULACIÓN Y ORGANIZACIÓN EN LAS CÉLULAS
Generalizando a partir de estos resultados se puede decir que la mayoría de las actividades de las células están basadas en reacciones químicas catalizadas por enzimas. La Bioquímica ha confirmado este punto de vista en numerosas ocasiones. Se ha llegado a establecer, que dentro de las células tienen lugar miles de reacciones químicas que operan de manera simultánea, y que éstas, son responsables de los fenómenos vitales característicos de los organismos vivos. Esta multitud de reacciones, son llevadas a cabo por una gran variedad de enzimas, cada una de las cuales, requiere un microambiente específico para llevar a cabo su función. Los diferentes microambientes están caracterizados por propiedades tales como, el pH, la concentración iónica, la disponibilidad de sustratos, etc.
Para trabajar de manera efectiva los microambientes necesitan estar separados unos de otros y las células utilizan una serie de mecanismos para llevar a cabo dicha separación. Al nivel más simple la superficie de los enzimas proporciona espacios que permiten un cierto aislamiento del ambiente local. Si los enzimas se combinan entre sí se generan complejos enzimáticos que presentan mejores oportunidades para conseguir el microambiente apropiado. Esto ha dado lugar a la canalización de sustratos y productos desde un enzima al siguiente a través de una serie ordenada de reacciones que constituyen las llamadas “rutas metabólicas”.
Los complejos de enzimas pueden llegar a formar verdaderas máquinas moleculares como los ribosomas, responsables de la síntesis de proteínas. A nivel superior, orgánulos subcelulares rodeados de membranas como el núcleo, las mitocondrias, cloroplastos etc, proporcionan una mayor posibilidad de generar compartimentos. Finalmente, la célula entera rodeada por una membrana separa el contenido celular total del mundo externo. Esta variedad de microambientes organizados da lugar a la compleja estructura de la célula y permite entender los procesos vitales a nivel celular.
Otro mecanismo usado para separar microambientes es utilizar cambios de la célula en el tiempo. Esto se pone de manifiesto durante el ciclo celular cuando los cambios tienen lugar en el ambiente local de los cromosomas. Así durante la mitosis, los cromosomas situados en el núcleo se condensan y tras la rotura de la membrana nuclear se liberan en el citoplasma para permitir que tenga lugar la separación de las cromátidas. Posteriormente, los cromosomas se descondensan y se confinan de nuevo en el núcleo para permitir que los enzimas de la síntesis de DNA (DNA polimerasas) operen durante la siguiente fase del ciclo. El DNA puede así asociarse con diferentes microambientes químicos.
Gracias a estos y otros paradigmas, los bioquímicos, convertidos en biólogos moleculares comparten desde hace tiempo la idea de que los fenómenos de la vida pueden ser entendidos en términos de Química.
Es importante entender que se trata de una forma de química altamente organizada.
Los miles de reacciones químicas intracelulares deben estar no solo ordenadas sino también reguladas para alcanzar una buena organización en las células
Un fisiólogo americano, Jaques Loeb, postuló ya en 1912 que la célula debía ser considerada como una máquina química y hay dos características de las máquinas importantes para entender esta comparación. La primera es conocer cómo están reguladas las reacciones químicas y la segunda, cómo se comunican unas con otras.
Respecto a la regulación de las reacciones enzimáticas, se ha utilizado en ocasiones la analogía con una máquina de vapor y mas en concreto, con una pieza de la misma llamada regulador que regula el flujo de vapor. El regulador se compone de dos bolas que giran alrededor de un eje. A medida que la máquina va más deprisa, las bolas se disponen horizontalmente por la fuerza centrífuga y automáticamente, reducen el flujo de vapor que va hacia la máquina desde la caldera con lo que se reduce su velocidad. Esta regulación hacia atrás (feedback) opera también en las células regulando el flujo a través de las rutas metabólicas. De este modo, los productos de una secuencia de reacciones enzimáticas cuando se acumulan regulan etapas tempranas de la ruta, actuando como inhibidores de las actividades de enzimas situadas al principio de la ruta, y reduciendo el flujo a través de ella. Es lo que se conoce como un bucle de retroalimentación.
Otro ejemplo de regulación es el que tiene lugar en los controles de edición que operan durante la síntesis de proteínas y la replicación de DNA. En estos casos, hay mecanismos de control que miden la fuerza de interacciones químicas. Así, durante la síntesis de proteínas, la interacción entre el codón en el mRNA y el anticodón en el tRNA es medida cuidadosamente y si la interacción es débil, porque el tRNA unido no es el correcto, este es rechazado. La regulación permite así que las reacciones químicas celulares puedan trabajar conjuntamente y generar un comportamiento que funciona con una lógica de eficiencia y economía celular.
Los ejemplos anteriores permiten entender el funcionamiento de la regulación en la vecindad de las reacciones químicas intracelulares pero además de una regulación local, se necesitan regulaciones de mayor alcance entre los diferentes microambientes y espacios intracelulares. Es necesario entonces, considerar mecanismos de señalización a distancia para asegurar la comunicación mientras se mantiene la separación. Se ha llegado a conocer, y nos hemos llegado a familiarizar con la existencia de las llamadas rutas de transducción de señal que forman parte de procesos de intercomunicación entre células, como la regulación hormonal, y también con procesos de señalización intracelular, menos conocidos, pero igualmente necesarios para que las actividades de la célula estén reguladas y coordinadas de manera apropiada. Hay también mecanismos de transporte que llevan sustancias químicas y componentes de un lugar a otro.
Por otra parte, del mismo modo que es necesario la señalización en el espacio hay también necesidad de una señalización en el tiempo, entre las diferentes etapas de la vida celular. Así, durante el ciclo celular, los sucesos que tienen lugar en el principio de la vida de una nueva célula son recordados y comunicados a otros sucesos más avanzados en el ciclo. Por ejemplo, si la replicación de DNA es incompleta, por errores en este proceso, es importante comunicar la situación a los mecanismos que llevan a cabo la siguiente etapa, la mitosis, para que la célula no intente dividirse hasta que se haya completado toda la replicación de DNA. Esto tiene lugar a través de vías de señalización. Se pueden encontrar ejemplos similares en los complejos procesos de diferenciación celular y el desarrollo de un organismo.
3. LA INFORMACIÓN EN LA BIOMOLÉCULAS Y SU TRANSFERENCIA EN LSO PROCESOS BIOQUÍMICOS
Como he mencionado anteriormente las células muestran una amplia variedad de comportamientos encaminados a un propósito, una característica de la vida que J. Monod, el científico y premio Nobel francés llamó “teleonomia”. Estas funciones incluyen la capacidad de comunicación para llevar a cabo lo homeostasis, la capacidad de adaptación a estímulos externos, la de reproducirse, y así sucesivamente. De modo, que una manera de considerar la organización biológica es considerarla como una organización que tiene un comportamiento dirigido esto es un propósito.
Esta idea trataría de explicar la organización biológica y las reacciones bioquímicas en términos de procesos lógicos e informativos que operan en las células a la manera a como lo hacen los circuitos en un ordenador.
Dos ejemplos ilustran esta idea, el significado de la estructura del DNA para la herencia y la regulación genética. En cuanto al primero, la doble hélice de DNA como descubrieron Watson y Crick está formada por dos cadenas complementarias de nucleótidos, y la asociación entre las cadenas depende de las reglas del apareamiento de las bases. Esta estructura de doble hélice es muy importante en los procesos de codificación de genes y de replicación del DNA. Sabiendo que los genes están constituidos por DNA y que contienen información, hay que destacar la capacidad de la secuencia de nucleótidos para almacenar información. Esta se encuentra en el orden y tipo de nucleótidos que constituyen la secuencia lineal de una cadena de DNA, de manera similar a cómo las letras forman las palabras y las frases. La secuencia de DNA de un gen es transcrita entonces en RNA, y esta a su vez, es traducida en la secuencia de aminoácidos de una proteína codificada por un gen.
La replicación de DNA tiene lugar por separación de las cadenas de nucleótidos utilizando las reglas del apareamiento de bases para construir nuevas cadenas complementarias. De este modo, el significado biológico que subyace en la capacidad codificadora y replicativa del DNA puede ser entendida en términos de información codificada en la estructura del DNA y el flujo de información que va de la secuencia del gen a la función de la proteína. La comprensión de la organización biológica que da lugar a la herencia viene así de transformar la bioquímica de estos procesos en cómo la información es comunicada y procesada.
El segundo ejemplo se refiere a la regulación genética. El conocimiento bioquímico de este proceso, ha conducido a la identificación y caracterización de proteínas represoras y activadoras que se unen a regiones específicas del DNA situadas antes y después del gen, o en su proximidad, y que conducen a cambios a nivel de la expresión del gen. Sin embargo, para generar el entendimiento biológico de este proceso, estas descripciones necesitan ser transformadas en estructuras lógicas, en los genes que son objeto de regulación. Un ejemplo de esto son los bucles de retroalimentación que regulan la transcripción genética, como mostraron Jacob y Monod en la regulación de los genes del operón lactosa en Escherichia coli.
Así pues, si la organización biológica puede ser entendida en términos de procesos lógicos e informativos generados por los mecanismos bioquímicos implicados en la replicación de DNA y en la expresión genética la siguiente cuestión es si esta nueva aproximación puede permitir entender otros aspectos de la biología celular. Se trataría entonces, de identificar los módulos lógicos e informativos que operan en las células en cada caso, y que derivan de los procesos bioquímicos, moleculares y biofísicos que tienen lugar en ellas.
Veamos el caso las rutas de señalización. La complejidad potencial de las rutas de señalización es considerable. Las conexiones entre las diferentes partes de una red pueden incluir bucles de retroalimentación positivos, negativos y mixtos que alimentan hacia delante y hacia atrás secuencias de señalización.
El sistema de control del ciclo celular por ejemplo contiene múltiples bucles de retroalimentación. Los llamados puntos de control del ciclo celular (checkpoints) se pueden considerar como un tipo de circuito de retroalimentación negativa para su regulación. Por ejemplo, en la mitosis, cuando se detectan cinetocoros, en el centrómero de los cromosomas, no unidos a microtúbulos se activa el sistema de regulación conocido como el control del ensamblaje del huso (SAC). Este punto de control detiene la progresión de la mitosis y permite a la célula un tiempo adicional para que los cinetocoros libres se unan a los microtúbulos que les correspondan. Por lo tanto, los cinetocoros desocupados activan una ruta de señalización que retroalimenta una secuencia de reacciones para disminuir el número de cinetocoros desocupados, es decir un circuito de retroalimentación negativa.
Los modelos de circuitos de retroalimentación positiva por otro lado pueden comportarse en una variedad de formas diferente. Un circuito de este tipo, puede amplificar señales en respuesta a una pequeña concentración de moléculas de entrada, dando lugar a una mayor concentración de moléculas de salida. Actúa entonces como un amplificador y convierte un pequeño cambio de entrada en un gran cambio de salida. La retroalimentación positiva permite también la generación de las denominadas ondas de actividad. Estudios en extractos de ovocitos de Xenopus han demostrado que el estado mitótico se propaga por ondas de actividad rápidas.
Las ondas de actividad pueden ser un mecanismo para coordinar eventos a grandes distancias. Ejemplos conocidos son los potenciales de acción en la transmisión del impulso nervioso y las ondas de calcio. En el sistema de señalización por ondas de Ca 2+ , los periodos de una señal oscilante, añaden una propiedad importante, la dinámica, una forma de canalizar más información que una simple señal de inicio o paro de una secuencia de reacciones.
En las células musculares la transmisión de señal puede ser conducida por una red de nanotubos de carbono semejante a lo que se encuentra en el microprocesador de un ordenador. De este modo, la información codificada en forma de moléculas cargadas es conducida a diversas partes de la célula de manera semejante a como la corriente eléctrica es conducida por circuitos en la placa base de un ordenador a las distintas partes que lo componen. Estas señales son responsables del comportamiento celular como cuando un músculo se relaja o se contrae. Lo más sorprendente, es que los circuitos celulares en este sistema, parecen ser más flexibles que los electrónicos y se reconfiguran para producir diferentes respuestas según la información recibida, constituyendo una verdadera “cell wide web”.
4. DE LA BIOLOGÍA MOLECULAS A LA BIOLOGÍA DE SISTEMAS
El gran desafío de la biología molecular es desentrañar la organización y las interacciones de las redes celulares que permiten procesos complejos. La complejidad surge de la existencia de interacciones no lineales y dinámicas entrelazadas entre un gran número de constituyentes celulares, como genes, proteínas, metabolitos, etc. El enfoque reduccionista ha permitido en muchos casos la identificación de la mayoría de los componentes e interacciones, pero no ofrece conceptos y métodos convincentes para comprender cómo pueden surgir las propiedades del sistema. Para comprender cómo y por qué las células funcionan de la forma en que lo hacen, se requieren datos cuantitativos sobre las concentraciones de los componentes para cuantificar los interacciones.
Una perspectiva completa de todo el sistema conocida como Biología de Sistemas es necesaria para que las propiedades de la red pueda entenderse cuantitativamente y ser incluso alterada a conveniencia.
Las claves en este nuevo paradigma están asociadas con avances en las llamadas tecnologías de alto rendimiento que caracterizan a las ómicas (genómica, proteómica, metabolómica, etc.).
Con respecto a la genómica, las técnicas de secuenciación han experimentado un desarrollo tal que se puede ahora llevar a cabo la secuenciación de genomas completos de manera totalmente automatizada. Los instrumentos de procesamiento combinados con capacidades de alto rendimiento están disponibles en muchos laboratorios de investigación. La secuenciación de un genoma de una célula se puede completar en horas o días. El análisis de los datos de secuenciación se ha visto facilitado con la ayuda de la bioinformática, que integra técnicas de diferentes disciplinas, como informática y matemáticas, para interpretar datos biológicos. La bioinformática no solo ayuda en la anotación el genoma sino que también proporciona información sobre las funciones de proteínas y homologías entre especies.
La información obtenida de la genética se puede entender con mayor precisión si damos un paso más allá al nivel transcripcional. Mediante la cuantificación del nivel de expresión de genes en diferentes condiciones, se han desarrollado matrices de ARN para facilitar la interpretación de la función del genoma y los patrones de regulación.
La técnica, llamada RNA-Seq permite la secuenciación directa de las transcripciones de ARN con resolución de una sola base. Más importante aún, proporciona una poderosa herramienta para comprender la dinámica transcriptómica al monitorizar los niveles de expresión génica.
Las proteínas por otra parte están presentes en todos los sistemas biológicos; su compleja estructura les permite realizar innumerables funciones, como transporte, catálisis, señalización y regulación. Los estudios experimentales de proteínas dependen en gran medida de tecnologías e instrumentación proteómica. Poderosas técnicas proteómicas pueden detectar pequeñas cantidades de una proteína específica y métodos de alto rendimiento como los que permiten analizar por ejemplo todas las fosfatasas y quinasas en el proteoma de E.coli.
Mientras que la Biología de Sistemas proporciona información sobre la estructura y función de proteínas naturales, la Biología sintética, conduce al diseño de proteínas que realizan funciones novedosas. Una aproximación es diseñar proteínas basadas en la modularidad. Las proteínas, se pueden desglosar en módulos: partes discretas de las mismas que realizan una función específica.
Los módulos pueden unirse a ligandos, transmitir información, catalizar una reacción o realizar otras muchas tareas. Por ejemplo, hay dominios de unión que median interacciones proteína-péptido mediante el reconocimiento de ciertas características peptídicas, como el dominio SH3 que reconoce secuencias ricas en prolina y el dominio PDZ que reconoce secuencias C-terminales.
La metabolómica por otro lado, se centra en el perfil y la dinámica de los metabolitos, y también revela la actividad de las reacciones enzimáticas celulares como las vías anabólicas y catabólicas.
Metodologías de identificación y cuantificación de metabolitos basadas en cromatografía de gases-espectrometría de masas (GC-MS), cromatografía de líquidos-espectrometría de masas (LC-MS) y resonancia magnética nuclear (RMN) se han desarrollado en las últimas décadas para estudiar perfiles y dinámicas de metabolitos Además, se han desarrollado herramientas de modelado metabólico, como el análisis de balance de flujo (FBA) y análisis de flujo metabólico (FMA) .
La combinación de metodologías analíticas metabólicas y herramientas de modelado ayuda a caracterizar redes metabólicas, identificar nuevas vías metabólicas y estudiar la respuestas de flujo metabólico a modificaciones genéticas o bajo diversas condiciones ambientales. Guiada por el conocimiento de la Biología de Sistemas, la Biología sintética tiene como objetivo diseñar la metabolómica, dirigiendo el flujo de metabolitos a rutas deseables. Esto incluye la construcción de nuevas rutas biosintéticas para la producción de moléculas e ingeniería de rutas de biodegradación para aplicaciones ambientales
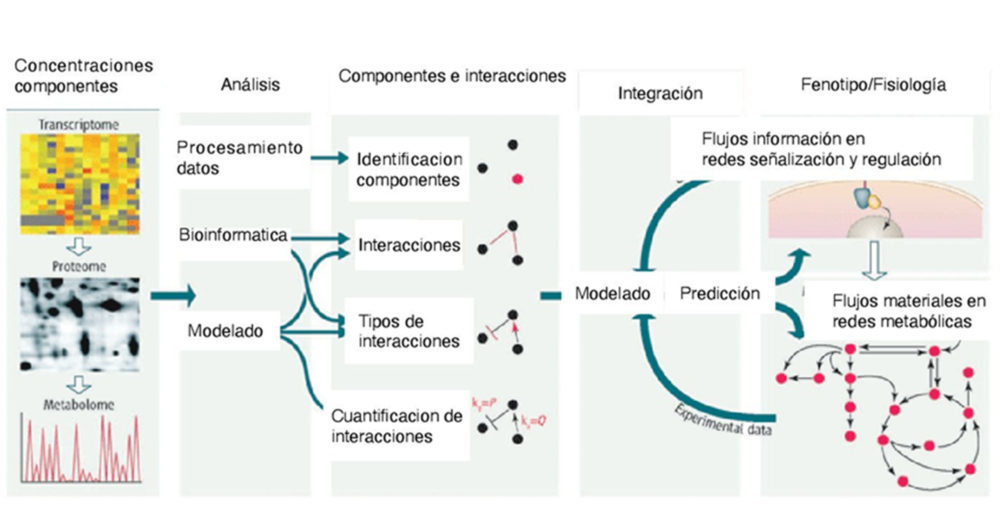
Mediante toda esta tecnología, la Biología molecular de Sistemas, opera mediante un conocimiento integral de las concentraciones de los componentes de un sistema para a partir de ellas inferir interacciones entre los componentes utilizando métodos computacionales . El objeto de los métodos de modelado computacional es predecir el estado de la red funcional a partir de las concentraciones e inferir los procesos de información que controla dicho estado (Fig.1).
Figura 1. Mapa de procesos en Biología de Sistemas
5. REFERENCIAS
- Allen G. Life science in the twentieth century. Cambridge: Cambridge
University Press,1978. - Judson, HF. The eighth day of creation. Makers of the revolution. New York Cold Spring Harbor Laboratory Press, 1996.
- Dressler D, Potter H. Discovering enzymes. New York: Scientific American Library, 1991.
- Alberts B, Bray D, Lewis J, Raff M et al. Molecular biology of the cell. New York: Garland, 2002.
- Monod J. Chance and necessity. London: Collins, 1972.
- Jacob F. The logic of living systems. A history of heredity. London: Penguin, 1974
- Nurse P. Understanding cells. Nature 2003; 424:883.
- Morgan, D.O. The Cell Cycle: Principles of Control. London UK: New Science Press Ltd; 2007.
- Ferrell JE Jr, Tsai TY, Yang Q. Modeling the cell cycle: why do certain circuits oscillate? Cell.2011; 144:874–885.
- Thomas R. On the relation between the logical structure of systems and their ability to generate multiple steady states or sustained oscillations. Springer Ser Synergetics. 1981; 9:180–193.
- Trunnell NB, Poon AC, Kim SY, Ferrell JE Jr. Ultrasensitivity in the regulation of Cdc25C by Cdk1. Mol Cell. 2011; 41:263–274.
- Duan J, Navarro-Dorado J, Clark J, Kinnear N, Meinke P. Schirmer E, Evans A. The cell-wide web coordinates cellular processes by directing site-specific Ca2+ flux across cytoplasmic nanocourses. Nature Comm. 10:2299 (2019)
- Sauer U.,Heineman M., Zamboni N. Getting closer to the whole picture. Science 2007,316, 550.