1. INTRODUCCIÓN
Desde hace varios años se conoce la relación entre las características ambientales y la conformación de la biodiversidad microbiana en los ecosistemas acuáticos, sobre todo en ecosistemas como las aguas de los manantiales mineromedicinales (1,2).
Se han realizado diversos estudios sobra la biodiversidad microbiana en el agua de manantiales mineromedicinal en varias partes del mundo, los cuales han señalado la presencia de una gran variedad de especies bacterianas en estos ecosistemas (3,4,5,6,7,8).
De igual forma, se ha venido observando que los ecosistemas acuáticos pueden albergar una microbiota bacteriana con resistencias naturales y adquiridas a los antibióticos, así como participar en la diseminación de los genes de resistencias a los antibióticos (9,10,11,12,13,14,15,16), lo cual ha permitido desarrollar el concepto de “Una Salud” o “One Health”, de manera de resaltar la importancia del medioambiente en los problemas de salud, como es el caso de la creciente resistencia y multirresistencia a los antimicrobianos por parte de las bacterias, situación que nos está afectando causando muchas muertes y que nos puede llevar a una próxima pandemia (17,18,19,20,21,22).
En este sentido, se ha acuñado el termino de resistomas ambientales, para describir la población microbiana resistente a los antibióticos (23). Los resistomas ambientales incluyen una gran variedad de especies bacterianas presentes en diferentes tipos de ecosistemas (24,25,26). Entre los géneros de bacterias encontrados cabe señalar a especies del género Staphylococcus, las cuales están presente principalmente en la superficie de animales y personas, pero también se les ha encontrado en los alimentos, el agua y el medioambiente (8,27,28,29,30,31,32).
Las bacterias del género Staphylococcus son células Gram positivas con forma de cocos dispuestas en racimos irregulares. Son anaerobios facultativos, catalasa positivos, inmóviles, fermentadores de azúcar, no formadores de esporas, capaces de crecer en altas concentraciones de NaCl y productores de pigmentos, que pueden variar de una especie a otra (33). La mayoría de los miembros del género Staphylococcus pueden sobrevivir en condiciones desfavorables. Son capaces de tolerar un amplio rango de temperaturas de 7 a 40 °C y baja actividad de agua, razón por la cual se encuentran ampliamente distribuidos y persisten en el medio ambiente, sobre todo el acuático (34).
Varias especies del género Staphylococcus son considerados patógenos de animales y humanos en donde pueden causar diversos tipos de infecciones y toxiinfecciones. La infección ocurre a menudo después del contacto con superficies contaminadas o por el consumo de alimentos y aguas contaminadas (28,35,36).
La resistencia a diversos grupos de antibióticos es otra característica resaltante de las especies de Staphylococcus aisladas principalmente de muestras clínicas animales y humanas, aunque también en muestras ambientales (37,38,39).
La mayoría de los estudios sobre la detección de genes de resistencia a antibióticos en especies del género Staphylococcus se han limitado a la especie Staphylococcus aureus, provenientes de muestras de alimentos y de muestras clínicas (40,41).
Entre las bacterias resistentes a los antimicrobianos, Staphylococcus aureus resistente a la meticilina (MRSA) es la segunda causa de morbilidad y mortalidad en todo el mundo (42).
Se han descrito altas tasas de resistencia a antibióticos como la oxacilina, penicilina, eritromicina, gentamicina y la vancomicina (40,43,44,45), lo cual ha llevado a que se señalen a cepas de Staphylococcus aureus, como una gran amenaza para la salud humana (37).
La incidencia de resistencia a los antibióticos en aislados acuáticos de especies de Staphylococcus se ha investigado muy poco, por lo que las implicaciones del contacto humano con aguas contaminadas con cepas de especies de Staphylococcus son desconocidas.
En Ecuador, las aguas mineromedicinales termales son frecuentemente utilizadas por un amplio sector de la población, quienes acuden a ellas con fines recreativos y en muchos casos con fines curativos. En este sentido, el objetivo del presente trabajo fue determinar la diversidad de especies de Staphylococcus y sus perfiles de resistencia a los antibióticos en aguas de balnearios mineromedicinales del Ecuador, de manera de conocer si existe en estos ecosistemas resistomas ambientales relacionados con las especies del género Staphylococcus que pudieran ser un riesgo para la salud del público que acude a estos sitios.
2. MATERIALES Y MÉTODOS
2.1. Materiales
2.1.1. Sitios de muestreos
Uno de los sitios donde se realizó parte de la investigación fue el balneario de aguas mineromedicinales de “Santagua de Chachimbiro” ubicado en el suroeste de la parroquia Tumbabiro, en el cantón Urcuqui, provincia Imbabura, en la Cordillera Occidental Andina ecuatoriana (Figura 1) a una altitud de 2672 m.s.n.m. y con una temperatura ambiente que varía entre los 16 y 22 °C. Las aguas termo minerales de Chachimbiro en el complejo Santagua, provienen de un acuífero hidrotermal profundo. El agua termal de este balneario se ha determinado que es de tipo clorurada sódica e hipertermal ya que la temperatura media del agua es de 55 °C (46).
Figura 1. Ubicación geográfica del Balneario “Santagua de Chachimbiro. Imbabura. Ecuador (Google Map, 2023).
Otro de los sitios donde se desarrolló el estudio fue en el balneario mineromedicinal de “Termas de Jamanco”, localizado al Nororiente de Ecuador, provincia de Napo, Cantón Quijos, parroquia Papallacta, Ecuador (Figura 2), a una altitud de 3400 m.s.n.m, con una temperatura ambiental promedio de 8 °C y una precipitación media anual mayor a 1000 mm. (47). Las aguas de este balneario han sido clasificadas de tipo cloruradas sódicas con temperaturas de 49,8 °C, consideradas como hipertermales (47).
Figura 2. Ubicación geográfica del Balneario “Termas de Jamanco”. Napo. Ecuador (Google Map, 2023).
El último de los sitios donde se llevó a cabo la investigación fue en el balneario de aguas mineromedicinales “Termas de Santa Ana”, ubicado en el cantón de Baños de Agua Santa de la Provincia de Tungurahua, Ecuador (Figura 3), a una altitud de 2031 m.s.n.m, con una temperatura ambiental promedio de 20 °C. Las aguas mineromedicinales termales del Balneario “Santa Ana”, son de tipo sulfatadas, magnésicas e hipertermales por tener una temperatura promedio de 35 °C (47).
Figura 3. Ubicación geográfica del Balneario “Termas de Santa Ana”. Tungurahua. Ecuador (Google Map, 2023).
2.1.2. Muestras
En el balneario de “Santagua de Chachimbiro” los puntos de la toma de muestra fueron cuatro. El primero, fue en la vertiente u ojo de agua. El segundo, en el tanque de almacenamiento. El tercer y cuarto punto fueron del agua de las piscinas termales del balneario. En todos los casos se tomó un volumen de 1 litro de muestras de agua, por duplicado en cada ocasión. Se realizaron dos muestreos en cada punto establecido, con un total de 16 muestras.
Para el balneario “Termas de Jamanco” los sitios de muestreos fueron 6. Las tres piscinas termales del complejo, el tanque de almacenamiento y las dos vertientes o nacientes de donde se toma el agua para el balneario. El volumen de muestra en cada sitio fue de 1 litro de muestras de agua, recolectadas por duplicado. Se realizaron dos muestreos, con un total de 24 muestras.
Respecto al balneario “Termas de Santa Ana”, se seleccionaron 3 sitios de muestreos, la naciente o vertiente del agua que ingresa al balneario, el tanque de almacenamiento del agua y la piscina termal del balneario. El volumen de muestra en cada sitio de muestreo fue de 1 litros de agua, recolectadas por duplicado. Se realizaron dos muestreos, con un total de 12 muestras.
En todos los casos los muestreos se realizaron de acuerdo con la norma ecuatoriana sobre muestras de agua (48), transportándose las muestras hasta el laboratorio de análisis según lo indicado en la normativa (49).
3. METODOLOGÍA
3.1. Siembra y aislamiento
Para el aislamiento de las colonias de Staphylococcus presentes en las muestras de agua, se utilizaron 2 técnica. La primera técnica fue la de siembra en profundidad (50) utilizando diluciones seriadas hasta 10-4 de cada una de las muestras en solución fisiológica, y posteriormente de cada una de las diluciones y de la muestra sin diluir, se realizaron siembras de un volumen de 1 mL en los agares Baird Parker y Manitol Salado, así como en placas Compact Dry X-SA para Staphylococcus, incubándose a 30 °C durante un tiempo de 48 a 72 horas. Posteriormente se purificaron las colonias crecidas en agar soya tripticasa para su posterior identificación.
La segunda técnica fue la de filtración por membrana (51). De cada una de las muestras recolectadas de agua se filtraron un volumen de 100 mL, utilizando filtros de 0,45 μm de diámetro y un tamaño de poro de 0,45 μm (Millipor). Posteriormente, las membranas se colocaron sobre placas de Petri con los agares Baird Parker y Manitol Salado, así como en placas de Compact Dry X-SA para Staphylococcus. Las placas con los filtros se incubaron a una temperatura de 30 °C durante 48-72 horas. Finalizado el tiempo de incubación las colonias amarillas fueron seleccionadas y purificadas en agar soya tripticasa para su posterior identificación.
3.2. Identificación de las colonias de Staphylococcus aisladas
Cada una de las colonias aisladas fueron purificadas en placas de Petri con agar soya tripticasa. Seguidamente se les realizó la coloración de Gram y la prueba de la oxidasa.
Las cepas presuntivas de Staphylococcus, purificadas en el agar Tripticasa-Soya, fueron sometidas a pruebas bioquímicas individuales, a fin de lograr su identificación a nivel de especie (52,53). De igual forma, se utilizó adicionalmente el método de identificación comercial de las galerías Novacyt-MicrogenTM para Staphylococcus (54).
3.3. Perfiles de resistencia a los antibióticos
La susceptibilidad a los antibióticos fue determinada a cada una de las cepas de Staphylococcus identificadas, a través del método de difusión en discos de Kirby-Bauer (55). Se utilizaron como cepas controles, la cepa ATCC 25922 de Escherichia coli y la cepa ATCC 25923 de Staphylococcus aureus. Se ensayaron los siguientes antibióticos: Eritromicina (ERY; 15 μg), gentamicina (GEN; 10 μg), oxacilina (OXA; 1 μg), penicilina (PEN; 10 U) y vancomicina (VAN; 30 μg).
Para la interpretación de los resultados de los antibiogramas se utilizó el manual del CLSI (56).
4. RESULTADOS
4.1. Diversidad de especies de Staphylococcus
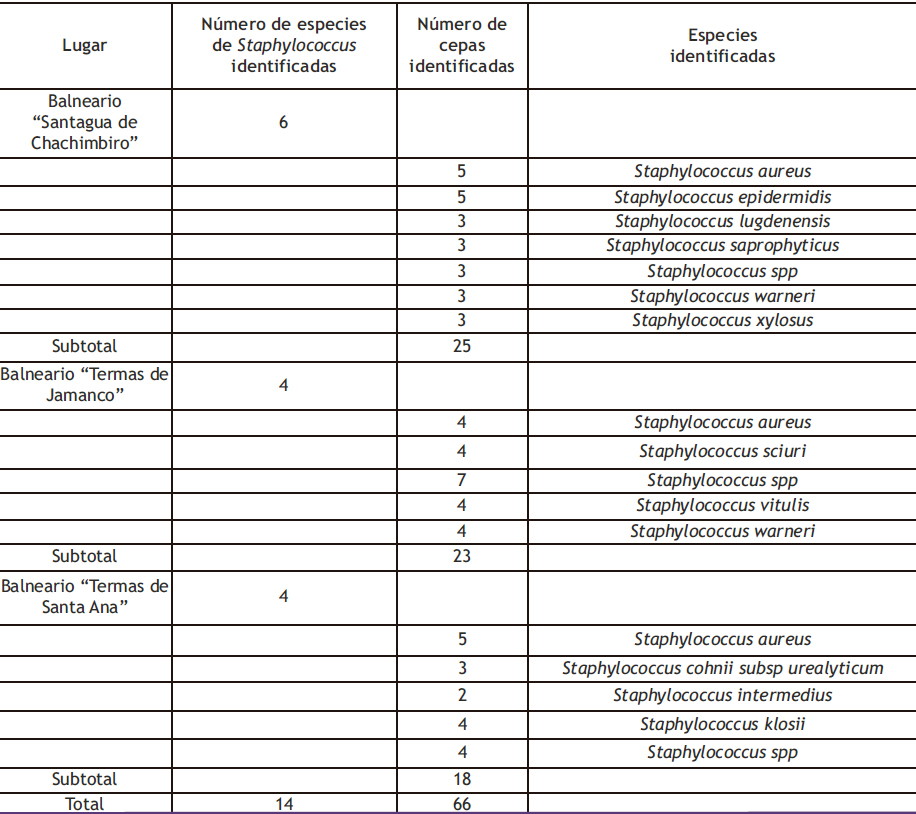
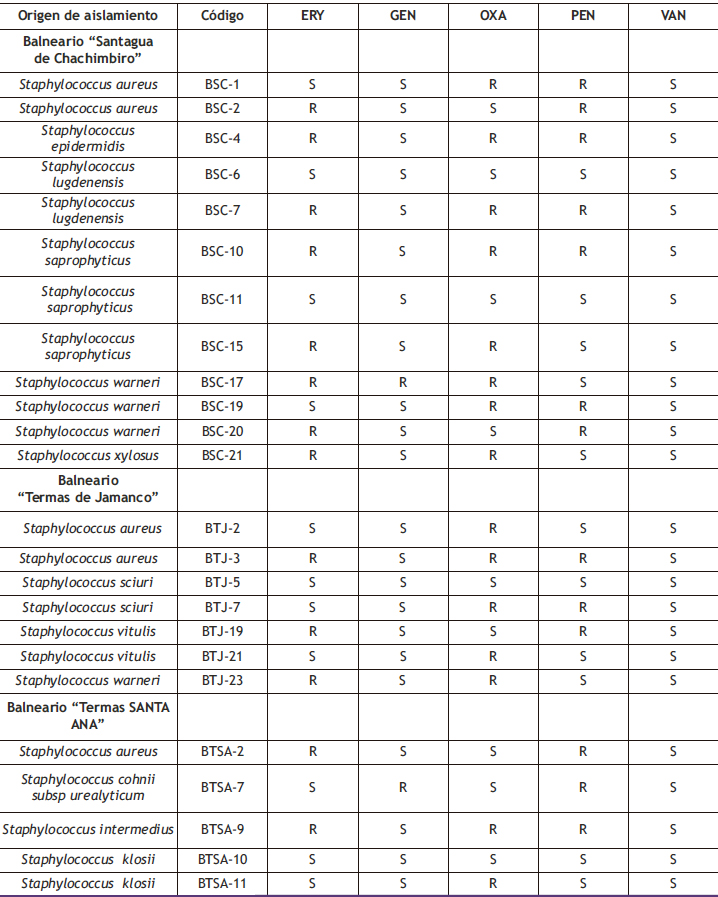
Al observar los datos señalados en la Tabla 1, se puede indicar que para el balneario de aguas mineromedicinales “Santagua de Chachimbiro” se logró identificar 25 cepas del género Staphylococcuspertenecientes a las especies: Staphylococcus aureus, Staphylococcus epidermidis, Staphylococcus lugdenensis, Staphylococcus saprophyticus, Staphylococcus spp Staphylococcus warneri y Staphylococcus xylosus (Ver Tabla 1).
Tabla 1. Diversidad de especies bacterianas del género Staphylococcus en aguas mineromedicinales de balnearios ecuatorianos
En el caso del agua del balneario mineromedicinal “Termas de Jamanco” se pudo identificar un total de 23 cepas del género Staphylococcus perteneciente a las especies, Staphylococcus aureus, Staphylococcus sciuri, Staphylococcus spp, Staphylococcus warneri y Staphylococcus vitulis (Ver Tabla 1).
Para el agua del balneario minero medicinal de “Termas de Santa Ana”, se pudo identificar una tola de 18 cepas del género Staphylococcus, correspondiendo a las especies Staphylococcus aureus, Staphylococcus cohnii subsp urealyticum, Staphylococcus intermedius, Staphylococcus klosii y Staphylococcus spp (Ver Tabla 1).
Al analizar los datos de la Tabla 1, se observa que las especies con mayor frecuencia de aislamiento, considerando los tres balnearios analizados, fueron Staphylococcus aureus (%), y Staphylococcus warneri (%).
4.2. Perfiles de resistencia a los antibióticos
En la Tabla 2 se resumen los resultados obtenidos en la determinación de los perfiles de resistencia y sensibilidad a los antibióticos de las especies del género Staphylococcus identificadas en los tres balnearios de agua mineromedicinal estudiados.
En la presente investigación se observaron las mayores resistencias para el caso de los antibióticos eritromicina, oxacilina y penicilina (Ver tabla 2).
Los antibióticos más efectivos contra las cepas de las diferentes especies de Staphylococcus, fue el antibióticos vancomicina, resultados que corresponde a la susceptibilidad que se ha indicado para este género en diferentes muestras de origen ambiental, animal y humano (Ver tabla 2).
Tabla 2. Perfiles de resistencia a los antibióticos de las especies bacterianas del género Staphylococcus aisladas en el agua mineromedicinales del balneario del Ecuador.
5. DISCUSIÓN
Desde hace varios años los estudios microbiológicos realizados en diferentes tipos de ecosistemas acuáticos han puesto en evidencia la presencia de cepas bacterianas pertenecientes a diversas especies del género Staphylococcusen estos ambientes, inclusive en las aguas minerales envasadas (3,27,34,57,58,59,60,61,62).
El género Staphylococcus alberga un gran número de especies cuyos miembros poseen un variado repertorio de características biológicas y bioquímicas que le han permitido sobrevivir y ocupar ambientes con diferentes condiciones fisicoquímicas y químicas (63,64,65).
Entre estos ambientes destaca los manantiales de aguas termales, denominados mineromedicinales por sus notorias propiedades y usos como medicamentos (66). Estudios microbiológicos realizados en manantiales mineromedicinales de varias partes del mundo han puesto en evidencia la presencia de especies de Staphylococcus en estos ecosistemas, denominado también ecosistemas extremófilos por sus condiciones extremas en el aspecto fisicoquímico y químico (1,3,4,5,8,67,68,69). En este sentido, los resultados del presente trabajo son similares respecto a la presencia de varias especies de este género (Ver tabla 1) en las aguas mineromedicinales estudiadas, aunque difieren en el aspecto de las especies identificadas y sus porcentajes de aislamientos.
De igual forma, en Ecuador existen varios estudios sobre el aislamiento y caracterización de este género bacteriano en muestras de aguas de balnearios mineromedicinales (24,70,71,72). Sin embargo, al comparar los resultados obtenidos en la presente investigación con los observados en otros balnearios de aguas mineromedicinales de otras provincias del Ecuador, se puede señalar que especies del género Staphylococcus se han aislados en estos tipos de ecosistemas, aunque existen variaciones en las especies identificadas y en la frecuencia de aislamiento, ratificando lo que han señalado diversos investigadores sobre la microbiota de los ecosistemas acuáticos, en el sentido de que es específica y que depende de las condiciones fisicoquímicas, geológicas y química de cada ecosistema (1,73).
En la mayoría de los estudios realizados en ecosistemas acuáticos mineromedicinales (3,4,53,74), las especies prevalente han sido Staphylococcus aureus y Staphylococcus epidermidis, resultados muy similares a los obtenidos en la presente investigación (Ver tabla 1), aunque la variedad de especies aisladas e identificadas ha sido mayor, muy probablemente debido a que estos ambiente son frecuentados por un gran número de personas que pueden ser portadoras de diferentes especies y cepas de Staphylococcus, las cuales pueden diseminarse en el agua mineromedicinal permaneciendo viables dada la resistencia de los miembros de este género a las condiciones de alta salinidad que presentan las aguas mineromedicinales, y además, por su capacidad de formar biofilm en diversos tipos de ecosistemas (15,75,76,77).
Los resultados respecto a los perfiles de sensibilidad y resistencia a los antibióticos por parte de las distintas cepas de las especies de Staphylococcus identificadas se resumen en la Tabla 2.
Las cepas de Staphylococcus presentan un perfil típico de susceptibilidad/resistencia a antibióticos (78,79), destacando la multirResistencia a 2 antibióticos en la mayoría de las cepas, así como la resistencia a los antibióticos eritromicina y a la oxacilina, además de la sensibilidad de todas las cepas analizadas a la vancomicina (Ver tabla 2)
Los resultados obtenidos en la presente investigación indican la presencia de una variada gama de perfiles de resistencia/sensibilidad a los antibióticos por parte de las cepas de las distintas especies de Staphylococcus analizadas, los cuales varían con respecto a otras investigaciones llevadas a cabo en otros tipos de ecosistemas acuáticos (12,80,81).
Los miembros del género Staphylococcus son bacterias ambientales ubicuas, capaces de colonizar rápidamente y causar infecciones oportunistas en humanos y animales, que también pueden de promover el intercambio de genes de resistencia a los antibióticos entre las bacterias autóctonas y las alóctonas presentes en los ambientes naturales, tales como los ecosistemas acuáticos (30,39).
Los resultados muestran que el mayor porcentaje de las cepas de las distintas especies de Staphylococcus fueron resistentes a los antibióticos eritromicina, oxacilina y penicilina (Ver tabla 2).
La penicilina es un antibiótico de la familia de los β-lactámicos. Se introdujo en la práctica clínica en 1941 y, sólo un año después, se describió la resistencia a la penicilina en algunas cepas de Staphylococcus aureus. Diez años después, más de la mitad de los aislamientos en grandes hospitales ya eran resistentes a la penicilina (82). La necesidad de crear compuestos resistentes a la acción de las β-lactamasas producidas por las células de Staphylococcus resistentes, llevó al desarrollo de otros antimicrobianos como la meticilina. Sin embargo, poco después de la introducción de este antibiótico, se aislaron también cepas resistentes al mismo (MRSA) (83). Actualmente, la Organización Mundial de la Salud (OMS) reconoce a las cepas de Staphylococcus resistentes a la meticilina como un patógeno de alta prioridad, ya que se ha observado la aparición de estas cepas en muestras alimentarias, ambientales, de animales y de humanos (37).
El mecanismo de resistencia a la meticilina se basa en la síntesis de la proteína PBP, con poca afinidad por la meticilina y otros β-lactámicos, que bloquea la llegada del antibiótico a su sitio diana y crea así resistencia (84). El gen mecA, que se encuentra en los loci genéticos llamados cromosomas en cassette estafilocócicos mec (SCCmec), es el principal gen responsable de la resistencia a la meticilina (85).
Con respecto al antibiótico eritromicina, el cual pertenece al grupo de los macrólidos de primera generación, pocos años después de su introducción en el tratamiento de infecciones por bacterias Gram positivas, aparecieron las primeras cepas de Staphylococcus resistentes a estos antibióticos. Hoy en día, la resistencia a los macrólidos está muy extendida en todo el mundo y un gran número de bacterias son resistentes a este tipo de antimicrobiano (86). La creciente resistencia a los macrólidos entre las cepas de Staphylococcus, sobre todo de origen clínico, es consecuencia de su uso en el tratamiento de infecciones por bacterias Gram positivas y generalmente se asocia con resistencia a los antibióticos lincosamida y estreptogramina B (87).
Los tres mecanismos principales de resistencia a los macrólidos en Staphylococcus son: (1) modificación del ribosoma bacteriano, (2) salida de macrólidos de la célula bacteriana/protección del ribosoma a través de proteínas de la familia de unión al ATP (ABC-F), e (3) inactivación enzimática (88,89). La modificación del sitio objetivo en el ribosoma causa una resistencia de amplio espectro a los macrólidos, mientras que el eflujo y la inactivación enzimática son de menos importancia. Los genes de resistencia a macrólidos se encuentran en plásmidos, transposones e islas genómicas y pueden transferirse fácilmente horizontalmente entre cepas y especies (88,89).
Otros de los resultados obtenidos en la investigación fue que una gran proporción de las cepas de Staphylococcus estudiadas resultaron sensibles al antibiótico gentamicina y todas fueron sensibles a la vancomicina (Ver tabla 2) .
La gentamicina es un antibiótico del grupo de los aminoglucósidos, utilizado como antibiótico de amplio espectro y acción bactericida para el tratamiento de infecciones causadas por bacterias Gram negativas y bacterias Gram positivas como Staphylococcus aureus o Enterococcus faecalis (90).
Se ha indicado que las bacterias del género Staphylococcus generan resistencia a través de diversos mecanismos, entre ellos las mutaciones en el sistema de permeabilidad membranal, a diferencia de las bacterias Gram negativas cuyo principal mecanismo de resistencia a la gentamicina es mediante la presencia de enzimas mediadas por plásmidos que alteran los aminoglucósidos (91).
En el caso de la vancomicina, el cual es un antibiótico del grupo de los glucopéptidos que se utiliza en casos de las cepas resistentes a la meticilina, se ha indicado que su efecto bactericida es a través de la inhibición de la síntesis de la pared celular, gracias a su gran afinidad por los precursores de esta, lo cual permite alterar la acción de la transglucosidasa por diversos impedimentos estéricos (90).
Recientemente se ha visto que la resistencia de bacterias Gram positivas a la vancomicina se encuentra asociada con la transferencia de genes van de enterococos (90). La resistencia a la vancomicina también puede deberse a la heterorresistencia de las células bacterianas (92).
Dado a que aún no se encuentra generalizada la resistencia a la gentamicina y vancomicina en las cepas de Staphylococcus, esto antibióticos se utilizan en los casos de las cepas resistentes a la meticilina (93), aunque en los últimos años ha ido aumentado la detección de cepas resistentes a los mismos (94).
En diversos estudios realizados en los últimos años en diferentes tipos de ecosistemas acuáticos, como glaciares, aguas de lagos volcánicos y los manantiales de aguas termales (16,74,95,96,97,98), han dejado al descubierto que en estos ambientes existen una diversidad de microorganismos con resistencias naturales a los antibióticos que han sobrevivido y se han adaptado a las condiciones extremas reinantes en esos ambientes, constituyendo un reservorio natural de bacterias y de genes asociados con la resistencia a los antibióticos denominados resistomas ambientales, los cuales han sido poco estudiados (10,99,100). Las escasas investigaciones realizadas sobre los resistomas ambientales han puesto en evidencia que antes de haberse descubierto y utilizados los antibióticos, las comunidades bacterianas han interactuado con los microorganismos productores de antibióticos y desarrollados mecanismos de resistencia que le han permitido su adaptación y proliferación en distintos ambientes acuáticos y terrestres, como una estrategia adaptativa creando nuevos nichos para sobrevivir ante la competencia con los microorganismos productores de sustancias antimicrobianas (101,102).
Tomando en consideración lo señalado en el párrafo anterior, es importante evaluar el problema de la resistencia a los antibióticos desde una perspectiva más global, como la que propone el enfoque “One Health” (103), donde el componente ambiental debe analizarse y monitorearse para estudiar las resistencias naturales detectadas, ya que representan una fuente importante de transmisión y dispersión, tanto de los microorganismos resistentes, como de sus genes (9,12,104). Recientemente se ha señalado que la resistencia a los antibióticos en cepas de bacterias ambientales se encuentra en la interfaz del “One Health” y que deben hacerse un mayor número de investigaciones para entender esta dinámica (11,105,106).
Los resultados obtenidos en el presente trabajo pueden ser el reflejo probablemente de la existencia de presiones selectivas naturales en los ecosistemas investigados, tales como la elevada concentración de sales y metales pesados presentes en este tipo de aguas, así como la temperaturas e incidencia de radiaciones solares elevadas, dada la situación altitudinal de los balnearios estudiados que se encuentran por encima de los 2000 metros sobre el nivel del mar. De igual forma, se debe considerar también como una presión selectiva, la posible presencia de microorganismos productores de antimicrobianos en estos ambientes, así como la presencia de especies de bacterias patógenas proveniente de fuentes externas que pudieran haber ayudado a la diseminación de los genes de resistencia, ello si se considera que estas aguas son utilizadas por una gran parte de la población que acude a ellas para buscar alivio a una variada gama de enfermedades.
En el caso de Ecuador, el presente trabajo sería el primero en señalar la presencia de especies del género Staphylococcus resistentes a diversos antibióticos en muestras de agua de manantiales mineromedicinales termales de las provincias de Imbabura, Napo y Tungurahua.
6. CONCLUSIONES
Se evidencia que en el agua mineromedicinal del balneario “Santagua de Chachimbiro”. están presentes una mayor variedad de especies de Staphylococcus en comparación a la observada en el agua de los otros balnearios estudiados.
Los resultados obtenidos en los perfiles de resistencia a los antibióticos de las distintas especies de Staphylococcus encontradas, indican la presencia de especies resistentes y multirresistentes a por lo menos 2 antimicrobianos, lo que evidencia el papel de las especies del género Staphylococcus en la diseminación y reservorios de los genes de resistencias a antibióticos en el ambiente y su posible papel en el establecimiento de resistomas ambientales.
Este trabajo señala la necesidad de estudiar y comprender la importancia de las aguas minerales naturales, como lo son las aguas mineromedicinales de uso público, como reservorios de la diversidad bacteriana y de resistomas ambientales siendo una línea base para futuras investigaciones desde el enfoque “One Health”, ya que es necesario tomar en cuenta la microbiota ambiental si se quiere combatir y mitigar el problema de la resistencia a los antibióticos.
Conflicto de Intereses
Los autores dejan constancias que no existe ningún tipo de conflicto de intereses con la investigación realizada y los resultados expresados en el presente artículo.
Agradecimientos
A la Dirección de Investigación de la Universidad Central del Ecuador por el financiamiento otorgado a través de los proyectos avanzados senior DI-011-2017 y DI-045-2022 que han permitido el desarrollo de una línea de investigación a la cual pertenece la presente investigación, así como a la Escuela Superior Politécnica del Chimborazo (ESPOCH), la Universidad Nacional del Chimborazo (UNACH), la Universidad Autónoma regional de los Andes (UNIANDES), la Universidad Politécnica Salesiana, la Universidad de los Andes de Venezuela (ULA) y la Universidad Complutense de Madrid, España por la ayuda, colaboración y asesoramiento prestado.
7. REFERENCIAS
- De la Rosa MC, Mosso MA. Diversidad microbiana de las aguas minerales termales. En: Panorama actual de las aguas minerales y minero-medicinales de España, pp. 153-158. Ed. A. López y J.L. Pinuaga. Instituto Tecnológico Geominero de España. Madrid. España. 2000.
- Barton L & McLean R. Enviromental Microbiology and Microbial ecology. First edition. Wiley & Sons. Inc. NJ. USA. 2019.
- De la Rosa MC, Andueza F, Sánchez M, Rodríguez C, Mosso M. Microbiología de las aguas mineromedicinales de los Balnearios de Jaraba. Anales de la Real Academia Nacional de Farmacia. 2004; 70 (Extraordinario): 51-74.
- De la Rosa MC, Fernández V, Pintado C, Rodríguez C. Microbiología del agua mineromedicinal del Balneario de Paracuellos de Jiloca. An. Real. Acad. Farm. 2018; 84 (Special Issue): 68‐80.
- Gutiérrez MG, Andueza F, Araque J, Lugo A, Chacón Z. Caracterización microbiológica y potencial biotecnológico de microorganismos aislados de las aguas termales de la Musuy, Municipio Rangel del Estado Mérida. Venezuela. Revista de la Sociedad Venezolana de Microbiología. 2018; 38 (1): 27-32.
- Valcheva N & Ignatov I. Physicochemical and microbiological characteristics of thermal healing spring waters in the district of Varna. Journal of Medicine, Physiology and Biophysics. 2019; 59: 10-16.
- Fang S & Yan J. Analysis of prokaryotic microbial diversity in hot spring water from Bantang (China) using the targeted amplicon analysis. All Life. 2022; 15(1): 325-339.
- Andueza-Leal FD, Araque-Rangel J, González-Escudero M, Sacoto D, León-Leal A, Gutiérrez MG, Flores S, Escobar-Arrieta S, & Medina-Ramírez G. Biodiversidad bacteriana en aguas de balnearios mineromedicinales de Ecuador y Venezuela. FIGEMPA: Investigación y Desarrollo, 2023; 15(1): 56-77.
- Baquero F, Martínez J, Cantón R. Antibiotics, and antibiotic resistance in water environments. Current Opinion in Biotechnology. 2008; 19 (3): 260-265.
- Ishii S. Ecology of Pathogens and Antibiotic-resistant Bacteria in Environments: Challenges and Opportunities. Microbes Environ. 2019; 34(1): 1-4.
- Rivera-Chavira BE, Palacios OA, Adame-Gallegos JR, Virginia G. La Resistencia Bacteriana a Antibióticos: Un Problema Ambiental y Multisectorial Microbial Antimicrobial Resistance: An Environmental and Multidisciplinary Problem. Revista Científica de la Universidad Autónoma de Coahuila. 2020; 12(24).
- Hu Y, Jiang L, Sun X, Wu J, Ma L, Zhou Y, Cui C. Risk assessment of antibiotic resistance genes in the drinking water system. Science of the Total Environment. 2021; 800: 149650.
- Zou HY, He LY, Gao FZ, Zhang M, Chen S, Wu DL, Ying GG. Antibiotic resistance genes in surface water and groundwater from mining affected environments. Science of the Total Environment. 2021; 772: 145516.
- Cohen T. The Next Pandemic: A Pragmatic and Ethical Discussion About the Looming Threat of Antibiotic Resistance. Voices in Bioethics. 2022; 8. https://doi.org/10.52214/vib.v8i.9509.
- Yin H, Cai Y, Li G, Wang W, Wong PK, An T. Persistence, and environmental geochemistry transformation of antibiotic-resistance bacteria/genes in water at the interface of natural minerals with light irradiation. Critical Reviews in Environmental Science and Technology. 2022; 52(13): 2270-2301.
- Andueza F, Acuña J, Escobar J, González-Escudero M, Araque J, Escobar-Arrieta S, González-Romero, Medina-Ramírez G. Pseudomonas species and their profiles of resistance to antibiotic in aquatic ecosystems of Ecuador. Revista de la Facultad de Farmacia. 2023; 65(1): 9-17.
- FAO-OIE-OMS-UNICEF-BM- UNSIC. Contributing to One World, One Health: a strategic framework for reducing risks of infectious diseases at the animal-human-ecosystems interface. Organización de las Naciones Unidas para la Agricultura y la Alimentación, la Organización Mundial de Sanidad Animal, la Organización Mundial de la Salud, el Fondo de las Naciones Unidas para la Infancia, el Banco Mundial y el Coordinador del Sistema de las Naciones Unidas para la Gripe. París. Francia. 2008. https://www.preventionweb.net/files/8627_OWOH14Oct08.pdf
- De Kraker MEA, Stewardson AJ, Harbarth S. ¿Will 10 million people die a year due to antimicrobial resistance by 2050? PLoS Med. 2016;13: e1002184.
- Leandro AS, Lopes RD, Martins CA, Rivas AV, da Silva I, Galvão SR, De Freitas RM. The adoption of the One Health approach to improve surveillance of venomous animal injury, vector-borne and zoonotic diseases in Foz do Iguaçu, Brazil. PLoS Negl Trop Dis 2021;15(2): e0009109. https://doi.org/10.1371/journal.pntd.0009109.
- Murray Christopher JL, Shunji Ikuta K, Sharara F, Swetschinski L, Robles Aguilar G, Gray A, Han C et al. Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. The Lancet. 2022; 399 (10325): 629 – 655.
- Ogunseitan OA. One Health and the Environment: From Conceptual Framework to Implementation Science, Environment: Science and Policy for Sustainable Development. 2022; 64:2, 11-21. DOI: https://doi.org/10.1080/00139157.2022.2021792
- Elnaiem A, Mohamed-Ahmed O, Zumla A, Mecaskey J, Charron N, Abakar MF & Dar O. Global and regional governance of One Health and implications for global health security. The Lancet; 2023; 401(10377): 688-704.
- Dante G, Sommer M. Genética de la resistencia microbiana. Investigación y Ciencias. 2014; Agosto: 28-34.
- Medina-Ramírez G, Naranjo C, Escobar S, Araque J, Djabayan P, Andueza F. Microbiota extremófila y resistomas ambientales de la fuente termal “Termas La Merced” Quito-Ecuador. FIGEMPA: Investigación y Desarrollo. 2016; 1(2): 33-37.
- Paun VI, Lavin P, Chifiriuc MC. & Purcarea C. First report on antibiotic resistance and antimicrobial activity of bacterial isolates from 13,000-year old cave ice core. Sci. Rep. 2021; 11: 514. https://doi.org/10.1038/s41598-020-79754-5.
- Hanna N, Tamhankar AJ & Lundborg CS. Antibiotic concentrations and antibiotic resistance in aquatic environments of the WHO Western Pacific and South-East Asia regions: a systematic review and probabilistic environmental hazard assessment. The Lancet Planetary Health. 2023; 7(1): e45-e54.
- Abdel-Gawad FK, Eweda WE, El-Taweel GE, Shehata SF, Abdel Tawab MA. Detection of Staphylococcus aureus from fish and water samples collected from Lake Qarun. International Journal of Scientific & Engineering Research. 2015; 6(8): 366-372.
- Haag AF, Fitzgerald JR, Penadés JR. Staphylococcus aureus in Animals. Microbiology Spectrum. 2019; 7(3): 7-3.
- Kozajda A, Jeżak K, Kapsa A. Airborne Staphylococcus aureus in different environments—a review. Environmental Science and Pollution Research. 2019; 26(34): 34741-34753.
- Zieliński W, Korzeniewska E, Harnisz M, Hubeny J, Buta M, Rolbiecki D. The prevalence of drug-resistant and virulent Staphylococcus spp. in a municipal wastewater treatment plant and their spread in the environment. Environment International. 2020; 143: 105914.
- Abdeen EE, Mousa WS, Abdelsalam SY, Heikal HS, Shawish RR, Nooruzzaman M, Abdeen A. Prevalence, and characterization of coagulase positive Staphylococci from food products and human specimens in Egypt. Antibiotics. 2021; 10(1): 75.
- Organji SR, Abulreesh HH, Elbanna K, Osman GE, Almalki MH. Diversity and characterization of Staphylococcus spp. in food and dairy products: a foodstuff safety assessment. Journal of Microbiology, Biotechnology and Food Sciences. 2021; 586-593.
- Bangieva D & Rusev V. Risk from Staphylococcus aureus in informally marketed raw cow milk. Food Sci Appl Biotechnol. 2019; 2:74–80.
- Faria C, Vaz-Moreira I, Serapicos E, Nunes OC, & Manaia CM. Antibiotic resistance in coagulase negative Staphylococci isolated from wastewater and drinking water. Science of the total environment. 2009; 407(12): 3876-3882.
- Cheung GY, Bae JS, Otto M. Pathogenicity, and virulence of Staphylococcus aureus. Virulence. 2021; 12(1): 547-569.
- Howden BP, Giulieri SG, Wong Fok Lung T, Baines SL, Sharkey LK, Lee JY & Stinear TP. (2023). Staphylococcus aureus host interactions and adaptation. Nature Reviews Microbiology. 2023; 1-16.
- World Health Organization (WHO). Global priority list of antibiotic-resistant bacteria to guide research, discovery, and development of new antibiotics. 2017. https://www.who.int/medicines/publications/WHO-PPL-Short_Summary_25Feb-ET_NM_WHO.pdf?ua=1
- Algammal AM, Hetta HF, Elkelish A, Alkhalifah DHH, Hozzein WN, Batiha GES, Mabrok MA. Methicillin-Resistant Staphylococcus aureus (MRSA): one health perspective approach to the bacterium epidemiology, virulence factors, antibiotic-resistance, and zoonotic impact. Infection and Drug Resistance. 2020; 13: 3255.
- Rossi CC, Pereira MF, Giambiagi de Marval M. Underrated Staphylococcus species and their role in antimicrobial resistance spreading. Genetics and Molecular Biology. 2020; 43.
- Idrees M, Sawant S, Karodia N, Rahman A. Staphylococcus aureus biofilm: Morphology, genetics, pathogenesis, and treatment strategies. International Journal of Environmental Research and Public Health. 2021; 18(14): 7602.
- Dos Santos IR, da Silva INM, de Oliveira Neto JR, de Oliveira NRL, de Sousa ARV, de Melo AM & Bailão EFLC. The presence of antibiotics and multidrug-resistant Staphylococcus aureus reservoir in a low-order stream spring in central Brazil. Brazilian Journal of Microbiology. 2023; 54(2): 997-1007.
- Antimicrobial Resistance Collaborators. (2022). Global burden of bacterial antimicrobial resistance in 2019: a systematic analysis. Lancet. 2022; 12;399(10325):629-655. Doi: https://doi.org/10.1016/S0140-6736(21)02724-0.
- Turner NA, Sharma-Kuinkel BK, Maskarinec SA, Eichenberger EM, Shah PP, Carugati M, Fowler VG. Methicillin-resistant Staphylococcus aureus: an overview of basic and clinical research. Nature Reviews Microbiology. 2019; 17(4): 203-218.
- Bruce SA, Smith JT, Mydosh JL, Ball J, Needle DB, Gibson R & Andam CP. Shared antibiotic resistance and virulence genes in Staphylococcus aureus from diverse animal hosts. Scientific Reports. 2022; 12(1): 4413.
- Selim S, Faried OA, Almuhayawi MS, Saleh FM, Sharaf M, El Nahhas N, & Warrad M. Incidence of vancomycin-resistant Staphylococcus aureus strains among patients with urinary tract infections. Antibiotics. 2022; 11(3): 408.
- Carrera Villacrés D & Guevara García P. Fuentes termales del Ecuador. Escuela Superior Politécnica del Ejercito (ESPE). Quito. Ecuador. 2017 http://repositorio.espe.edu.ec/xmlui/handle/21000/11739.
- Burbano N, Becerra S, Pasquel E. Aguas termo minerales en el Ecuador. INAMHI. Quito. Ecuador. 2013. https://issuu.com/inamhi/docs/termalismo
- Instituto Ecuatoriano de Normalización (INEN). NTE INEN 2 176-98 Agua. Calidad del agua. Muestreo. Técnicas de muestreo. Instituto Ecuatoriano de Normalización. Quito-Ecuador. 1998a.
- Instituto Ecuatoriano de Normalización (INEN). NTE INEN 2 169-98 Agua. Calidad del agua. Muestreo. Manejo y conservación de muestras. Instituto Ecuatoriano de Normalización. Quito-Ecuador. 1998b.
- Rice EW, Baird RB, Eaton AD. Standard methods for the examination of water and wastewater. 23 Edition. American Public Health Association. American Water Works Association. Water Environment Federation. Washington. USA. 2017.
- United Nations Environment Programe-World Health Organization (UNEP-WHO). Determination of Staphylococcus aureus in sea water and sewage by the membrane filtration (MF) culture method. Reference methods for marine pollution. Studie 28. Rev. I. UNEP-WHO. Athens. Greece. 1995.
- MacFaddin J. Pruebas Bioquímica individuales. Pruebas bioquímicas para la identificación de bacterias de importancia clínica. 3a ed. Editorial Médica Panamericana Buenos Aires. Argentina. 2003.
- Andueza F. Diversidad Microbiana de las Aguas Mineromedicinales de los Balnearios de Jaraba. Tesis Doctoral. Universidad Complutense de Madrid, España. 2007.
- Microgen Bioproducts. Microgen® Staph-ID. identification system for Staphylococci of clinical, veterinary, and environmental importance. UK. 2017.https://microgenbioproducts.com/wp-content/uploads/sites/8/2017/11/Microgen-Bioproducts-Flier-Staph-ID-LOW-REZ.pdf
- Bauer AW, Kirby MM, Sherris JC, Truck M. Antibiotic susceptibility testing by a standardized single disk method. Am. J. Clin. Pathol. 1966; 45(4): 493-496.
Clinical and Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptibility testing Clinical and Laboratory Standards Institute Wayne, PA. 32 edition. USA. 2022. - Meldrum R. Survey of Staphylococcus aureus contamination in a hospital’s spa hydrotherapy pools. Commun. Dis. Public Health. 2001; 4: 205-208.
- Andueza F. Calidad bacteriológica del agua mineral envasada expendida en la ciudad de Mérida, Venezuela. Perspectiva (Cajamarca). Facultad de Farmacia y Bioquímica. UPAGU. 2013; 14 (16): 41-49.
- Plano LRW, Shibata T, Garza AC, Jonathan Kish, Jay M. Fleisher, Christopher D. Sinigalliano, Maribeth L. Gidley, Kelly Withum, Samir M. Elmir, Suzanne Hower, Charlene R. Jackson, John B. Barrett, Timothy Cleary, Maureen Davidson, Johnnie Davis, Sampa Mukherjee, Lora E. Fleming & Helena M. Solo-Gabriele (2013). Human-associated methicillin-resistant Staphylococcus aureus from a subtropical recreational marine beach. Microb Ecol 2013; 65:1039–51.
- Araque J. Microbiota bacteriana heterótrofa aerobia mesófila del agua envasada. Tesis de Maestría. Postgrado de Microbiología. Facultad de Farmacia y Bioanálisis. Universidad de los Andes. Mérida. Venezuela. 2014.
- Gómez P., Lozano C., Benito D., Estepa V., Tenorio C., Zarazaga M., & Torres C. Characterization of Staphylococci in urban wastewater treatment plants in Spain, with detection of methicillin resistant Staphylococcus aureus ST398. Environmental pollution, 2016, 212, 71-76.
- Oladipo AO, Oladipo OG, Bezuidenhout CC. Multi-drug resistance traits of methicillin-resistant Staphylococcus aureus and other Staphylococcal species from clinical and environmental sources. J Water Health 2019; 17:930–43.
- Kloos WE. Natural Populations of the Genus Staphylococcus. Annual Review of Microbiology, 1980; 34:559-592.
- Götz F, Bannerman T, Schleifer KH. The Genera Staphylococcus and Macrococcus. The Prokaryotes. 2006:5–75. doi: https://doi.org/10.1007/0-387-30744-3_1
- Liebeke M & Lalk M. Staphylococcus aureus metabolic response to changing environmental conditions–a metabolomics perspective. International Journal of Medical Microbiology. 2014; 304(3-4), 222-229.
- Armijo M & San Martin J. Curas Balnearias y Climáticas. Talasoterapia y Helioterapia. Ed. Complutense. Madrid. España. 1994.
- Baker GC, Gaffar S, Cowan DA, Suharto AR. Bacterial community analysis of Indonesian hot springs. FEMS. Microbiol. Lett. 2001; 200: 103-109.
- Mosso MA, Sánchez M, De la Rosa MC. Microbiología del agua mineromedicinal de los balnearios de Alhama de Granada. An. R. Acad. Nac. Farm. 2002; 68: 381-405.
- Shahina J, Sandhiya D, Rafiq S. Bacteriological quality assessment of groundwater and surface water in Chennai. Nat Environ Pollut Technol 2020;19.
- Andueza F, Jácome A, Cortez S, Medina G, Arciniegas S, Parra Y, Araque J. Microbiota of the thermal water of the spa «Piscinas El Cachaco”, Calicali, Province of Pichincha, Ecuador. Anales de la Real Academia Nacional de Farmacia. 2018; 84(4): 247-254.
- Sacoto D & Andueza F. Microbiología del agua termal del balneario Ilalo. Pichincha. Ecuador. FIGEMPA: Investigación y desarrollo. 2020;1 (1): 18-25. https://doi.org/10.29166/revfig.v1i1.2178.
- Andueza Leal F, Araque J, Parra Y, Arciniegas S, Guailla R, Escobar S, Medina G. (2020). Diversidad bacteriana en aguas mineromedicinales del balneario “Urauco”. Pichincha. Ecuador. An. Real Acad. Farm. 2020; 86 (1): 9 – 18. http://dx.doi.org/10.15568/anranf.2020.89.or01
- Norton DC & LeChevallier WM. A pilot study of bacteriological population changes through potable water treatment and distribution. Appl Environ Microbiol. 2000; 66:268–76.
- Andueza F, González, Ibáza D, Vásquez P, Viteri F, Villacís L, Araque J, Escobar-Arrieta S, González-Romero A, Medina-Ramírez G, Álviarez E. Diversity and profiles of resistance to antibiotic in species of the genus Aeromonas isolated from the aquatic ecosystems of Ecuador. Anales de la Real Academia de Farmacia. 2022; 88 (4): 713-720.
- Vaish M, Price-Whelan A, Reyes-Robles T, Liu J, Jereen A, Christie S & Krulwich TA. Roles of Staphylococcus aureus Mnh1 and Mnh2 antiporters in salt tolerance, alkali tolerance, and pathogenesis. Journal of Bacteriology. 2018; 200(5), 10-1128.
- Schilcher K & Horswill AR. Staphylococcal biofilm development: structure, regulation, and treatment strategies. Microbiology and Molecular Biology Reviews. 2020; 84(3): 10-1128.
- François P, Schrenzel J, & Götz F. Biology and Regulation of Staphylococcal Biofilm. International Journal of Molecular Sciences. 2023; 24(6), 5218.
- Mamani E., Luján D., & Pajuelo G. Perfil de sensibilidad y resistencia de Staphylococcus aureus: Experiencia en el Hospital Nacional Hipólito Unanue. In Anales de la Facultad de Medicina, 2006; Vol. 67 (2), 120-124.
- Assouma F. F., Sina H., Dossou A. D., Socohou A., Hounsou M. C., Avogbe P. H., Boya B, Mousse W, Adjanohoun A, & Baba-Moussa, L. (2023). Antibiotic resistance profiling of pathogenic Staphylococcus species from urinary tract infection patients in Benin. BioMed Research International, 2023, 6364128. https://doi.org/10.1155/2023/6364128
- Abimbola Olumide Adekanmbi, Olubukola Funmi Soyoye, Olawale Olufemi Adelowo. Characterization of methicillin-resistance gene mecA in coagulase negative Staphylococci (CoNS) recovered from wastewater of two healthcare facilities in Niger. Gene Reports. 2019; 17: 100541.
- Mamman GP, Angulu CN, Musa G, & Angulu S. Identification and antibiotic susceptibility profile of methicillin and erythromycin resistant genes in clinical and environmental strains of Staphylococcus aureus in Minna Nigeria. Bayero Journal of Pure and Applied Sciences. 2022; 15(1): 195-201.
- Shlaes DM & Projan SJ. Antimicrobial resistance versus the discovery and development of new antimicrobials. In: Mayer’s DL (ed). BT—Antimicrobial Drug Resistance: Mechanisms of Drug Resistance. Totowa, NJ: Humana Press, 2009, 43–50.
- Stapleton PD & Taylor PW. Methicillin resistance in Staphylococcus aureus: mechanisms and modulation. Sci Prog. 2002; 85:57–72.
- Fishovitz J., Hermoso J. A., Chang M., & Mobashery S. (2014). Penicillin‐binding protein 2a of methicillin‐resistant Staphylococcus aureus. IUBMB life, 2014; 66(8), 572-577.
- Shore A. C., Deasy E. C., Slickers P., Brennan G., O’Connell B., Monecke S., Ehricht R, & Coleman, D. C. Detection of Staphylococcal cassette chromosome mec type XI carrying highly divergent mecA, mecI, mecR1, blaZ, and ccr genes in human clinical isolates of clonal complex 130 methicillin-resistant Staphylococcus aureus. Antimicrobial agents and chemotherapy, 2011, 55(8), 3765-3773.
- Van Hoek AHAM, Mevius D, Guerra B, Mullany P, Roberts AP, Aarts HJM. Acquired Antibiotic Resistance Genes: An Overview. Front. Microbiol. 2011; 2: 203.
- Yao W, Xu G, Li D, Bai B, Wang H, Cheng H, Zheng J, Sun X, Lin Z, Deng Q & Yu Z (2019). Staphylococcus aureus with an erm-mediated constitutive macrolide-lincosamide-streptogramin B resistance phenotype has reduced susceptibility to the new ketolide, solithromycin. BMC Infect. Dis. 2019; 19: 175.
- Feßler AT, Wang Y, Wu C, Schwarz S. Mobile macrolide resistance genes in Staphylococci. Plasmid. 2018; 99: 2–10.
- Miklasińska-Majdanik M. Mechanisms of Resistance to Macrolide Antibiotics among Staphylococcus aureus. Antibiotics 2021; 10: 1406. https://doi.org/10.3390/antibiotics10111406
- Rodríguez-Carranza (2015). Gentamicina: Antimicrobianos. In: Rodríguez Carranza R. eds. Vademécum Académico de Medicamentos. McGraw-Hill Education; 2015. Accessed mayo 12, 2024.https://accessmedicina.mhmedical.com/content.aspx?bookid=1552§ionid=90370876
- Mlynarczyk-Bonikowska B., Kowalewski C., Krolak-Ulinska A., & Marusza W. (2022). Molecular mechanisms of drug resistance in Staphylococcus aureus. International journal of molecular sciences, 2022, 23(15), 8088.
- Nunes A. P. F., Teixeira L. M., Iorio N. L. P., Bastos C. C. R., de Sousa Fonseca L., Souto-Padrón T., & Dos Santos K. R. N. (2006). Heterogeneous resistance to vancomycin in Staphylococcus epidermidis, Staphylococcus haemolyticus and Staphylococcus warneri clinical strains: characterisation of glycopeptide susceptibility profiles and cell wall thickening. International journal of antimicrobial agents, 2006; 27(4), 307-315.
- Hope R., Livermore D. M., Brick G., Lillie M., & Reynolds R. (2008). Non-susceptibility trends among Staphylococci from bacteraemias in the UK and Ireland, 2001–06. Journal of Antimicrobial Chemotherapy, 2008; 62 (suppl 2), ii65-ii74.
- Szemraj M., Glajzner P. & Sienkiewicz M. Decreased susceptibility to vancomycin and other mechanisms of resistance to antibiotics in Staphylococcus epidermidis as a therapeutic problem in hospital treatment. Sci Rep 2023, 13, 13629. https://doi.org/10.1038/s41598-023-40866-3
- Rafiq M, Hayat M, Anesio AM, Jamil SUU, Hassan N, Shah AA. Recovery of metallo-tolerant and antibiotic resistant psychrophilic bacteria from Siachen glacier, Pakistan. PLoS ONE, 2017; 12(7): e0178180. https://doi.org/10.1371/journal.pone.0178180
- González M, Viteri F, Villacis L, Escobar J, Araujo L, González A, Medina G, Araque J, Andueza F. Perfiles de susceptibilidad a los antibióticos en cepas del género Bacillus aisladas de ambientes acuáticos extremos del Ecuador. An. Real Acad. Farm. 2021; 87 (1): 27–34.
- Nawaz S., Rafiq M., Pepper I. L., Betancourt W. Q., Shah A. A., & Hasan F. Prevalence and abundance of antibiotic-resistant genes in culturable bacteria inhabiting a non-polar passu glacier, karakorum mountains range, Pakistan. World Journal of Microbiology and Biotechnology, 2023; 39(4), 94. https://doi.org/10.1007/s11274-023-03532-4
- Sajjad W., Ali B., Niu H., Ilahi N., Rafiq, M., Bahadur A. & Kang, S. High prevalence of antibiotic-resistant and metal-tolerant cultivable bacteria in remote glacier environment. Environmental Research, 2023; 239, 117444.
- Nimaichand S., Philip, J. S., Prasad, R., Nori, S. R. C., Divakaran, S. J., Ramaraj, S. K. & Nelson-Sathi, S. Shotgun Metagenomics Unravels the Bacterial Diversity, Resistome Profile and Genetic Exchange in a Tropical Urban Environment, 2023, Preprint (Version 1) available at Research Square. https://doi.org/10.21203/rs.3.rs-3169772/v1.
- Zhang Z. & Ju F. Future Climate Scenarios Aggravate Health Risks of Soil Microbiome by Reshaping Resistome and Pathogenome. bioRxiv, 2024, 2024-01.
- Letten AD, Hall AR, Levine JM. Using ecological coexistence theory to understand antibiotic resistance and microbial competition. Nat. Ecol. Evol. 2021; 5: 431–441 https://doi.org/10.1038/s41559-020-01385-w.
- Malard LA, & Guisan A. Into the microbial niche. Trends in Ecology & Evolution. 2023; https://doi.org/10.1016/j.tree.2023.04.015
- World Health Organization (WHO). A health perspective on the role of the environment in One Health Copenhagen: World Health Organization Regional Office for Europe. 2022.
- Bengtsson-Palme J., Abramova A., Berendonk T. U., Coelho L. P., Forslund S. K., Gschwind R. & Zahra R. Towards monitoring of antimicrobial resistance in the environment: For what reasons, how to implement it, and what are the data needs? Environment international, 2023, 108089.
- Aslam B., Khurshid M., Arshad M. I., Muzammil S., Rasool M., Yasmeen N. & Baloch, Z. Antibiotic resistance: one health one world outlook. Frontiers in cellular and infection microbiology, 2021, 11, 771510.
- Ahmad N., Joji R. M. & Shahid, M. Evolution and implementation of One Health to control the dissemination of antibiotic-resistant bacteria and resistance genes: A review. Frontiers in Cellular and Infection Microbiology, 2023, 12, 1065796.