Page 54 - 88_01
P. 54
ANALES
RANF
www.analesranf.com
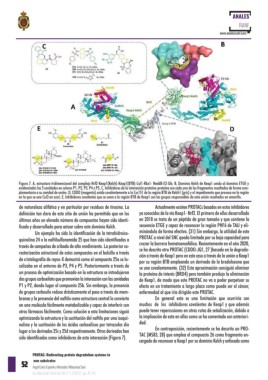
Figura 7. A, estructura tridimensional del complejo Nrf2-Keap1(Kelch)-Keap1(BTB)-Cul1-Rbx1- Nedd8-E2-Ub; B, Dominio Kelch de Keap1 unido al dominio ETGE y
evidenciados las 5 cavidades en colores P1, P2, P3, P4 y P5; C, Inhibidores de la interacción proteína-proteína con cada uno de los fragmentos resaltados de forma com-
plementaria a su cavidad de unión; D, CDDO (magenta) unido covalentemente a la Cys151 de la región BTB de Kelch1 (gris) y el impedimento que provoca en la región
en la que se une Cul3 en azul; E, Inhibidores covalentes que se unen a la región BTB de Keap1 con las grupos responsables de esta unión resaltados en amarillo.
de naturaleza alifática y en particular por residuos de tirosina. La Actualmente existen PROTACs basados en estos inhibidores
definición tan clara de este sitio de unión ha permitido que en los ya conocidos de la vía Keap1- Nrf2. El primero de ellos desarrollado
últimos años un elevado número de compuestos hayan sido identi- en 2018 se trata de un péptido de gran tamaño y que contiene la
ficado y desarrollado para actuar sobre este dominio Kelch. secuencia ETGE y capaz de reconocer la región PHF6 de TAU y eli-
minándola de forma efectiva. (31) Sin embargo, la utilidad de este
Un ejemplo ha sido la identificación de la tetrahidroiso- PROTAC a nivel del SNC queda limitada por su baja capacidad para
quinolina 24 o la naftilsulfonamida 25 que han sido identificadas a cruzar la barrera hematoencefálica. Recientemente en el año 2020,
través de campañas de cribado de alto rendimiento. La posterior ca- se ha descrito otro PROTAC (CDDO-JQ1, 27 )basado en la degrada-
racterización estructural de estos compuestos en el bolsillo a través ción a través de Keap1 pero en este caso a través de la unión a Keap1
de cristalografía de rayos-X demostró como el compuesto 25a se lo- por su región BTB empleando un derivado de la bradoloxona que
calizaba en el entorno de P3, P4 y P5. Posteriormente a través de se une covalentemente. (32) Esta aproximación consiguió eliminar
un proceso de optimización basado en la estructura se introdujeron la proteína de interés (BRD4) pero también produjo la eliminación
dos grupos carboxilato que promovían la interacción con las cavidades de Keap1, de modo que este PROTAC no va a poder perpetuar su
P1 y P2, dando lugar al compuesto 25b. Sin embargo, la presencia efecto en un tratamiento a largo plazo como puede ser el cáncer,
de grupos carboxilo reduce drásticamente el paso a través de mem- enfermedad al que iría dirigido este PROTAC.
branas y la presencia del naftilo como estructura central lo convierte
en una molécula fácilmente metabolizable y capaz de interferir con En general esta es una limitación que ocurriría con
otros fármacos fácilmente. Como solución a esta limitaciones siguió muchos de los inhibidores covalentes de Keap1 y que además
optimizando la estructura y la sustitución del naftilo por una isoqui- puede tener repercusiones en otras rutas de señalización, debido a
nolina y la sustitución de los ácidos carboxílicos por tetrazoles dio la implicación de esta en ellas como se ha comentado con anteriori-
lugar a los derivados 25c y 25d respectivamente. Otros derivados han dad.
sido identificados como inhibidores de esta interacción (Figura 7).
En contraposición, recientemente se ha descrito un PRO-
TAC (MS83, 28) que emplea el compuesto 26 como fragmento en-
cargado de reconocer a Keap1 por su dominio Kelch y enfocado como
PROTAC: Redirecting protein degradation systems to
52 new substrates
Ángel Cores Esperón y Mercedes Villacampa Sanz
An. Real Acad. Farm.Vol. 88. nº 1 (2022) · pp. 45-59