Page 70 - 73_01
P. 70
RAFAEL GIRALDO SUÁREZ AN. R. ACAD. NAC. FARM.
detenido ante la primera base de adenina del molde, al no haber sido
incluido en la reacción el UTP complementario.
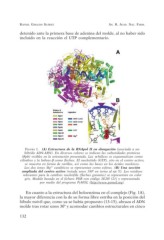
FIGURA 1. (A) Estructura de la RNApol II en elongación (asociada a un
híbrido ADN-ARN). En diversos colores se indican las subunidades proteicas
(Rpb) visibles en la orientación presentada. Las a-hélices se esquematizan como
cilindros y la hebras-ß como flechas. El nucleótido (GTP), sito en el centro activo,
se muestra en forma de varillas, así como las bases en los ácidos nucleicos.
Los dos iones Mg2+ catalíticos se representan como esferas. (B) Una sección
ampliada del centro activo (rotada unos 180o en torno al eje Y). Los residuos
relevantes para la catálisis nucleófila (flechas granates) se representan en color
gris. Modelo basado en el fichero PDB con código 2E2H (21) y representado
por medio del programa PyMOL (http://www.pymol.org).
En cuanto a la estructura del holoenzima en el complejo (Fig. 1A),
la mayor diferencia con la de su forma libre estriba en la posición del
lóbulo móvil que, como ya se había propuesto (13-15), abraza el ADN
molde tras rotar unos 30º y acomodar cambios estructurales en cinco
132