Page 30 - Demo
P. 30
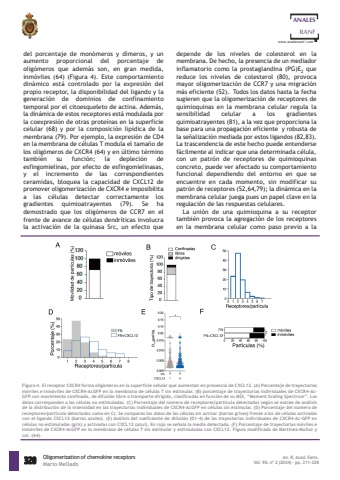
depende de los niveles de colesterol en lamembrana. De hecho, la presencia de un mediadorinflamatorio como la prostaglandina (PG)E2 quereduce los niveles de colesterol (80), provocamayor oligomerizaci%u00f3n de CCR7 y una migraci%u00f3nm%u00e1s eficiente (52). Todos los datos hasta la fechasugieren que la oligomerizaci%u00f3n de receptores dequimioquinas en la membrana celular regula lasensibilidad celular a los gradientesquimioatrayentes (81), a la vez que proporciona labase para una propagaci%u00f3n eficiente y robusta dela se%u00f1alizaci%u00f3n mediada por estos ligandos (82,83).La trascendencia de este hecho puede entendersef%u00e1cilmente al indicar que una determinada c%u00e9lula,con un patr%u00f3n de receptores de quimioquinasconcreto, puede ver afectado su comportamientofuncional dependiendo del entorno en que seencuentre en cada momento, sin modificar supatr%u00f3n de receptores (52,64,79); la din%u00e1mica en lamembrana celular juega pues un papel clave en laregulaci%u00f3n de las respuestas celulares. La uni%u00f3n de una quimioquina a su receptortambi%u00e9n provoca la agregaci%u00f3n de los receptoresen la membrana celular como paso previo a ladel porcentaje de mon%u00f3meros y d%u00edmeros, y unaumento proporcional del porcentaje deolig%u00f3meros que adem%u00e1s son, en gran medida,inm%u00f3viles (64) (Figura 4). Este comportamientodin%u00e1mico est%u00e1 controlado por la expresi%u00f3n delpropio receptor, la disponibilidad del ligando y lageneraci%u00f3n de dominios de confinamientotemporal por el citoesqueleto de actina. Adem%u00e1s,la din%u00e1mica de estos receptores est%u00e1 modulada porla coexpresi%u00f3n de otras prote%u00ednas en la superficiecelular (68) y por la composici%u00f3n lip%u00eddica de lamembrana (79). Por ejemplo, la expresi%u00f3n de CD4en la membrana de c%u00e9lulas T modula el tama%u00f1o delos olig%u00f3meros de CXCR4 (64) y en %u00faltimo t%u00e9rminotambi%u00e9n su funci%u00f3n; la depleci%u00f3n deesfingomielinas, por efecto de esfingomielinasas,y el incremento de las correspondientesceramidas, bloquea la capacidad de CXCL12 depromover oligomerizaci%u00f3n de CXCR4 e imposibilitaa las c%u00e9lulas detectar correctamente losgradientes quimioatrayentes (79). Se hademostrado que los olig%u00f3meros de CCR7 en elfrente de avance de c%u00e9lulas dendr%u00edticas involucrala activaci%u00f3n de la quinasa Src, un efecto que320ANALESRANFwww.analesranf.comOligomerization of chemokine receptorsMario Mellado An. R. Acad. Farm.Vol. 90. n%u00ba 2 (2024) %u00b7 pp. 311-328 Figura 4. El receptor CXCR4 forma olig%u00f3meros en la superficie celular que aumentan en presencia de CXCL12. (A) Porcentaje de trayectoriasm%u00f3viles e inm%u00f3viles de CXCR4-AcGFP en la membrana de c%u00e9lulas T sin estimular. (B) porcentaje de trayectorias individuales de CXCR4-AcGFP con movimiento confinado, de difusi%u00f3n libre o transporte dirigido, clasificadas en funci%u00f3n de su MSS, %u201cMoment Scaling Spectrum%u201d. Losdatos corresponden a las c%u00e9lulas no estimuladas. (C) Porcentaje del n%u00famero de receptores/part%u00edcula detectados seg%u00fan se extrae de an%u00e1lisisde la distribuci%u00f3n de la intensidad en las trayectorias individuales de CXCR4-AcGFP en c%u00e9lulas sin estimular. (D) Porcentaje del n%u00famero dereceptores/part%u00edcula detectados como en C). Se comparan los datos de las c%u00e9lulas sin activar (barras grises) frente a los de c%u00e9lulas activadascon el ligando CXCL12 (barras azules). (E) An%u00e1lisis del coeficiente de difusi%u00f3n (D1%u20134) de las trayectorias individuales de CXCR4-Ac-GFP enc%u00e9lulas no estimuladas (gris) y activadas con CXCL12 (azul). En rojo se se%u00f1ala la media detectada. (F) Porcentaje de trayectorias m%u00f3viles einm%u00f3viles de CXCR4-AcGFP en la membrana de c%u00e9lulas T sin estimular y estimuladas con CXCL12. Figura modificada de Mart%u00ednez-Mu%u00f1oz ycol. (64).