1. INTRODUCCIÓN
Los seres vivos reaccionamos e interactuamos con el entorno, recibiendo estímulos que procesamos y convertimos en respuestas para adaptarnos al mismo. Todas esas reacciones son fruto de la complejidad de células que se organizan en tejidos y órganos y que se comunican activando y reprimiendo armónicamente miles de moléculas. Si descendemos al mundo de la unidad biológica mínima de la vida, la célula, descubriremos de nuevo la complejidad de componentes, orgánulos, moléculas, reacciones químicas que de manera coordinada en tiempo y espacio permiten a las células responder a los estímulos y realizar sus funciones, dividirse, modular su metabolismo, promover o reprimir su expresión génica, organizar su citoesqueleto o incluso moverse. Desde un mar de células en división, la coordinación temporal y espacial del movimiento de células y moléculas permite la generación de un embrión (1), la formación de un tubo neural, los procesos de mitosis (2), la modificación de la forma celular (3) o el cerramiento de heridas (4) entre otras muchas funciones. Sin embargo, quizás sea el sistema inmunológico el paradigma del movimiento celular. Su funcionamiento requiere del continuo tráfico leucocitario en labor de patrulla a través del torrente circulatorio y los diferentes órganos y tejidos. Esta capacidad permite extender la respuesta inmunológica por todo el organismo. Así garantiza una respuesta ubicua, contra agentes extraños procedentes del exterior y frente a células del propio organismo que han sufrido alguna alteración patológica (5).
2. EL MOVIMIENTOS CELULAR ES CLAVE PARA LA FUNCIÓN DEL SISTEMA INMUNOLÓGICO
Las células del sistema inmunológico se generan en la médula ósea de los organismos adultos y deben salir a circulación periférica para alojarse en los órganos linfoides secundarios, bazo, ganglios linfáticos, estructuras linfoides asociadas a mucosas, donde tendrá lugar el inicio de la respuesta inmunológica (6,7). Dentro de los órganos linfáticos secundarios, los leucocitos siguen en continuo movimiento. Por ejemplo, las células B, en los folículos linfoides de los ganglios linfáticos, se mueven para facilitar la captura de antígenos que en forma soluble o presentados por macrófagos, les permitirá activarse. Las células T y las células dendríticas en los ganglios, gracias a su movimiento constante aumentan las posibilidades de encontrarse y así hacen posible que un antígeno presentado por una célula presentadora de antígeno sea reconocido específicamente por el TCR de una célula T. Se establece entonces una sinapsis inmunológica que será responsable de la activación y proliferación de la célula T, generando clones de células hijas con una especificidad de antígeno determinada (8).
Las células ya activadas que reconocen un determinado antígeno, salen a circulación periférica y se distribuyen por el organismo. Sin embargo, los vasos sanguíneos son estructuras cerradas y en su mayor parte no son permeables a la salida de leucocitos. En consecuencia, cuando en un tejido existe una infección, las diversas poblaciones leucocitarias necesitan ser reclutadas desde el torrente circulatorio hacia el foco inflamatorio; es el proceso de extravasación y permite la infiltración tisular de las células del sistema inmunológico de forma específica, ordenada y secuencial (9,10) para luchar eficazmente contra la infección.
La extravasación celular es un proceso complejo y en él participan de forma orquestada muchas proteínas, selectinas y sus ligandos, integrinas, moléculas de adhesión, quimioquinas y sus receptores, entre otras. Del correcto entendimiento entre ellas y de la sucesión temporal de eventos se consigue la salida ordenada de los leucocitos, y su llegada en tiempo y espacio adecuado al tejido donde la respuesta inmunológica es necesaria (10). Es importante además considerar que el movimiento no se limita a un mero desplazamiento en dos dimensiones, sino que las células sufren cambios estructurales para conseguir avanzar y superar las barreras que en forma de matriz extracelular y de otras células presentes en el entorno dificultan su avance (11–13).
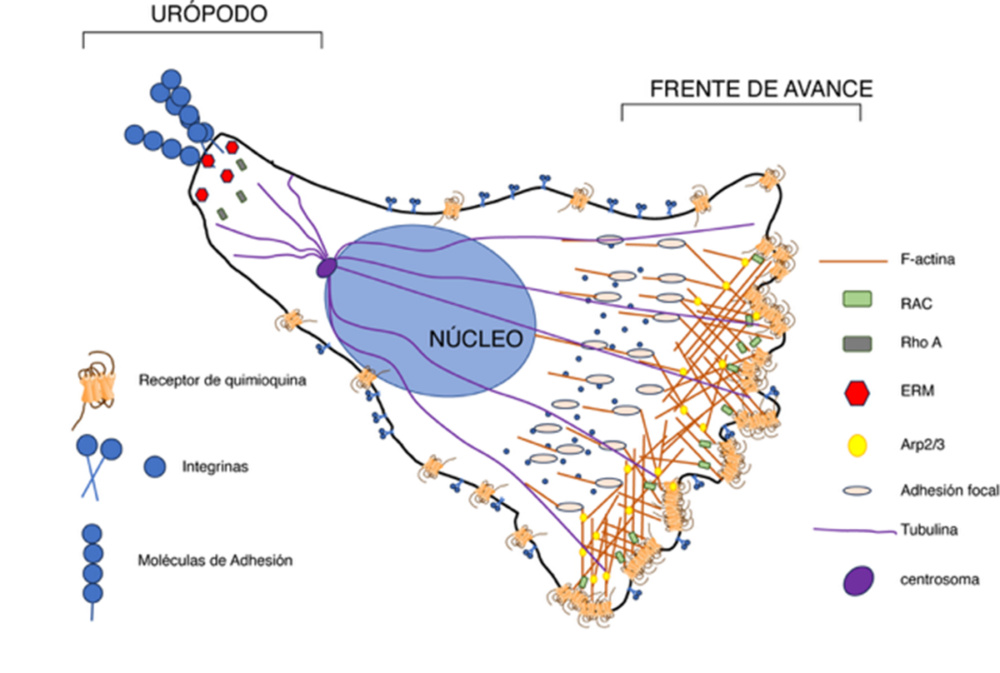
La señalización generada por la combinación de la unión de integrinas y sus ligandos, las moléculas de adhesión, y la exposición a quimioquinas inmovilizadas por glicosaminoglicanos sobre la superficie de las células endoteliales, induce cambios en la morfología de los leucocitos. Esto provoca una distribución asimétrica del contenido celular (14), originando un eje antero-posterior en la célula. Los leucocitos extienden un frente rico en actina polimerizada, F-actina, denominado lamelipodio, el cual constituye el frente de avance, mientras que en el extremo opuesto, se forma el urópodo, una zona más delgada que se eleva sobre la célula endotelial. En el urópodo se acumulan moléculas de adhesión como ICAM1, ICAM2 o CD44, que facilitan la interacción con otras células y que además interaccionan con las proteínas ERM (erzina, ranexina y monensina), organizando así toda la red de microtúbulos y filamentos intermedios implicados en los procesos de retracción de la célula durante la migración (15). Mientras, en el lamelipodio se redistribuyen los receptores de quimioquinas actuando como una “nariz celular” para detectar el gradiente quimioatrayente y dirigir el movimiento. En el lamelipodio también se acumulan moléculas señalizadoras que posibilitan la polimerización de actina dependiente de GTPasas, Rac, RhoA y cdc42 (16). Todo ello se traduce finalmente en la generación de un acúmulo asimétrico de lípidos fosforilados y de F-actina que permite una polarización celular estable y orientada a la trayectoria (Figura 1).
Figura 1. Polarización durante la migración de células T. Representación esquemática de la polarización de las células T en migración, que implica la reorganización orquestada del citoesqueleto de actina y de la red de microtúbulos. En la parte frontal de la célula, frente de avance, la estimulación por quimioquinas induce la polimerización de la actina, proceso en el que participa la GTPasa Rac-1 y el complejo efector Arp2/3 que conduce a la ramificación necesaria para la extensión del lamelipodio. Los receptores de quimioquinas de la zona del frente de avance se organizan formando oligómeros lo que facilita el reconocimiento del sentido del gradiente quimioatrayente. Se produce también, mediado por las quimioquinas, la activación de las integrinas que se unen a las moléculas de adhesión presentes en el sustrato formando adhesiones focales. En la parte posterior, la fosforilación dependiente de RhoA de las proteínas ERM induce su segregación selectiva al urópodo, donde reclutan moléculas de adhesión transmembrana. En el proceso, la red de tubulina facilita la polarización de los orgánulos celulares y la generación de fuerzas de tracción que posibilitan el movimiento. Figura modificada de las publicadas por Mastrogiovanni y col. (100) y Kenific y col. (101).
3. QUIMIOQUINAS: MOLÉCULAS CLAVES EN LA GENERACIÓN DE GRADIENTES QUIMIOATRAYENTES
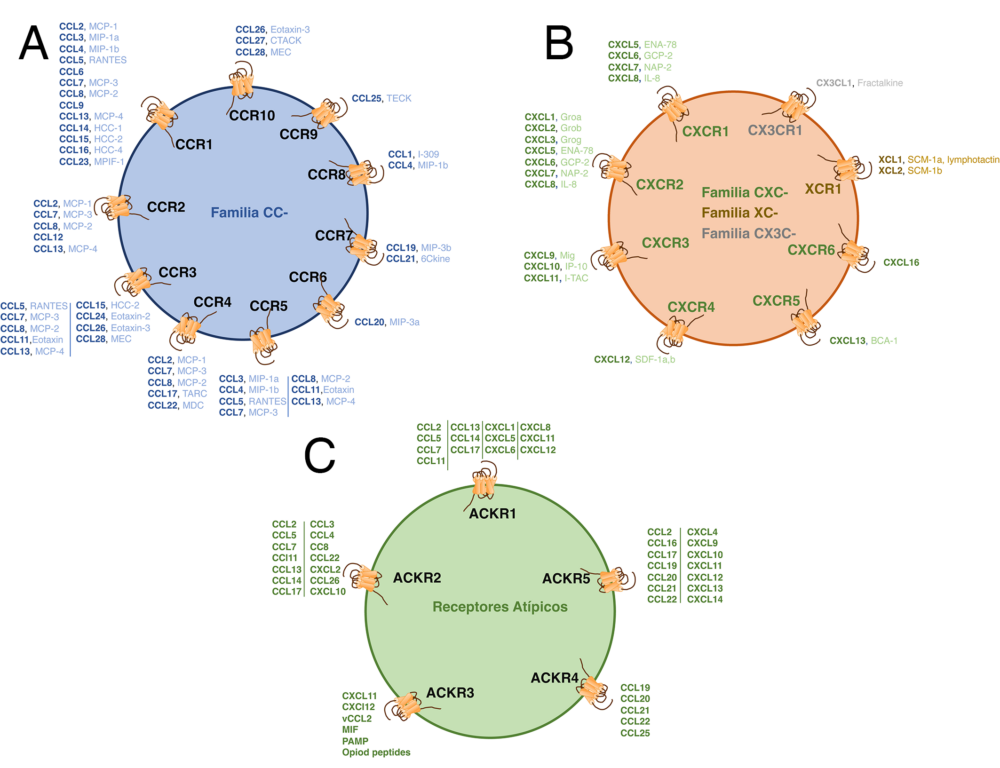
Las citoquinas quimioatrayentes o quimioquinas son una familia de proteínas de bajo peso molecular (7-12 kDa), con un alto grado de homología, que originalmente fueron identificadas por su capacidad de atraer poblaciones leucocitarias específicas (17). Estructuralmente, las aproximadamente 50 quimioquinas descubiertas hasta la fecha en humanos, son proteínas muy relacionadas entre sí que en un principio fueron clasificadas con criterios estructurales, en base a la posición de cuatro residuos de cisteína altamente conservados, en quimioquinas CC, CXC, C y CX3C (18). Sin embargo, en la actualidad la clasificación más extendida atiende al tipo de proceso biológico en el que participan, lo que está directamente relacionado con que su expresión sea constitutiva o inducible (19). Las primeras juegan un papel relevante en la homeostasis del sistema inmunológico, entre ellas cabe destacar CXCL12, CCL19, o CCL21. El segundo grupo se relaciona con procesos inflamatorios, entre ellas las quimioquinas CCL2, CCL3, CCL5 o CXCL8, por ejemplo.
Las quimioquinas ejercen sus acciones por unión a receptores presentes en la membrana de la célula diana. Estos receptores, aproximadamente 20 en humanos, pertenecen a la familia de receptores de siete dominios transmembrana acoplados a proteínas G, GPCRs (G Protein-Coupled Receptors) (Figura 2A, B). Constan de una única cadena polipeptídica que cruza siete veces la membrana celular e inician su ruta de señalización activando proteínas G, proteínas que unen nucleótidos de guanina (20), mayoritariamente Gi aunque también se han asociado a proteínas Gq y G11/12 (21).
Atendiendo al ligando que unen, los receptores se clasifican en cuatro grupos denominados CCR, CXCR, CX3CR y XCR. El sistema es complejo pues existe una alta promiscuidad entre las quimioquinas y los receptores que las unen, de hecho, una quimioquina puede unirse a distintos receptores con similar afinidad (CCL5, por ejemplo, une CCR1, CCR3 y CCR5), y un mismo receptor puede interaccionar con varias quimioquinas como es el caso de CCR2 que interacciona con CCL2, CCL7, CCL8 y CCL13. Sin embargo, también existen pares ligando/receptor altamente específicos como es el caso de CXCL12 y su receptor CXCR4 (22) (Figura 2A, B). La complejidad es mayor si consideramos que una misma célula puede expresar sobre su superficie a lo largo de su vida o incluso simultáneamente más de un receptor (23). El resultado final es una compleja red de interacciones que gobierna el correcto funcionamiento del sistema. Además, existen receptores denominados atípicos capaces de unir quimioquinas de las familias CC y CXC y que no activan una proteína G, su señalización está ligada a la activación de b-arrestinas y no inducen migración celular, aunque si son internalizados en respuesta al ligando (Figura 2C). Su función se relaciona con el control de los niveles de quimioquinas en el entorno extracelular (24) y con la modulación de la función de los receptores quimioatrayentes (25,26). Por último, algunos virus codifican receptores de quimioquinas (citomegalovirus (CMV) o Herpesvirus saimirii (HHV8)) e incluso quimioquinas (i.e. Moluscum contagiosum) (27). El significado biológico de estos receptores y ligandos codificados por virus está relacionado con la manipulación del sistema inmunológico durante la infección viral de las células huésped.
Tomando como ejemplo la unión de CXCL12 a su receptor CXCR4, se ha propuesto que la unión del ligando sigue un modelo en dos pasos (28,29). Primero se produce la interacción del cuerpo central de la quimioquina con el N-terminal de CXCR4, es el “Chemokine Recognition Site 1” (30) (CRS1). Esta interacción hace que el ligando se posicione en una orientación óptima sobre el receptor y permite que su región N-terminal penetre profundamente en el receptor y se una a la región CRS2, “Chemokine Recognition Site 2”. Este CRS2 se localiza en el interior del barril que forman las siete a-hélices transmembranas incluidas en la bicapa lipídica. La estructura de la quimioquina puede variar si hablamos de monómeros o de dímeros de ligando y también depende de la concentración de ligando en el medio. Además, se han descrito dímeros no disociables de CXCL12 que se unen a CXCR4, inducen la movilización de Ca2+, pero no median la migración celular in vitro (31), lo que sugiere funciones específicas de los ligandos asociadas a la estabilización de distintas conformaciones del receptor. En todo caso, la unión del ligando a los receptores promueve cambios conformacionales en los dominios transmembrana de estos, que son transmitidos por una cadena de residuos “señalizadores” presentes a lo largo de las a-hélices transmembrana (30) y cuya función es conectar los cambios conformacionales iniciados por la unión del ligando con la unión y activación de moléculas señalizadoras, como la proteína G, con los bucles intracelulares y región C-terminal del receptor.
La mayoría de las quimioquinas, son de carácter básico, lo que explica que se unan tanto a los dominios extracelulares de los receptores, como a glucosaminoglicanos (GAGs) presentes en la superficie del endotelio (32). De hecho, los GAGs actúan a modo de moléculas presentadoras, ya que poseen un elevado número de residuos ácidos. La interacción quimioquina-GAGs, facilita la retención de las quimioquinas en la superficie celular y además produce el efecto de aumentar su concentración local, de manera que, por ejemplo, permanezcan asociadas en presencia de corrientes provocadas por el flujo sanguíneo en los capilares, facilitando de ese modo su reconocimiento por los leucocitos (33).
Figura 2. Las redes de señalización de quimioquinas humanas son muy promiscuas. Hay 23 receptores y aproximadamente 50 ligandos en la red de señalización de quimioquinas humanas, incluidos diez miembros de la familia CCR (A), seis CXCR (B), un CX3CR (B) y un XCR (B). Las quimioquinas CCL se muestran en tonos azules, CXCL en tonos verdes, CX3CL en gris y XCL en marrón. En todos los casos en negrilla se expresa la nueva nomenclatura de las quimioquinas y en tono normal la antigua denominación. Además hay cinco AKCR caracterizados (C) que regulan los niveles extracelulares de algunas quimioquinas y modulan la función de los receptores de quimioquinas convencionales. Figura modificada de Bogacka y col. (102).
4. LOS RECEPTORES DE QUIMIOQUINAS ADOPTAN DISTINTAS CONFORMACIONES EN LA MEMBRANA CELULAR
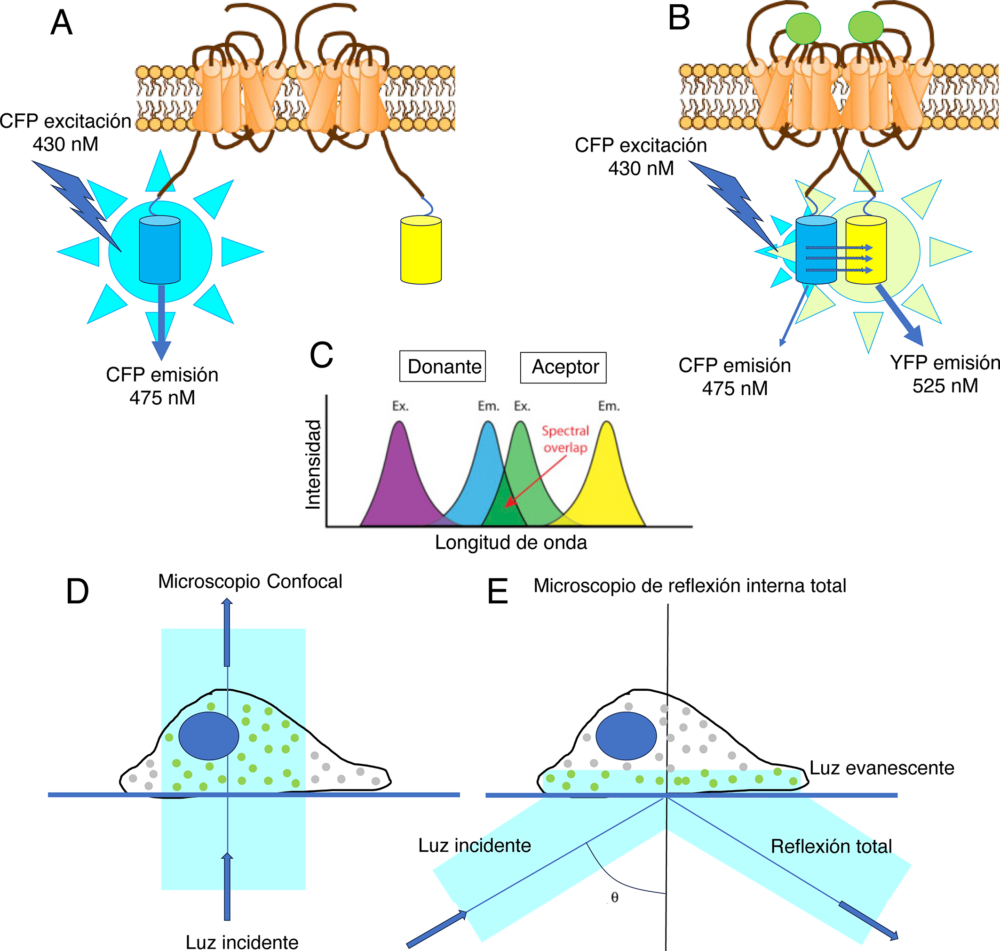
Los métodos más tradicionales para el estudio de la función de las quimioquinas (34,35) han generado una visión demasiado estática de los sistemas. Debemos recordar que el movimiento celular es un proceso dinámico que se realiza a favor de gradiente de concentración de moléculas quimioatrayentes y las células deben decidir en fracciones de segundo sobre la presencia de diferentes gradientes para continuar avanzando. Además, es un proceso influido por muchos factores de la propia célula y de su entorno. En la actualidad el uso de técnicas de microscopía óptica avanzada nos proporciona una visión mucho más dinámica que nos permite abordar y analizar con otra perspectiva todos estos procesos (36). Por ejemplo, la transferencia de energía resonante (RET), basada en fluorescencia (FRET) o en luminiscencia (BRET), que tiene su fundamento en que una molécula (fluorescente o luminiscente) excitada ceda energía a otra que se encuentra suficientemente cerca siempre que pueda excitarse con la longitud de onda de la energía emitida por la primera, nos permite detectar y evaluar las interacciones proteína-proteína o proteína lípido en células vivas de manera dinámica (37,38) (Figura 3A-C). Gracias a ello, hoy sabemos que los receptores de quimioquinas además de monómeros forman dímeros en la membrana celular; homodímeros si son complejos formados por el mismo receptor o heterodímeros si los complejos los forman receptores diferentes (39). Algunos autores han indicado que estos complejos se forman durante la síntesis y maduración de los receptores en el aparato de Golgi y así alcanzan la membrana celular (40). Gracias a estas tecnologías de imagen sabemos también que las conformaciones de estos receptores no son estáticas, sino muy dinámicas, modulándose por la presencia de los ligandos y la expresión de los propios receptores (41). Este concepto dinámico concuerda con el modelo que explica la función de los GPCRs, según el cual las conformaciones del receptor sin ligando están en equilibrio entre estados activos e inactivos y que es la unión del ligando quien estabiliza una conformación activa, desplazando el equilibrio (42,43). Los dímeros de receptores de quimioquinas existen en ausencia de ligando y algunos estudios muestran que los valores de eficiencia de FRET y BRET no están afectados por los ligandos específicos, sugiriendo que las conformaciones de los receptores no son moduladas por la unión del ligando (44). Por ejemplo, se ha descrito que CCL2 no promueve cambios conformacionales en los heterodímeros CXCR4/CCR2 (45). Sin embargo, otros estudios si detectan variaciones en la eficiencia FRET o BRET tras la adición del ligando. Es el caso de CXCL12 que aumenta la eficiencia de BRET entre homodímeros CXCR4 (45) o CXCL8 que suprime la eficiencia FRET para los heterodímeros CXCR1/CXCR2 mientras que estabiliza los valores de FRET para ambas formas homodiméricas (41). Los ligandos no desencadenan la dimerización del receptor pero parecen estabilizar la conformación activa del mismo, como se ha descrito para CCL2 y CCL11 en CCR2 (41,46).
Muchos autores han demostrado el impacto de estos complejos diméricos en las funciones de las quimioquinas (47–51). Hauser y col. asociaron los tetrámeros de CCR7 con la activación de Src y la integración de distintas vías de señalización necesarias para una migración celular eficaz (52). La dimerización/oligomerización de los receptores de quimioquinas afecta a diferentes aspectos de la fisiología del receptor, como la transducción de señales o los procesos de internalización mediados por ligando, que a su vez influyen en procesos como la activación y migración celular (53). Los complejos de receptores se acumulan en estructuras celulares especializadas, como el frente de avance de una célula en migración o la sinapsis inmunológica (54–56). Por lo tanto, es posible que la agregación de receptores facilite, en lugares específicos de la célula, el acoplamiento eficiente del receptor con moléculas de señalización (57) o incluso la modulación de la actividad de otros receptores también presentes (58).
Otra observación interesante es la diversidad farmacológica asociada a estos complejos de receptores. Mientras que AMD3100, un antagonista específico de CXCR4, bloquea la unión de ligandos específicos de otro receptor como CCR2, TAK779, el antagonista de CCR2 y CCR5 (59), inhibe potentemente la unión de CXCL12 en células que coexpresan CXCR4 (60), proporcionando un excelente ejemplo de inhibición alostérica entre heterodímeros de receptores de quimioquinas (55).
Figura 3. Esquema de funcionamiento de técnicas para evaluar conformaciones de receptores: FRET Y TIRF-M. (A-C) Tecnología FRET: (A, B) La transferencia de energía resonante entre fluorocromos se basa en el hecho de que una molécula fluorescente donante (por ejemplo, “Cyan Fluorescent protein, CFP”) en un estado excitado puede transferir una parte de su energía por acoplamiento dipolo-dipolo no radiante a una molécula fluorescente aceptora (por ejemplo “Yellow fluorescent protein, YFP”) si la distancia entre ellas es menor de 10 A, indicando entonces que las proteínas acopladas a los fluorocromos interaccionan. La tecnología implica la fusión de proteínas fluorescentes donantes y aceptoras a moléculas de interés. La coexpresión de constructos de fusión en células vivas permite estudiar su interacción en tiempo real de manera cuantitativa. (C) Es imprescindible que exista solapamiento entre la longitud de onda a la que emite el fluorocromo dador y la que necesita el aceptor para excitarse. Tecnología TIRF (D,E): (D) Los microscopios tradicionales de campo amplio iluminan toda la profundidad de una muestra. (E) La microscopía de fluorescencia de reflexión interna total (TIRF) se utiliza para adquirir imágenes de una sección muy fina con alta resolución. Para ello se utiliza un rayo láser que se coloca con precisión en el plano focal posterior de los objetivos. De este modo se crea un ángulo ideal para la reflexión interna total del haz láser, de modo que también pueda ser recogido por la lente del objetivo. Por lo tanto, el rayo láser nunca se transmite a través de una muestra durante el TIRF, y en su lugar sólo una pequeña área de aproximadamente 200 nm del cubreobjetos es iluminada por un campo evanescente. Este campo evanescente está causado por la diferencia de índices de refracción entre la muestra y el cubreobjetos de vidrio sobre el que se asienta, y es responsable de la elevada relación señal-ruido de las imágenes TIRF.
5. OLIGOMERACIÓN DE RECEPTORES DE QUIMIOQUINAS: UN PASO MÁS EN LA COMPLEJIDAD DE ESTOS MEDIADORES INFLAMATORIOS
Otra tecnología de imagen óptica avanzada es la microscopia de reflexión interna total de la fluorescencia, del inglés “Total internal reflection fluorescence microscopy” (TIRF-M). El TIRF-M permite la visualización y el análisis de moléculas individuales en células vivas y es por lo tanto de máximo interés para estudiar la dinámica de proteínas y lípidos en la membrana celular. Se utiliza la observación de la interfaz (61,62) entre dos medios con diferentes índices de difracción, como son el agua y el vidrio. La técnica depende de la iluminación de la muestra con un ángulo de incidencia tal que permita la reflexión total de la luz y la consecuente creación de un campo electromagnético, denominado “campo evanescente”, que excita los fluoróforos cerca de la interfaz mientras el resto de la célula permanece en oscuridad. Como resultado, el TIRF-M reduce la profundidad de excitación a unos 50-100 nm en la célula, lo que permite analizar los parámetros dinámicos y cinéticos de las proteínas y lípidos en la interfaz célula-sustrato, es decir en el contexto de la membrana plasmática de una célula viva en contacto con un sustrato depositado sobre el cristal (Figura 3D, E). El uso de esta tecnología aplicado al campo de las quimioquinas ha revelado que los receptores evaluados hasta la fecha pueden formar homo- y heterodímeros, pero también oligómeros de mayor orden (52,63,64). La caracterización in silico de la estructura de CXCR4 no sólo confirma la existencia de dímeros de receptores, sino que también nos muestra que existen interacciones entre dímeros de CXCR4 en complejos de mayor orden (65,66) y localiza en las regiones transmembrana los dominios implicados en estas interacciones.
En 1972, basado en principios termodinámicos de organización de lípidos y proteínas, se definió el modelo mosaico fluídico de la estructura de las membranas celulares (67). Se trata de un modelo dinámico y que explica que los componentes de la membrana están en continuo movimiento debido sobre todo a energías térmicas. El modelo incluye que los componentes proteicos y lipídicos están limitados en su movilidad rotacional y lateral en el plano de la membrana, lo que influye en las interacciones lípido-lípido, proteína-proteína y lípido-proteína, algo que también se ve afectado por las interacciones célula-matriz extracelular o célula-célula. El citoesqueleto celular resulta crítico en la dinámica de las membranas (68,69) pues el equilibrio polimerización/ despolimerización de actina crea regiones donde las proteínas pueden estar temporalmente confinadas y permite que proteínas que tienen una interacción directa con la actina puedan dificultar la difusión de otras proteínas que se mueven en el entorno (70). También influye la formación de dominios lipídicos específicos fuertemente empaquetados que condicionan el movimiento de las proteínas embebidas en ellos (71,72).
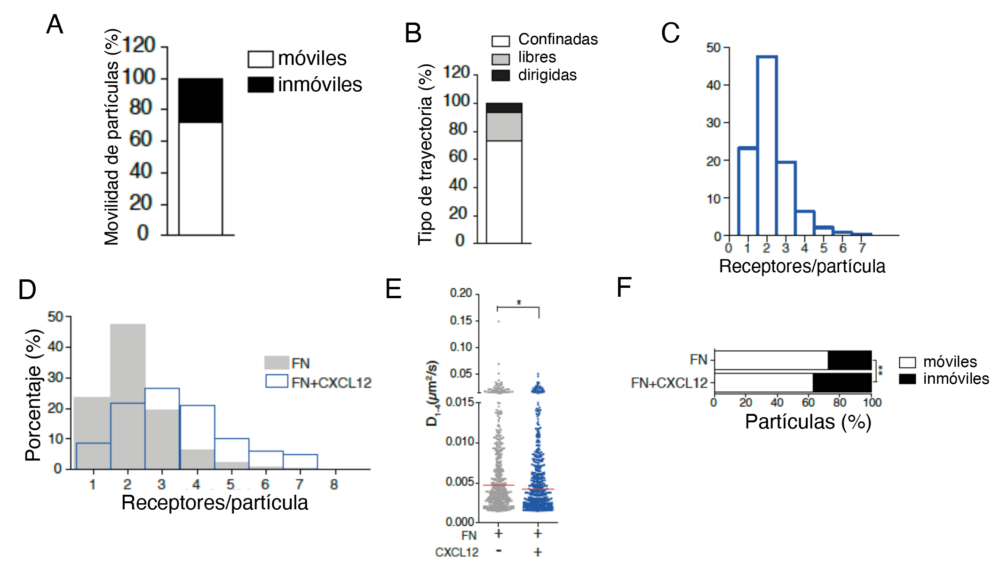
De hecho muchos receptores, también los miembros de la familia de GPCRs, se integran en esos dominios lipídicos densamente empaquetados. Este es el caso de los receptores muscarínicos M1 y M2 (73), del receptor de bradiquinina (74), del receptor b-adrenérgico (75), del receptor de Angiotensina 1 (76) y también de algunos de receptores de quimioquinas como CCR5 y CXCR4 (77). Aunque el término balsa lipídica del inglés “lipid raft” es controvertido pues su descripción se basa en el uso de técnicas de fraccionamiento de membranas y en la resistencia al tratamiento con detergentes de lípidos y proteínas, la existencia de interacciones lípido-lípido que se organizan lateralmente en las membranas como dominios con diferente estructura y composición está fuera de toda duda (78). En este contexto, los experimentos de TIRF-M han demostrado que en situación basal, sin estimular, los receptores de quimioquinas se organizan en entidades no agrupadas (monómeros y dímeros) y en pequeños nanoclusters (grupos de ≥3 receptores) y que la unión del ligando promueve la agrupación de los receptores en complejos de mayor tamaño, que contienen hasta 10-18 receptores, con una reducción significativa del porcentaje de monómeros y dímeros, y un aumento proporcional del porcentaje de oligómeros que además son, en gran medida, inmóviles (64) (Figura 4). Este comportamiento dinámico está controlado por la expresión del propio receptor, la disponibilidad del ligando y la generación de dominios de confinamiento temporal por el citoesqueleto de actina. Además, la dinámica de estos receptores está modulada por la coexpresión de otras proteínas en la superficie celular (68) y por la composición lipídica de la membrana (79). Por ejemplo, la expresión de CD4 en la membrana de células T modula el tamaño de los oligómeros de CXCR4 (64) y en último término también su función; la depleción de esfingomielinas, por efecto de esfingomielinasas, y el incremento de las correspondientes ceramidas, bloquea la capacidad de CXCL12 de promover oligomerización de CXCR4 e imposibilita a las células detectar correctamente los gradientes quimioatrayentes (79). Se ha demostrado que los oligómeros de CCR7 en el frente de avance de células dendríticas involucra la activación de la quinasa Src, un efecto que depende de los niveles de colesterol en la membrana. De hecho, la presencia de un mediador inflamatorio como la prostaglandina (PG)E2 que reduce los niveles de colesterol (80), provoca mayor oligomerización de CCR7 y una migración más eficiente (52). Todos los datos hasta la fecha sugieren que la oligomerización de receptores de quimioquinas en la membrana celular regula la sensibilidad celular a los gradientes quimioatrayentes (81), a la vez que proporciona la base para una propagación eficiente y robusta de la señalización mediada por estos ligandos (82,83). La trascendencia de este hecho puede entenderse fácilmente al indicar que una determinada célula, con un patrón de receptores de quimioquinas concreto, puede ver afectado su comportamiento funcional dependiendo del entorno en que se encuentre en cada momento, sin modificar su patrón de receptores (52,64,79); la dinámica en la membrana celular juega pues un papel clave en la regulación de las respuestas celulares.
Figura 4. El receptor CXCR4 forma oligómeros en la superficie celular que aumentan en presencia de CXCL12. (A) Porcentaje de trayectorias móviles e inmóviles de CXCR4-AcGFP en la membrana de células T sin estimular. (B) porcentaje de trayectorias individuales de CXCR4-Ac-GFP con movimiento confinado, de difusión libre o transporte dirigido, clasificadas en función de su MSS, “Moment Scaling Spectrum”. Los datos corresponden a las células no estimuladas. (C) Porcentaje del número de receptores/partícula detectados según se extrae de análisis de la distribución de la intensidad en las trayectorias individuales de CXCR4-AcGFP en células sin estimular. (D) Porcentaje del número de receptores/partícula detectados como en C). Se comparan los datos de las células sin activar (barras grises) frente a los de células activadas con el ligando CXCL12 (barras azules). (E) Análisis del coeficiente de difusión (D1–4) de las trayectorias individuales de CXCR4-Ac-GFP en células no estimuladas (gris) y activadas con CXCL12 (azul). En rojo se señala la media detectada. (F) Porcentaje de trayectorias móviles e inmóviles de CXCR4-AcGFP en la membrana de células T sin estimular y estimuladas con CXCL12. Figura modificada de Martínez-Muñoz y col. (64).
La unión de una quimioquina a su receptor también provoca la agregación de los receptores en la membrana celular como paso previo a la generación de vesículas de internalización ricas en clatrina, un proceso que involucra a dinamina y a otras proteínas adaptadoras como b-arrestinas y adaptina-2 (AP-2) (84,85). Es pues relevante determinar si la oligomerización de los receptores mediada por el ligando y determinada por TIRF-M corresponde, como hemos indicado, a un evento de señalización o se trata de complejos de internalización del receptor. Experimentos realizados con CXCR4 como modelo demostraron que en presencia de agentes inhibidores de la internalización como Pit-Stop2 (86), la oligomerización del receptor promovida por CXCL12 se mantiene. Además, el receptor mutante CXCR4K239E/V242A/L246A, que se expresa en la membrana igual que el receptor CXCR4 salvaje y no presenta diferencias en la unión de CXCL12, se internaliza normalmente pero no genera oligómeros detectables por TIRF-M ni promueve migración celular (64). Los datos experimentales indican pues que el proceso de oligomerización y el de agregación previo a la internalización del receptor son procesos independientes.
Varios datos experimentales remarcan el papel esencial de la dinámica del citoesqueleto de actina en la oligomerización de los receptores de quimioquinas promovidas por ligando. El tratamiento de las células con Latrunculina A, un inhibidor de la polimerización de actina por secuestro de monómeros de G-actina (87), bloquea por completo la oligomerización de CXCR4 mediada por CXCL12 y aumenta la difusión libre de los receptores embebidos en la membrana celular (64). Por otro lado, los datos obtenidos utilizando TIRF-M en un mutante natural de CXCR4, CXCR4R334X, que se asocia al síndrome de WHIM, una inmunodeficiencia hereditaria grave (88), indican que, en la membrana celular, este mutante es incapaz de oligomerizar en presencia de CXCL12 y de hecho las células que lo expresan tampoco se orientan a favor de gradiente (89). CXCR4R334X presenta una deleción de los últimos 19 aminoácidos en su región C terminal (90) que le impide, en presencia de CXCL12, ser fosforilado por quinasas asociadas a receptores acoplados a proteínas G (GRKs) y por lo tanto unir b-arrestina y ser internalizado (91) lo que le convierte en un mutante de ganancia de función. Sin embargo, b-arrestina también juega un papel importante como plataforma que asocia otras moléculas señalizadoras. En concreto, su papel es crítico para conectar estos receptores con el citoesqueleto de actina (92). Su presencia condiciona la dinámica de fosforilación /desfosforilación de cofilina, una proteína de unión a actina que cataliza sus procesos de polimerización/despolimerización (93).
La dinámica de la actina es un proceso complejo y en él intervienen muchas proteínas y reguladores, entre ellas las GTPasas Rac, Rho y Cdc42 y multitud de GEFs (guanosin exchange factors) y GAPs (GTPase-activating proteins) que regulan la actividad de las referidas GTPasas (94,95). Aunque la ruta exacta de señalización no está completamente dilucidada, las evidencias indican que también en el caso de los receptores de quimioquinas la dinámica del citoesqueleto de actina controla los procesos de oligomerización y por ende la difusión en la membrana (64,68).
El proceso de oligomerización se inicia una vez que el ligando se une al receptor, es por lo tanto un proceso asociado a la activación del mismo. Así, el bloqueo de la señalización de CXCR4 por tratamiento con toxina de pertussis (PTx), un agente que bloquea la actividad de la proteína Gi, conlleva la eliminación de la oligomerización. El proceso también requiere una correcta dinámica del citoesqueleto de actina, lo que implica directamente la asociación de b-arrestinas al receptor (96). La oligomerización de CXCR4 se bloquea en células deficientes en b-arrestina 1 y, como se ha comentado anteriormente, también en receptores mutantes que no pueden asociar/activar b-arrestinas. Por otro lado, implica a cofilina, una proteína de regula la polimerización y despolimerización de actina y que conecta el proceso con b-arrestinas y por ende con el propio receptor. La participación de la GTPasa Rac en la oligomerización de los receptores de quimioquinas ha sido asimismo propuesta en células dendríticas, donde su presencia se detecta en el frente de avance (97). La falta de oligomerización se ha asociado con la falta de orientación de la célula en los gradientes quimioatrayentes. Si bien es razonable que la célula que no oligomeriza sus receptores en el frente de avance no detecte correctamente el gradiente, el frente de avance celular es una zona rica en actina polimerizada (98) que puede favorecer la oligomerización. La realidad es que la formación de un frente de avance es un proceso dirigido en el que participan muchas proteínas y lípidos que también pueden condicionar la respuesta final de la célula (99).
6. CONCLUSIONES
En los últimos años se ha avanzado notablemente en el conocimiento de la función de las quimioquinas y sus receptores. Gracias a nuevas técnicas de cristalización y al desarrollo de la criomicroscopía conocemos la estructura de algunos de estos receptores, como se regula su expresión, como se produce la unión del ligando, los cambios estructurales que se generan y las rutas de señalización que se activan. Además, el uso de nuevas tecnologías, entre ellas las de imagen óptica avanzada y el desarrollo de algoritmos para su cuantificación, nos ha permitido determinar que los receptores pueden adoptar distintas conformaciones dinámicas en la membrana celular, que también condicionan las respuestas funcionales de estos mediadores inflamatorios. Entender esa dinámica en la membrana y como se modifica por la composición lipídica de la misma, la expresión de otras proteínas que condicionan la difusión de los receptores y/o la dinámica del citoesqueleto de actina es crítico para tener una visión completa del sistema si queremos intervenir terapéuticamente en su función.
Agradecimientos
Este trabajo ha sido financiado por el Ministerio de Ciencia, Innovación y Universidades (PID2020-114980RB-I00 apoyado por MCIN/AEI/10.13039/501100011033/FEDER, UE) y por el Programa de Biomedicina de la Comunidad de Madrid, España (S2022/BMD-7274 RETAR-A-COVID-CM). Muchas gracias a los miembros actuales y anteriores del grupo de señalización por quimioquinas del Centro Nacional de Biotecnología/CSIC por su excelente trabajo que ha inspirado esta revisión.
7. REFERENCIAS
- Franz CM, Jones GE, Ridley AJ. Cell Migration in Development and Disease. Dev Cell. 2002 Feb 1;2(2):153–8.
- Yang H, Ganguly A, Cabral F. Inhibition of Cell Migration and Cell Division Correlates with Distinct Effects of Microtubule Inhibiting Drugs. J Biol Chem [Internet]. 2010 Oct 10 [cited 2024 Jul 1];285(42):32242. Available from: /pmc/articles/PMC2952225/
- Maeda YT, Inose J, Matsuo MY, Iwaya S, Sano M. Ordered Patterns of Cell Shape and Orientational Correlation during Spontaneous Cell Migration. PLoS One [Internet]. 2008 Nov 17 [cited 2024 Jul 1];3(11):e3734. Available from: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0003734
- Dubey S, Jaiswal B, Gupta A. TIP60 acts as a regulator of genes involved in filopodia formation and cell migration during wound healing. Journal of Biological Chemistry [Internet]. 2022 Jul 1 [cited 2024 Jul 1];298(7):102015. Available from: http://www.jbc.org/article/S0021925822004550/fulltext
- Delgado M, Lennon-Duménil AM. How cell migration helps immune sentinels. Front Cell Dev Biol. 2022 Oct 4;10.
- Takihara Y, Higaki T, Yokomizo T, Umemoto T, Ariyoshi K, Hashimoto M, et al. Bone marrow imaging reveals the migration dynamics of neonatal hematopoietic stem cells. Communications Biology 2022 5:1 [Internet]. 2022 Aug 2 [cited 2024 Jul 1];5(1):1–10. Available from: https://www.nature.com/articles/s42003-022-03733-x
- Hampton HR, Chtanova T. Lymphatic migration of immune cells. Front Immunol [Internet]. 2019 May 28 [cited 2024 Jul 1];10(MAY):451736. Available from: www.frontiersin.org
- Worbs T, Förster R. T cell migration dynamics within lymph nodes during steady state: an overview of extracellular and intracellular factors influencing the basal intranodal T cell motility. Curr Top Microbiol Immunol [Internet]. 2009 [cited 2024 Jul 1];334(1):71–105. Available from: https://pubmed.ncbi.nlm.nih.gov/19521682/
- Vestweber D. Relevance of endothelial junctions in leukocyte extravasation and vascular permeability. Ann N Y Acad Sci [Internet]. 2012 [cited 2024 Jul 1];1257(1):184–92. Available from: https://pubmed.ncbi.nlm.nih.gov/22671605/
- Nourshargh S, Alon R. Leukocyte Migration into Inflamed Tissues. Immunity. 2014 Nov 20;41(5):694–707.
- Yamada KM, Sixt M. Mechanisms of 3D cell migration. Nature Reviews Molecular Cell Biology 2019 20:12 [Internet]. 2019 Oct 3 [cited 2024 Jul 1];20(12):738–52. Available from: https://www.nature.com/articles/s41580-019-0172-9
- Bodor DL, Pönisch W, Endres RG, Paluch EK. Of Cell Shapes and Motion: The Physical Basis of Animal Cell Migration. Dev Cell [Internet]. 2020 Mar 9 [cited 2024 Jul 1];52(5):550–62. Available from: https://pubmed.ncbi.nlm.nih.gov/32155438/
- Bergert M, Chandradoss SD, Desai RA, Paluch E. Cell mechanics control rapid transitions between blebs and lamellipodia during migration. Proc Natl Acad Sci U S A [Internet]. 2012 Sep 4 [cited 2024 Jul 1];109(36):14434–9. Available from: https://www.pnas.org/doi/abs/10.1073/pnas.1207968109
- Sánchez-Madrid F, Del Pozo MA. Leukocyte polarization in cell migration and immune interactions. EMBO J [Internet]. 1999 Feb 1 [cited 2024 Jul 2];18(3):501–11. Available from: https://www.embopress.org/doi/10.1093/emboj/18.3.501
- Serrador JM, Alonso-Lebrero JL, Del Pozo MA, Furthmayr H, Schwartz-Albiez R, Calvo J, et al. Moesin Interacts with the Cytoplasmic Region of Intercellular Adhesion Molecule-3 and Is Redistributed to the Uropod of T Lymphocytes during Cell Polarization. Journal of Cell Biology [Internet]. 1997 Sep 22 [cited 2024 Jul 2];138(6):1409–23. Available from: http://www.jcb.org
- Steffen A, Ladwein M, Dimchev GA, Hein A, Schwenkmezger L, Arens S, et al. Rac function is crucial for cell migration but is not required for spreading and focal adhesion formation. J Cell Sci [Internet]. 2013 Oct 15 [cited 2024 Jul 2];126(20):4572–88. Available from: https://dx.doi.org/10.1242/jcs.118232
- Hughes CE, Nibbs RJB. A guide to chemokines and their receptors. FEBS J [Internet]. 2018 Aug 1 [cited 2024 Jul 2];285(16):2944–71. Available from: https://onlinelibrary.wiley.com/doi/full/10.1111/febs.14466
- Nomiyama H, Osada N, Yoshie O. Systematic classification of vertebrate chemokines based on conserved synteny and evolutionary history. Genes Cells [Internet]. 2013 Jan [cited 2024 Jul 2];18(1):1–16. Available from: https://pubmed.ncbi.nlm.nih.gov/23145839/
- Griffith JW, Sokol CL, Luster AD. Chemokines and chemokine receptors: positioning cells for host defense and immunity. Annu Rev Immunol [Internet]. 2014 [cited 2024 Jul 2];32:659–702. Available from: https://pubmed.ncbi.nlm.nih.gov/24655300/
- Schulz O, Hammerschmidt SI, Moschovakis GL, Förster R. Chemokines and Chemokine Receptors in Lymphoid Tissue Dynamics. Annu Rev Immunol [Internet]. 2016 May 20 [cited 2024 Jul 2];34:203–42. Available from: https://pubmed.ncbi.nlm.nih.gov/26907216/
- Shi G, Partida-Sánchez S, Misra RS, Tighe M, Borchers MT, Lee JJ, et al. Identification of an alternative G{alpha}q-dependent chemokine receptor signal transduction pathway in dendritic cells and granulocytes. J Exp Med [Internet]. 2007 Oct 29 [cited 2024 Jul 3];204(11):2705–18. Available from: https://pubmed.ncbi.nlm.nih.gov/17938235/
- Horuk R. Chemokine receptors. Cytokine Growth Factor Rev [Internet]. 2001 [cited 2024 Jul 2];12(4):313–35. Available from: https://pubmed.ncbi.nlm.nih.gov/11544102/
- Coughlan CM, McManus CM, Sharron M, Gao ZY, Murphy D, Jaffer S, et al. Expression of multiple functional chemokine receptors and monocyte chemoattractant protein-1 in human neurons. Neuroscience. 2000 May 1;97(3):591–600.
- Haraldsen G, Rot A. Coy decoy with a new ploy: Interceptor controls the levels of homeostatic chemokines. Eur J Immunol [Internet]. 2006 Jul 1 [cited 2024 Jul 2];36(7):1659–61. Available from: https://onlinelibrary.wiley.com/doi/full/10.1002/eji.200636327
- Levoye A, Balabanian K, Baleux F, Bachelerie F, Lagane B. CXCR7 heterodimerizes with CXCR4 and regulates CXCL12-mediated G protein signaling. Blood [Internet]. 2009 Jun 11 [cited 2024 Jul 2];113(24):6085–93. Available from: https://dx.doi.org/10.1182/blood-2008-12-196618
- Sierro F, Biben C, Martínez-Muñoz L, Mellado M, Ransohoff RM, Li M, et al. Disrupted cardiac development but normal hematopoiesis in mice deficient in the second CXCL12/SDF-1 receptor, CXCR7. Proc Natl Acad Sci U S A [Internet]. 2007 Sep 11 [cited 2024 Jul 2];104(37):14759–64. Available from: https://www.pnas.org/doi/abs/10.1073/pnas.0702229104
- Alcami A. Viral mimicry of cytokines, chemokines and their receptors. Nat Rev Immunol [Internet]. 2003 Jan [cited 2024 Jul 2];3(1):36–50. Available from: https://pubmed.ncbi.nlm.nih.gov/12511874/
- Crump MP, Gong JH, Loetscher P, Rajarathnam K, Amara A, Arenzana-Seisdedos F, et al. Solution structure and basis for functional activity of stromal cell-derived factor-1; dissociation of CXCR4 activation from binding and inhibition of HIV-1. EMBO J [Internet]. 1997 Dec 1 [cited 2024 Jul 2];16(23):6996–7007. Available from: https://pubmed.ncbi.nlm.nih.gov/9384579/
- Gupta SK, Pillarisetti K, Thomas RA, Aiyar N. Pharmacological evidence for complex and multiple site interaction of CXCR4 with SDF-1alpha: implications for development of selective CXCR4 antagonists. Immunol Lett [Internet]. 2001 Aug 1 [cited 2024 Jul 2];78(1):29–34. Available from: https://pubmed.ncbi.nlm.nih.gov/11470148/
- Wescott MP, Kufareva I, Paes C, Goodman JR, Thaker Y, Puffer BA, et al. Signal transmission through the CXC chemokine receptor 4 (CXCR4) transmembrane helices. Proc Natl Acad Sci U S A [Internet]. 2016 Aug 30 [cited 2024 Jul 2];113(35):9928–33. Available from: https://www.pnas.org/doi/abs/10.1073/pnas.1601278113
- Veldkamp CT, Seibert C, Peterson FC, De La Cruz NB, Haugner JC, Basnet H, et al. Structural Basis of CXCR4 Sulfotyrosine Recognition by the Chemokine SDF-1/CXCL12. Sci Signal [Internet]. 2008 Sep 16 [cited 2024 Jul 2];1(37):ra4. Available from: /pmc/articles/PMC2692298/
- Thompson S, Martínez-Burgo B, Sepuru KM, Rajarathnam K, Kirby JA, Sheerin NS, et al. Regulation of Chemokine Function: The Roles of GAG-Binding and Post-Translational Nitration. Int J Mol Sci [Internet]. 2017 Aug 3 [cited 2024 Jul 2];18(8). Available from: https://pubmed.ncbi.nlm.nih.gov/28771176/
- Proudfoot AEI, Johnson Z, Bonvin P, Handel TM. Glycosaminoglycan Interactions with Chemokines Add Complexity to a Complex System. Pharmaceuticals 2017, Vol 10, Page 70 [Internet]. 2017 Aug 9 [cited 2024 Jul 2];10(3):70. Available from: https://www.mdpi.com/1424-8247/10/3/70/htm
- Trettel F, Di Bartolomeo S, Lauro C, Catalano M, Ciotti MT, Limatola C. Ligand-independent CXCR2 dimerization. J Biol Chem [Internet]. 2003 Oct 17 [cited 2024 Jul 2];278(42):40980–8. Available from: https://pubmed.ncbi.nlm.nih.gov/12888558/
- Vila-Coro AJ, Rodri´guez M, Rodri´guez-Frade R, Marti´n A, Marti´n M, Ana DE, et al. The chemokine SDF-lα triggers CXCR4 receptor dimerization and activates the JAK/STAT pathway. The FASEB Journal [Internet]. 1999 Oct 1 [cited 2024 Jul 2];13(13):1699–710. Available from: https://onlinelibrary.wiley.com/doi/full/10.1096/fasebj.13.13.1699
- Harrison C, van der Graaf PH. Current methods used to investigate G protein coupled receptor oligomerisation. J Pharmacol Toxicol Methods. 2006 Jul 1;54(1):26–35.
- Pfleger KDG, Eidne KA. Illuminating insights into protein-protein interactions using bioluminescence resonance energy transfer (BRET). Nat Methods [Internet]. 2006 [cited 2024 Jul 2];3(3):165–74. Available from: https://pubmed.ncbi.nlm.nih.gov/16489332/
- Pfleger KDG, Seeber RM, Eidne KA. Bioluminescence resonance energy transfer (BRET) for the real-time detection of protein-protein interactions. Nat Protoc [Internet]. 2006 Jun [cited 2024 Jul 2];1(1):337–45. Available from: https://pubmed.ncbi.nlm.nih.gov/17406254/
- Muñoz LM, Holgado BL, Martínez-A C, Rodríguez-Frade JM, Mellado M. Chemokine receptor oligomerization: a further step toward chemokine function. Immunol Lett [Internet]. 2012 Jul 30 [cited 2024 Jul 2];145(1–2):23–9. Available from: https://pubmed.ncbi.nlm.nih.gov/22698180/
- Wilson S, Wilkinson G, Milligan G. The CXCR1 and CXCR2 Receptors Form Constitutive Homo- and Heterodimers Selectively and with Equal Apparent Affinities. Journal of Biological Chemistry. 2005 Aug 5;280(31):28663–74.
- Martínez Muñoz L, Lucas P, Navarro G, Checa AI, Franco R, Martínez-A. C, et al. Dynamic Regulation of CXCR1 and CXCR2 Homo- and Heterodimers. The Journal of Immunology [Internet]. 2009 Dec 1 [cited 2024 Jul 2];183(11):7337–46. Available from: https://dx.doi.org/10.4049/jimmunol.0901802
- S.-H. Park P. Ensemble of G protein-coupled receptor active states. Curr Med Chem [Internet]. 2012 Feb 10 [cited 2024 Jul 2];19(8):1146–54. Available from: https://pubmed.ncbi.nlm.nih.gov/22300048/
- Weis WI, Kobilka BK. The Molecular Basis of G Protein-Coupled Receptor Activation. Annu Rev Biochem [Internet]. 2018 Jun 20 [cited 2024 Jul 2];87:897–919. Available from: https://pubmed.ncbi.nlm.nih.gov/29925258/
- Babcock GJ, Farzan M, Sodroski J. Ligand-independent dimerization of CXCR4, a principal HIV-1 coreceptor. J Biol Chem [Internet]. 2003 Jan 31 [cited 2024 Jul 2];278(5):3378–85. Available from: https://pubmed.ncbi.nlm.nih.gov/12433920/
- Percherancier Y, Berchiche YA, Slight I, Volkmer-Engert R, Tamamura H, Fujii N, et al. Bioluminescence resonance energy transfer reveals ligand-induced conformational changes in CXCR4 homo- and heterodimers. J Biol Chem [Internet]. 2005 Mar 18 [cited 2024 Jul 2];280(11):9895–903. Available from: https://pubmed.ncbi.nlm.nih.gov/15632118/
- Wang J, Norcross M. Dimerization of chemokine receptors in living cells: key to receptor function and novel targets for therapy. Drug Discov Today [Internet]. 2008 Jul [cited 2024 Jul 2];13(13–14):625–32. Available from: https://pubmed.ncbi.nlm.nih.gov/18598920/
- Barroso R, Martínez Muñoz L, Barrondo S, Vega B, Holgado BL, Lucas P, et al. EBI2 regulates CXCL13-mediated responses by heterodimerization with CXCR5. The FASEB Journal [Internet]. 2012 Dec 1 [cited 2024 Jul 2];26(12):4841–54. Available from: https://onlinelibrary.wiley.com/doi/full/10.1096/fj.12-208876
- Choi WT, Tian S, Dong CZ, Kumar S, Liu D, Madani N, et al. Unique ligand binding sites on CXCR4 probed by a chemical biology approach: implications for the design of selective human immunodeficiency virus type 1 inhibitors. J Virol [Internet]. 2005 Dec 15 [cited 2024 Jul 2];79(24):15398–404. Available from: https://pubmed.ncbi.nlm.nih.gov/16306611/
- Del Prete A, Sozio F, Schioppa T, Ponzetta A, Vermi W, Calza S, et al. The atypical receptor CCRL2 is essential for NK cell-dependent resistance against lung cancer. Cancer Immunol Res [Internet]. 2019 Nov 11 [cited 2024 Jul 2];7(11):1775. Available from: /pmc/articles/PMC7176487/
- Ge B, Lao J, Li J, Chen Y, Song Y, Huang F. Single-molecule imaging reveals dimerization/oligomerization of CXCR4 on plasma membrane closely related to its function. Scientific Reports 2017 7:1 [Internet]. 2017 Dec 4 [cited 2024 Jul 2];7(1):1–9. Available from: https://www.nature.com/articles/s41598-017-16802-7
- Sohy D, Yano H, de Nadai P, Urizar E, Guillabert A, Javitch JA, et al. Hetero-oligomerization of CCR2, CCR5, and CXCR4 and the Protean Effects of “Selective” Antagonists. J Biol Chem [Internet]. 2009 Nov 11 [cited 2024 Jul 2];284(45):31270. Available from: /pmc/articles/PMC2781525/
- Hauser MA, Schaeuble K, Kindinger I, Hauck CR, Boyman O, Legler Correspondence DF. Inflammation-Induced CCR7 Oligomers Form Scaffolds to Integrate Distinct Signaling Pathways for Efficient Cell Migration. Immunity [Internet]. 2016 [cited 2024 Jul 2];44:59–72. Available from: http://dx.doi.org/10.1016/j.immuni.2015.12.010
- Kramp BK, Sarabi A, Koenen RR, Weber C. Heterophilic chemokine receptor interactions in chemokine signaling and biology. Exp Cell Res [Internet]. 2011 [cited 2024 Jul 2];317(5):655–63. Available from: https://pubmed.ncbi.nlm.nih.gov/21146524/
- Cascio G, Martín-Cófreces NB, Rodríguez-Frade JM, López-Cotarelo P, Criado G, Pablos JL, et al. CXCL12 Regulates through JAK1 and JAK2 Formation of Productive Immunological Synapses. The Journal of Immunology [Internet]. 2015 Jun 1 [cited 2024 Jul 2];194(11):5509–19. Available from: https://dx.doi.org/10.4049/jimmunol.1402419
- Kallikourdis M, Trovato AE, Anselmi F, Sarukhan A, Roselli G, Tassone L, et al. The CXCR4 mutations in WHIM syndrome impair the stability of the T-cell immunologic synapse. Blood [Internet]. 2013 Aug 1 [cited 2024 Jul 2];122(5):666–73. Available from: https://pubmed.ncbi.nlm.nih.gov/23794067/
- Nieto M, Frade JMR, Sancho D, Mellado M, Martinez-A C, Sánchez-Madrid F. Polarization of Chemokine Receptors to the Leading Edge during Lymphocyte Chemotaxis. J Exp Med [Internet]. 1997 Jul 7 [cited 2024 Jul 2];186(1):153. Available from: /pmc/articles/PMC2198956/
- Wu CY, Tsai YY, Chen SY, Lin YP, Shin JW, Wu CC, et al. Interaction of Zap70 and CXCR4 receptor at lamellipodia that determines the directionality during Jurkat T cells chemotaxis. Mol Immunol. 2017 Oct 1;90:245–54.
- Fernández-Arenas E, Calleja E, Martínez-Martín N, Gharbi SI, Navajas R, García-Medel N, et al. β-arrestin-1 mediates the TCR-triggered re-routing of distal receptors to the immunological synapse by a PKC-mediated mechanism. EMBO Journal [Internet]. 2014 Mar 18 [cited 2024 Jul 3];33(6):559–77. Available from: https://www.embopress.org/doi/10.1002/embj.201386022
- Baba M, Nishimura O, Kanzaki N, Okamoto M, Sawada H, Iizawa Y, et al. A small-molecule, nonpeptide CCR5 antagonist with highly potent and selective anti-HIV-1 activity. Proc Natl Acad Sci U S A [Internet]. 1999 May 11 [cited 2024 Jul 12];96(10):5698–703. Available from: https://www.pnas.org/doi/abs/10.1073/pnas.96.10.5698
- Sohy D, Parmentier M, Springael JY. Allosteric transinhibition by specific antagonists in CCR2/CXCR4 heterodimers. J Biol Chem [Internet]. 2007 Oct 12 [cited 2024 Jul 3];282(41):30062–9. Available from: https://pubmed.ncbi.nlm.nih.gov/17715128/
- Sako Y, Uyemura T. Total Internal Reflection Fluorescence Microscopy for Single-molecule Imaging in Living Cells. Cell Struct Funct. 2002 Oct;27(5):357–65.
- Schneckenburger H. Total internal reflection fluorescence microscopy: Technical innovations and novel applications. Curr Opin Biotechnol. 2005;16(1 SPEC. ISS.):13–8.
- Martínez-Muñoz L, Barroso R, Dyrhaug SY, Navarro G, Lucas P, Soriano SF, et al. CCR5/CD4/CXCR4 oligomerization prevents HIV-1 gp120IIIB binding to the cell surface. Proc Natl Acad Sci U S A [Internet]. 2014 May 13 [cited 2024 Jul 3];111(19):E1960–9. Available from: https://www.pnas.org/doi/abs/10.1073/pnas.1322887111
- Martínez-Muñ Oz L, Miguel Rodríguez-Frade J, Sá Nchez-Madrid F, García-Parajo MF, Mellado Correspondence M. Separating Actin-Dependent Chemokine Receptor Nanoclustering from Dimerization Indicates a Role for Clustering in CXCR4 Signaling and Function. Mol Cell [Internet]. 2018 [cited 2024 Jul 3];70:106–19. Available from: https://doi.org/10.1016/j.molcel.2018.02.034
- Di Marino D, Conflitti P, Motta S, Limongelli V. Structural basis of dimerization of chemokine receptors CCR5 and CXCR4. Nature Communications 2023 14:1 [Internet]. 2023 Oct 13 [cited 2024 Jul 3];14(1):1–16. Available from: https://www.nature.com/articles/s41467-023-42082-z
- Wu B, Chien EYT, Mol CD, Fenalti G, Liu W, Katritch V, et al. Structures of the CXCR4 chemokine GPCR with small-molecule and cyclic peptide antagonists. Science [Internet]. 2010 Nov 19 [cited 2024 Jul 3];330(6007):1066–71. Available from: https://pubmed.ncbi.nlm.nih.gov/20929726/
- Singer SJ, Nicolson GL. The fluid mosaic model of the structure of cell membranes. Science [Internet]. 1972 [cited 2024 Jul 3];175(4023):720–31. Available from: https://pubmed.ncbi.nlm.nih.gov/4333397/
- Mattila PK, Batista FD, Treanor B. Dynamics of the actin cytoskeleton mediates receptor cross talk: An emerging concept in tuning receptor signaling. Journal of Cell Biology [Internet]. 2016 Feb 1 [cited 2024 Jul 3];212(3):267–80. Available from: www.jcb.org/cgi/doi/10.1083/jcb.201504137
- Bezanilla M, Gladfelter AS, Kovar DR, Lee WL. Cytoskeletal dynamics: A view from the membrane. J Cell Biol [Internet]. 2015 May 5 [cited 2024 Jul 3];209(3):329. Available from: /pmc/articles/PMC4427793/
- Bethani I, Skånland SS, Dikic I, Acker-Palmer A. Spatial organization of transmembrane receptor signalling. EMBO Journal [Internet]. 2010 Aug 18 [cited 2024 Jul 3];29(16):2677–88. Available from: https://www.embopress.org/doi/10.1038/emboj.2010.175
- Kusumi A, Koyama-Honda I, Suzuki K. Molecular Dynamics and Interactions for Creation of Stimulation-Induced Stabilized Rafts from Small Unstable Steady-State Rafts. Traffic [Internet]. 2004 Apr 1 [cited 2024 Jul 3];5(4):213–30. Available from: https://onlinelibrary.wiley.com/doi/full/10.1111/j.1600-0854.2004.0178.x
- Kusumi A, Suzuki K. Toward understanding the dynamics of membrane-raft-based molecular interactions. Biochim Biophys Acta Mol Cell Res. 2005 Dec 30;1746(3):234–51.
- Feron O, Smith TW, Michel T, Kelly RA. Dynamic targeting of the agonist-stimulated m2 muscarinic acetylcholine receptor to caveolae in cardiac myocytes. Journal of Biological Chemistry [Internet]. 1997 Jul 11 [cited 2024 Jul 3];272(28):17744–8. Available from: http://www.jbc.org/article/S0021925818392548/fulltext
- De Weerd WFC, Leeb-Lundberg LMF. Bradykinin sequesters B2 bradykinin receptors and the receptor-coupled Galpha subunits Galphaq and Galphai in caveolae in DDT1 MF-2 smooth muscle cells. J Biol Chem [Internet]. 1997 [cited 2024 Jul 3];272(28):17858–66. Available from: https://pubmed.ncbi.nlm.nih.gov/9211942/
- Dupree P, Parton RG, Raposol G, Kurzchalia2 T V, Simons3 K. Caveolae and sorting in the trans-Golgi network of epithelial cells. EMBO J [Internet]. 1993 Apr [cited 2024 Jul 3];12(4):1597. Available from: /pmc/articles/PMC413373/?report=abstract
- Ishizaka N, Griendling KK, Lassè B, Alexander RW. Angiotensin II Type 1 Receptor Relationship With Caveolae and Caveolin After Initial Agonist Stimulation. 1998 [cited 2024 Jul 3]; Available from: http://ahajournals.org
- Mañes S, Lacalle RA, Gómez-Moutón C, Del Real G, Mira E, Martínez-A C. Membrane raft microdomains in chemokine receptor function. Semin Immunol [Internet]. 2001 [cited 2024 Jul 3];13(2):147–57. Available from: https://pubmed.ncbi.nlm.nih.gov/11308298/
- Levental I, Levental KR, Heberle FA. Lipid Rafts: Controversies Resolved, Mysteries Remain. Trends Cell Biol [Internet]. 2020 May 1 [cited 2024 Jul 3];30(5):341–53. Available from: http://www.cell.com/article/S0962892420300313/fulltext
- Gardeta SR, García-Cuesta EM, D’Agostino G, Soler Palacios B, Quijada-Freire A, Lucas P, et al. Sphingomyelin Depletion Inhibits CXCR4 Dynamics and CXCL12-Mediated Directed Cell Migration in Human T Cells. Front Immunol [Internet]. 2022 Jul 12 [cited 2024 Jul 3];13:925559. Available from: https://www.
- Krone W, Klass A, Nagele H, Behnke B, Greten H. Effects of prostaglandins on LDL receptor activity and cholesterol synthesis in freshly isolated human mononuclear leukocytes. J Lipid Res. 1988 Dec 1;29(12):1663–9.
- Bray D. Protein molecules as computational elements in living cells. Nature 1995 376:6538 [Internet]. 1995 Jul 27 [cited 2024 Jul 3];376(6538):307–12. Available from: https://www.nature.com/articles/376307a0
- Cho W, Stahelin R V. Membrane-protein interactions in cell signaling and membrane trafficking. Annu Rev Biophys Biomol Struct [Internet]. 2005 [cited 2024 Jul 3];34:119–51. Available from: https://pubmed.ncbi.nlm.nih.gov/15869386/
- Gurry T, Kahramanoǧullari O, Endres RG. Biophysical Mechanism for Ras-Nanocluster Formation and Signaling in Plasma Membrane. PLoS One [Internet]. 2009 Jul 9 [cited 2024 Jul 3];4(7):e6148. Available from: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0006148
- Neel NF, Schutyser E, Sai J, Fan GH, Richmond A. Chemokine receptor internalization and intracellular trafficking. Cytokine Growth Factor Rev. 2005 Dec 1;16(6):637–58.
- Marchese A. Endocytic trafficking of chemokine receptors. Curr Opin Cell Biol [Internet]. 2014 Apr [cited 2024 Jul 3];27(1):72. Available from: /pmc/articles/PMC4268779/
- Morinelli TA, Walker LP, Velez JCQ, Ullian ME. Clathrin-dependent internalization of the angiotensin II AT₁A receptor links receptor internalization to COX-2 protein expression in rat aortic vascular smooth muscle cells. Eur J Pharmacol [Internet]. 2015 Feb 5 [cited 2024 Jul 5];748:143–8. Available from: https://pubmed.ncbi.nlm.nih.gov/25542758/
- Fujiwara I, Zweifel ME, Courtemanche N, Pollard TD. Latrunculin A Accelerates Actin Filament Depolymerization in Addition to Sequestering Actin Monomers. Curr Biol [Internet]. 2018 Oct 8 [cited 2024 Jul 5];28(19):3183-3192.e2. Available from: https://pubmed.ncbi.nlm.nih.gov/30270183/
- Zmajkovicova K, Pawar S, Maier-Munsa S, Maierhofer B, Wiest I, Skerlj R, et al. Genotype–phenotype correlations in WHIM syndrome: a systematic characterization of CXCR4WHIM variants. Genes & Immunity 2022 23:6 [Internet]. 2022 Sep 12 [cited 2024 Jul 5];23(6):196–204. Available from: https://www.nature.com/articles/s41435-022-00181-9
- García-Cuesta EM, Rodríguez-Frade JM, Gardeta SR, D’Agostino G, Martínez P, Palacios BS, et al. Altered CXCR4 dynamics at the cell membrane impairs directed cell migration in WHIM syndrome patients. Proc Natl Acad Sci U S A [Internet]. 2022 May 24 [cited 2024 Jul 3];119(21):e2119483119. Available from: https://www.pnas.org/doi/abs/10.1073/pnas.2119483119
- Hernandez PA, Gorlin RJ, Lukens JN, Taniuchi S, Bohinjec J, Francois F, et al. Mutations in the chemokine receptor gene CXCR4 are associated with WHIM syndrome, a combined immunodeficiency disease. Nat Genet [Internet]. 2003 May 1 [cited 2024 Jul 3];34(1):70–4. Available from: https://pubmed.ncbi.nlm.nih.gov/12692554/
- McCormick PJ, Segarra M, Gasperini P, Gulino AV, Tosato G. Impaired Recruitment of Grk6 and β-Arrestin2 Causes Delayed Internalization and Desensitization of a WHIM Syndrome-Associated CXCR4 Mutant Receptor. PLoS One [Internet]. 2009 [cited 2024 Jul 3];4(12):e8102. Available from: https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0008102
- Min J, DeFea K. β-arrestin-dependent actin reorganization: Bringing the right players together at the leading edge. Mol Pharmacol. 2011 Nov;80(5):760–8.
- Borroni EM, Cancellieri C, Vacchini A, Benureau Y, Lagane B, Bachelerie F, et al. β-arrestin-dependent activation of the cofilin pathway is required for the scavenging activity of the atypical chemokine receptor D6. Sci Signal [Internet]. 2013 Apr 30 [cited 2024 Jul 3];6(273). Available from: https://www.science.org/doi/10.1126/scisignal.2003627
- Spiering D, Hodgson L. Dynamics of the Rho-family small GTPases in actin regulation and motility. Cell Adh Migr [Internet]. 2011 [cited 2024 Jul 3];5(2):170–80. Available from: https://pubmed.ncbi.nlm.nih.gov/21178402/
- Bos JL, Rehmann H, Wittinghofer A. GEFs and GAPs: critical elements in the control of small G proteins. Cell [Internet]. 2007 Jun 1 [cited 2024 Jul 5];129(5):865–77. Available from: https://pubmed.ncbi.nlm.nih.gov/17540168/
- D’Agostino G, Artinger M, Locati M, Perez L, Legler DF, Bianchi ME, et al. β-Arrestin1 and β-Arrestin2 Are Required to Support the Activity of the CXCL12/HMGB1 Heterocomplex on CXCR4. Front Immunol [Internet]. 2020 Sep 18 [cited 2024 Jul 3];11:550824. Available from: www.frontiersin.org
- Laufer JM, Hauser MA, Kindinger I, Purvanov V, Pauli A, Legler DF. Chemokine Receptor CCR7 Triggers an Endomembrane Signaling Complex for Spatial Rac Activation. Cell Rep [Internet]. 2019 Oct 22 [cited 2024 Jul 29];29(4):995-1009.e6. Available from: http://www.cell.com/article/S2211124719312197/fulltext
- Wittmann T, Bokoch GM, Waterman-Storer CM. Regulation of leading edge microtubule and actin dynamics downstream of Rac1. J Cell Biol [Internet]. 2003 Jun 6 [cited 2024 Jul 5];161(5):845. Available from: /pmc/articles/PMC2172968/
- Ridley AJ. Life at the leading edge. Cell [Internet]. 2011 Jun 24 [cited 2024 Jul 3];145(7):1012–22. Available from: https://pubmed.ncbi.nlm.nih.gov/21703446/
- Mastrogiovanni M, Juzans M, Alcover A, Di Bartolo V. Coordinating Cytoskeleton and Molecular Traffic in T Cell Migration, Activation, and Effector Functions. Front Cell Dev Biol [Internet]. 2020 Oct 21 [cited 2024 Jul 18];8:591348. Available from: www.frontiersin.org
- Kenific CM, Wittmann T, Debnath J. Autophagy in adhesion and migration. J Cell Sci [Internet]. 2016 Oct 15 [cited 2024 Jul 18];129(20):3685–93. Available from: https://dx.doi.org/10.1242/jcs.188490
- Bogacka J, Pawlik K, Ciapała K, Ciechanowska A, Mika J. CC Chemokine Receptor 4 (CCR4) as a Possible New Target for Therapy. Int J Mol Sci. 2022 Dec 1;23(24).